首页R语言根据基因的表达分成高低两组,并绘制的K-M曲线生存曲线
R语言根据基因的表达分成高低两组,并绘制的K-M曲线生存曲线
时间: 2023-06-12 13:03:44 浏览: 410
首先,需要准备好基因表达数据和生存数据。假设我们有一个名为`gene_expression`的数据框,其中包含基因表达数据,还有一个名为`survival_data`的数据框,其中包含生存数据,包括生存时间和是否死亡两列。
接下来,可以使用`survival`包中的`surv_cutpoint()`函数将基因表达数据分成高低两组。该函数会返回分组后的数据框和分组阈值。
```R
library(survival)
# 将基因表达数据分成高低两组
cutpoint <- surv_cutpoint(gene_expression, survival_data$time, survival_data$status)
```
接下来,可以使用`survival`包中的`survfit()`函数计算生存曲线,并绘制K-M曲线。
```R
# 计算生存曲线
fit <- survfit(Surv(survival_data$time, survival_data$status) ~ cutpoint$cutpoint, data = gene_expression)
# 绘制K-M曲线
plot(fit, xlab = "Time", ylab = "Survival Probability")
legend("bottomleft", legend = c("Low Expression", "High Expression"), lty = 1:2, col = 1:2)
```
这样就可以得到基于基因表达数据的K-M曲线生存曲线。需要注意的是,这种方法只是一种基于基因表达数据进行分组的方法,并不能保证这两组之间的生物学差异具有统计显著性。因此,需要进一步进行生物学分析,以确定这些差异是否具有生物学意义。
阅读全文
CSDN会员
开通CSDN年卡参与万元壕礼抽奖
大家在看
惠普HP45喷墨打印头规格书
惠普HP45喷墨打印头和台湾IUT300是兼容 打印高度12.7mm IUT308打印头25.4mm 的也可以参考这个 打印头头点数一样只是放到一排 上面 这个打印机真实DIP300的 半点虚拟的600DPI 用于喷码机上面比较多 墨水可以用快干的
清华virtuoso简明教程
清华virtuoso简明教程,很详细的介绍了virtuoso,读者读后会很清楚的了解并运用
定向耦合器与三分贝电桥.pdf
定向耦合器是微波与雷达馈线技术中广泛应用的元件之一,它是一种四端口器件
西门子博途V18系统手册
西门子博途V18系统手册
智能变电站SCD文件的集成工具 南瑞继保设计工具
智能变电站SCD文件的集成工具 南瑞继保设计工具 61850 支持win11操作系统
最新推荐
Unity绘制二维动态曲线
Unity绘制二维动态曲线 Unity绘制二维动态曲线是指在Unity中使用Texture2D和RawImage控件来绘制二维动态曲线的技术。该技术可以实现真正的二维曲线绘制,相比使用Line Render可以节省计算和渲染。 在本文中,我们...
C语言绘制余弦、正弦曲线
C语言绘制余弦、正弦曲线 本文主要介绍了使用C语言绘制余弦、正弦曲线的相关代码,具有参考价值。下面是对标题、描述、标签和部分内容的解释和知识点总结: 首先,C语言绘制余弦曲线的代码使用了反余弦函数acos...
C#画笔Pen绘制光滑模式曲线的方法
本篇文章将深入探讨如何使用C#的画笔类(Pen)来绘制光滑模式的曲线,并介绍相关属性的设置技巧。 首先,`Pen` 类是C#中用于绘制线条、轮廓和其他形状的基本工具。它可以控制线条的颜色、宽度、线型等特性。在示例...
Qt 鼠标/触屏绘制平滑曲线(支持矢量/非矢量方式)
"Qt 鼠标/触屏绘制平滑曲线(支持矢量/非矢量方式)" Qt 鼠标/触屏绘制平滑曲线是 Qt 编程中的一种常见技术,用于在用户界面中绘制平滑曲线。这种技术可以应用于触摸屏设备和鼠标设备上,支持矢量和非矢量两种方式...
Python图形绘制操作之正弦曲线实现方法分析
这篇文档主要关注如何使用Python来绘制正弦曲线,这是数学和科学领域常见的图形。我们将详细探讨如何利用numpy和matplotlib.pyplot两个库来实现这一目标。 首先,numpy是Python的一个科学计算库,它提供了大量的...
掌握HTML/CSS/JS和Node.js的Web应用开发实践
资源摘要信息:"本资源摘要信息旨在详细介绍和解释提供的文件中提及的关键知识点,特别是与Web应用程序开发相关的技术和概念。"
知识点一:两层Web应用程序架构
两层Web应用程序架构通常指的是客户端-服务器架构中的一个简化版本,其中用户界面(UI)和应用程序逻辑位于客户端,而数据存储和业务逻辑位于服务器端。在这种架构中,客户端(通常是一个Web浏览器)通过HTTP请求与服务器端进行通信。服务器端处理请求并返回数据或响应,而客户端负责展示这些信息给用户。
知识点二:HTML/CSS/JavaScript技术栈
在Web开发中,HTML、CSS和JavaScript是构建前端用户界面的核心技术。HTML(超文本标记语言)用于定义网页的结构和内容,CSS(层叠样式表)负责网页的样式和布局,而JavaScript用于实现网页的动态功能和交互性。
知识点三:Node.js技术
Node.js是一个基于Chrome V8引擎的JavaScript运行时环境,它允许开发者使用JavaScript来编写服务器端代码。Node.js是非阻塞的、事件驱动的I/O模型,适合构建高性能和高并发的网络应用。它广泛用于Web应用的后端开发,尤其适合于I/O密集型应用,如在线聊天应用、实时推送服务等。
知识点四:原型开发
原型开发是一种设计方法,用于快速构建一个可交互的模型或样本来展示和测试产品的主要功能。在软件开发中,原型通常用于评估概念的可行性、收集用户反馈,并用作后续迭代的基础。原型开发可以帮助团队和客户理解产品将如何运作,并尽早发现问题。
知识点五:设计探索
设计探索是指在产品设计过程中,通过创新思维和技术手段来探索各种可能性。在Web应用程序开发中,这可能意味着考虑用户界面设计、用户体验(UX)和用户交互(UI)的创新方法。设计探索的目的是创造一个既实用又吸引人的应用程序,可以提供独特的价值和良好的用户体验。
知识点六:评估可用性和有效性
评估可用性和有效性是指在开发过程中,对应用程序的可用性(用户能否容易地完成任务)和有效性(应用程序是否达到了预定目标)进行检查和测试。这通常涉及用户测试、反馈收集和性能评估,以确保最终产品能够满足用户的需求,并在技术上实现预期的功能。
知识点七:HTML/CSS/JavaScript和Node.js的特定部分使用
在Web应用程序开发中,开发者需要熟练掌握HTML、CSS和JavaScript的基础知识,并了解如何将它们与Node.js结合使用。例如,了解如何使用JavaScript的AJAX技术与服务器端进行异步通信,或者如何利用Node.js的Express框架来创建RESTful API等。
知识点八:应用领域的广泛性
本文件提到的“基准要求”中提到,通过两层Web应用程序可以实现多种应用领域,如游戏、物联网(IoT)、组织工具、商务、媒体等。这说明了Web技术的普适性和灵活性,它们可以被应用于构建各种各样的应用程序,满足不同的业务需求和用户场景。
知识点九:创造性界限
在开发Web应用程序时,鼓励开发者和他们的合作伙伴探索创造性界限。这意味着在确保项目目标和功能要求得以满足的同时,也要勇于尝试新的设计思路、技术方案和用户体验方法,从而创造出新颖且技术上有效的解决方案。
知识点十:参考资料和文件结构
文件名称列表中的“a2-shortstack-master”暗示了这是一个与作业2相关的项目文件夹或代码库。通常,在这样的文件夹结构中,可以找到HTML文件、样式表(CSS文件)、JavaScript脚本以及可能包含Node.js应用的服务器端代码。开发者可以使用这些文件来了解项目结构、代码逻辑和如何将各种技术整合在一起以创建一个完整的工作应用程序。
管理建模和仿真的文件
管理Boualem Benatallah引用此版本:布阿利姆·贝纳塔拉。管理建模和仿真。约瑟夫-傅立叶大学-格勒诺布尔第一大学,1996年。法语。NNT:电话:00345357HAL ID:电话:00345357https://theses.hal.science/tel-003453572008年12月9日提交HAL是一个多学科的开放存取档案馆,用于存放和传播科学研究论文,无论它们是否被公开。论文可以来自法国或国外的教学和研究机构,也可以来自公共或私人研究中心。L’archive ouverte pluridisciplinaire
计算机体系结构概述:基础概念与发展趋势
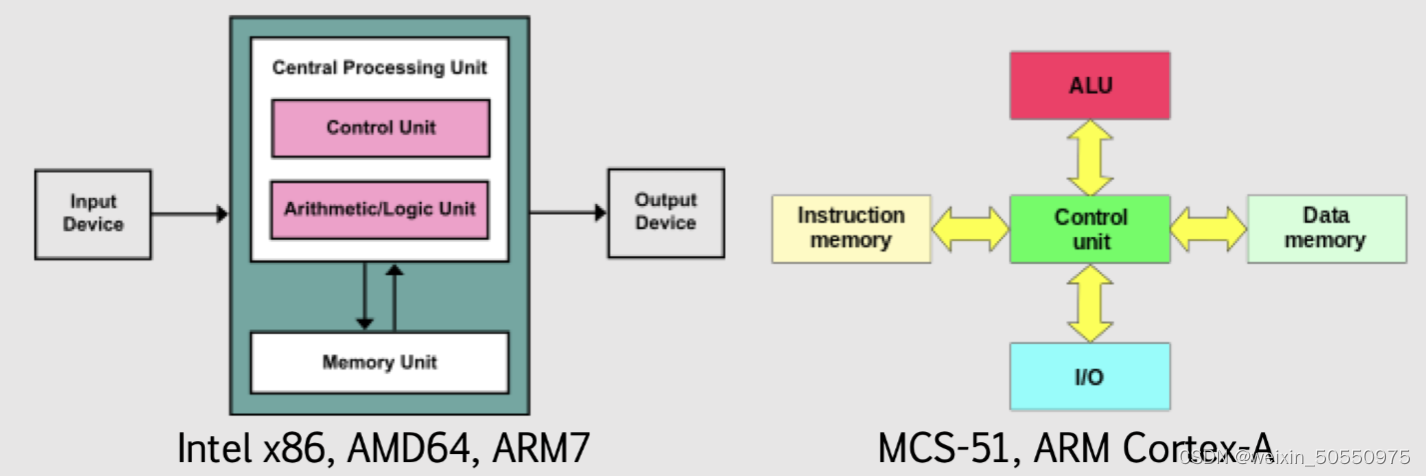
# 摘要
计算机体系结构作为计算机科学的核心领域,经历了从经典模型到现代新发展的演进过程。本文从基本概念出发,详细介绍了冯·诺依曼体系结构、哈佛体系结构以及RISC和CISC体系结构的设计原则和特点。随后,文章探讨了现代计算机体系结构的新发展,包括并行计算体系结构、存储体系结构演进和互连网络的发展。文中还深入分析了前沿技术如量子计算机原理、脑启发式计算以及边缘计算和物联网的结合。最后,文章对计算机体系结构未来的发展趋
int a[][3]={{1,2},{4}}输出这个数组
`int a[][3]={{1,2},{4}}` 定义了一个二维数组,它有两行三列,但是只填充了前两行的数据。第一行是 {1, 2},第二行是 {4}。
当你尝试输出这个数组时,需要注意的是,由于分配的空间是固定的,所以对于只填充了两行的情况,第三列是未初始化的,通常会被默认为0。因此,常规的打印方式会输出类似这样的结果:
```
a[0][0]: 1
a[0][1]: 2
a[1][0]: 4
a[1][1]: (未初始化,可能是0)
```
如果需要展示所有元素,即使是未初始化的部分,可能会因为语言的不同而有不同的显示方式。例如,在C++或Java中,你可以遍历整个数组来输出:
`
勒玛算法研讨会项目:在线商店模拟与Qt界面实现
资源摘要信息: "lerma:算法研讨会项目"
在本节中,我们将深入了解一个名为“lerma:算法研讨会项目”的模拟在线商店项目。该项目涉及多个C++和Qt框架的知识点,包括图形用户界面(GUI)的构建、用户认证、数据存储以及正则表达式的应用。以下是项目中出现的关键知识点和概念。
标题解析:
- lerma: 看似是一个项目或产品的名称,作为算法研讨会的一部分,这个名字可能是项目创建者或组织者的名字,用于标识项目本身。
- 算法研讨会项目: 指示本项目是一个在算法研究会议或研讨会上呈现的项目,可能是为了教学、展示或研究目的。
描述解析:
- 模拟在线商店项目: 项目旨在创建一个在线商店的模拟环境,这涉及到商品展示、购物车、订单处理等常见在线购物功能的模拟实现。
- Qt安装: 项目使用Qt框架进行开发,Qt是一个跨平台的应用程序和用户界面框架,所以第一步是安装和设置Qt开发环境。
- 阶段1: 描述了项目开发的第一阶段,包括使用Qt创建GUI组件和实现用户登录、注册功能。
- 图形组件简介: 对GUI组件的基本介绍,包括QMainWindow、QStackedWidget等。
- QStackedWidget: 用于在多个页面或视图之间切换的组件,类似于标签页。
- QLineEdit: 提供单行文本输入的控件。
- QPushButton: 按钮控件,用于用户交互。
- 创建主要组件以及登录和注册视图: 涉及如何构建GUI中的主要元素和用户交互界面。
- QVBoxLayout和QHBoxLayout: 分别表示垂直和水平布局,用于组织和排列控件。
- QLabel: 显示静态文本或图片的控件。
- QMessageBox: 显示消息框的控件,用于错误提示、警告或其他提示信息。
- 创建User类并将User类型向量添加到MainWindow: 描述了如何在项目中创建用户类,并在主窗口中实例化用户对象集合。
- 登录和注册功能: 功能实现,包括验证电子邮件、用户名和密码。
- 正则表达式的实现: 使用QRegularExpression类来验证输入字段的格式。
- 第二阶段: 描述了项目开发的第二阶段,涉及数据的读写以及用户数据的唯一性验证。
- 从JSON格式文件读取和写入用户: 描述了如何使用Qt解析和生成JSON数据,JSON是一种轻量级的数据交换格式,易于人阅读和编写,同时也易于机器解析和生成。
- 用户名和电子邮件必须唯一: 在数据库设计时,确保用户名和电子邮件字段的唯一性是常见的数据完整性要求。
- 在允许用户登录或注册之前,用户必须选择代表数据库的文件: 用户在进行登录或注册之前需要指定一个包含用户数据的文件,这可能是项目的一种安全或数据持久化机制。
标签解析:
- C++: 标签说明项目使用的编程语言是C++。C++是一种高级编程语言,广泛应用于软件开发领域,特别是在性能要求较高的系统中。
压缩包子文件的文件名称列表:
- lerma-main: 这可能是包含项目主要功能或入口点的源代码文件或模块的名称。通常,这样的文件包含应用程序的主要逻辑和界面。
通过这些信息,可以了解到该项目是一个采用Qt框架和C++语言开发的模拟在线商店应用程序,它不仅涉及基础的GUI设计,还包括用户认证、数据存储、数据验证等后端逻辑。这个项目不仅为开发者提供了一个实践Qt和C++的机会,同时也为理解在线商店运行机制提供了一个良好的模拟环境。