ML350G4服务器电源线路图解析
需积分: 5 38 浏览量
更新于2024-08-05
收藏 441KB PDF 举报
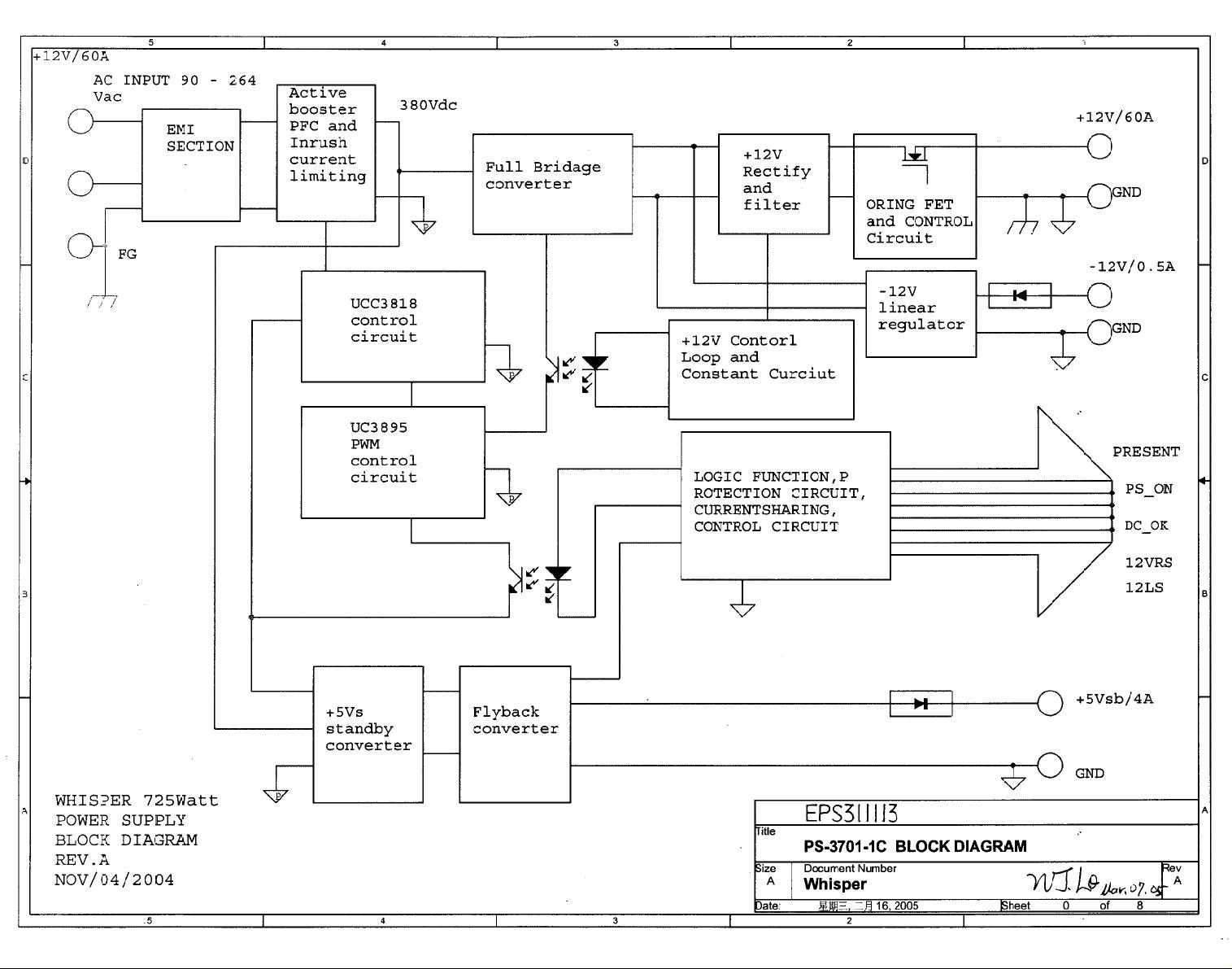
"ML350G4服务器电源线路图PS-3701-1C_10.pdf" 描述了一款戴尔ML350G4服务器的电源模块,其中涉及了电源输入、电压转换、保护电路以及控制信号等多个关键部分的设计。该文档是电源线路的详细图纸,包含各种连接器、电子元件和控制信号的标识。
ML350G4服务器电源主要由以下几个核心部分组成:
1. **输入电源接口**:通常包括RTN(返回线)端口,提供电源输入的负极连接,如J4连接器,用于连接到电源插座。
2. **电压转换**:电源模块提供多种电压输出,如12VOUT和5VSB,这些电压用于服务器内部不同组件的供电。12VOUT是主要的工作电压,而5VSB则是待机电源,即使在服务器关闭时也保持供电,以支持基本的管理功能。
3. **保护电路**:包括PFC(功率因数校正)和过压/欠压保护。PFC_OUT2和PFC_OK信号监控输入电源的功率因数,确保高效能和低谐波干扰。过压和欠压保护则通过监测+12VDET和+5VSB_DET来防止电源或设备受到损坏。
4. **控制信号**:如PSON(电源开启),DCOK(直流就绪),PGDET(电源良好检测)等,这些信号负责监测电源状态并与其他系统组件通信。例如,PSON信号用于启动电源,而DCOK表示电源已稳定并准备好为系统供电。
5. **热管理和风扇控制**:VFAN2和GND_FAN等连接器管理服务器内部的风扇,以确保散热。ACL(自动冷却逻辑)可能涉及温度传感器和风扇速度控制,以优化冷却效率。
6. **电源继电器**:如PWR-RLY和VCC-RLY,它们在电源开启和关闭过程中切换电源路径,确保安全可靠的电源传输。
7. **接地**:GND_FAN、P_GND、VCC1等标识不同的接地连接,保证电路的稳定性。
8. **电流感应**:ISEN和T202.6-R209等元件可能涉及到电流检测,用于监控电源负载和保护电路。
9. **电源输出监测**:+12VOUT_58A可能表示一个大电流的12V输出,通过R483-R468等电阻进行电流采样和监测。
10. **其他功能**:如+16VSB可能为辅助电源,提供额外的待机电压,而ACL_MB可能与主板上的自动冷却功能相关。
这个电源线路图是服务器维护和故障排查的重要参考,它揭示了电源模块如何将输入电源转换为服务器所需的多种电压,并提供了监控和保护机制以确保系统的稳定运行。对于技术人员来说,理解这个图可以帮助诊断电源问题,优化性能,以及进行必要的硬件升级或更换。
2023-06-09 上传
2020-03-21 上传
2023-10-23 上传
2023-07-12 上传
2023-07-12 上传
2023-06-10 上传
2023-06-12 上传
2023-05-24 上传
2023-06-10 上传
qxdnzx
- 粉丝: 0
- 资源: 25
最新资源
- Material Design 示例:展示Android材料设计的应用
- 农产品供销服务系统设计与实现
- Java实现两个数字相加的基本代码示例
- Delphi代码生成器:模板引擎与数据库实体类
- 三菱PLC控制四台电机启动程序解析
- SSM+Vue智能停车场管理系统的实现与源码分析
- Java帮助系统代码实现与解析
- 开发台:自由职业者专用的MEAN堆栈客户端管理工具
- SSM+Vue房屋租赁系统开发实战(含源码与教程)
- Java实现最大公约数与最小公倍数算法
- 构建模块化AngularJS应用的四边形工具
- SSM+Vue抗疫医疗销售平台源码教程
- 掌握Spring Expression Language及其应用
- 20页可爱卡通手绘儿童旅游相册PPT模板
- JavaWebWidget框架:简化Web应用开发
- 深入探讨Spring Boot框架与其他组件的集成应用