荟萃分析校准误差揭示基因座可靠性:全基因组关联研究的新挑战
20 浏览量
更新于2024-06-18
收藏 1.38MB PDF 举报
荟萃分析是全基因组关联研究(GWAS)中常用的一种方法,它通过对多个独立研究的结果进行汇总,以增强单个研究发现的统计力量,从而识别与疾病相关的基因座。然而,一项由Masahiro Kanai、Roy Elzur、Wei Zhou等研究人员发表在《细胞基因组学》上的最新研究揭示了一个重要的问题:在应用标准工具对GWAS的荟萃分析进行精细定位时,可能会出现不可靠的结果,这被称为关联校准误差。
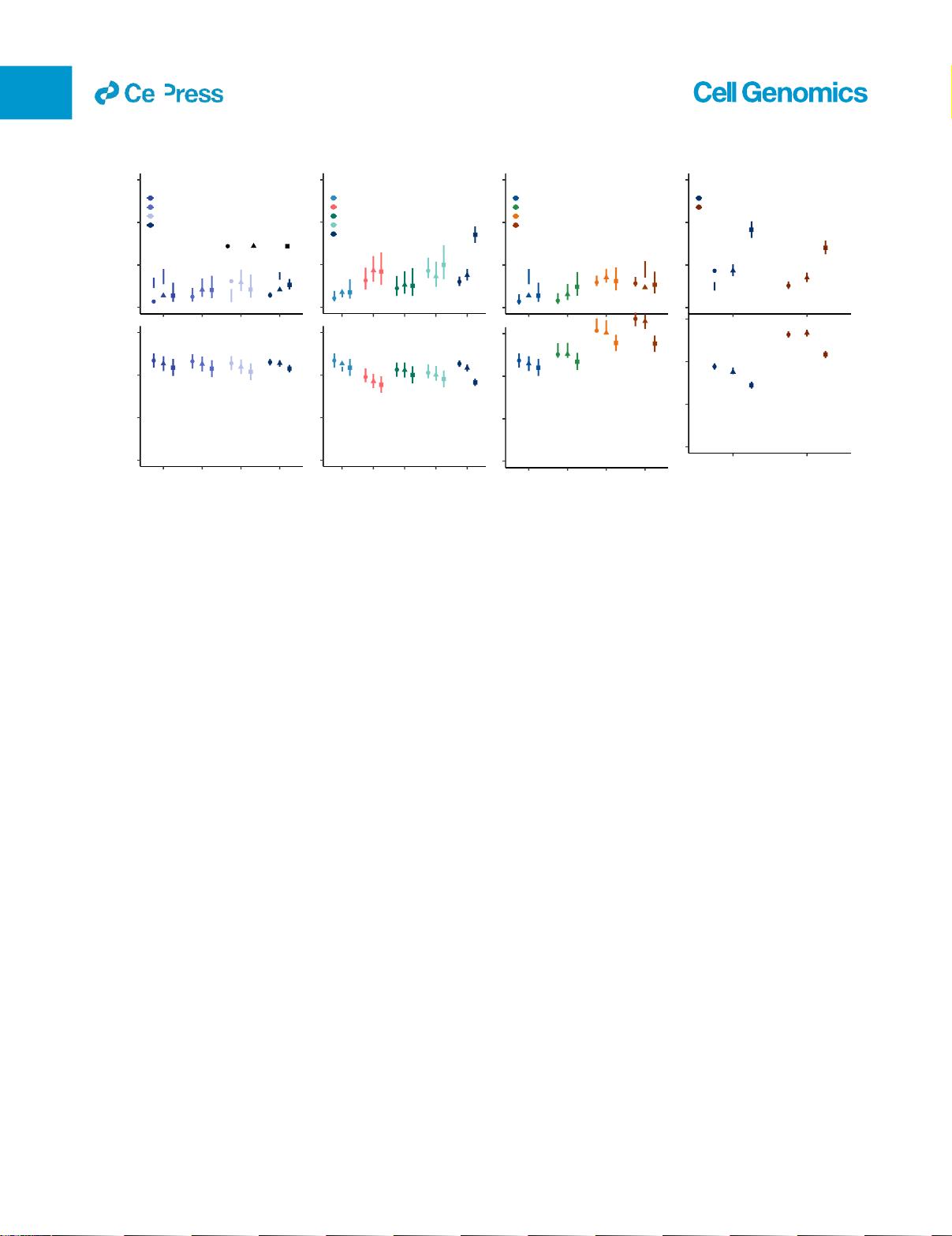
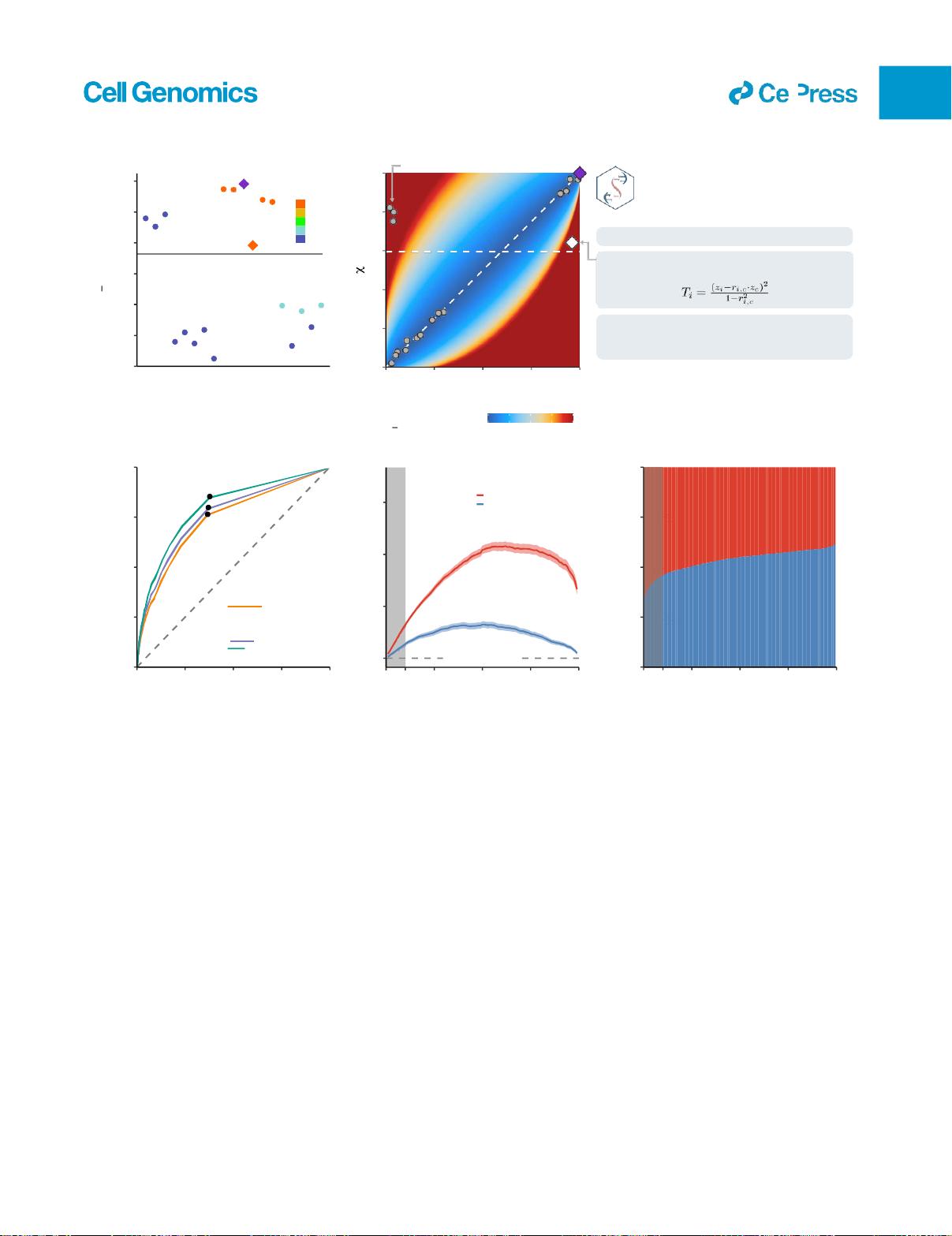
该研究通过大规模的模拟和真实数据的深入分析,指出在跨异质队列的精细映射过程中,现有的校准方法可能不足以准确捕捉到所有潜在的变异效应。作者们使用了名为SLALOM(Single Loci Analysis of Linkage Disequilibrium Maps)的方法来识别那些在荟萃分析汇总统计中可能导致误导的可疑基因座。他们发现,这些可疑的位点可能是由于局部遗传连锁不平衡(Linkage Disequilibrium, LD)关联统计中的离群值检测导致的。
SLALOM不仅帮助识别了在单变量分辨率下可能存在的问题,还提供了一种新的视角来评估在多组数据整合时可能出现的校准偏差。研究人员强调了在进行基因座定位时,有必要重新考虑现有的统计方法,并可能需要开发更精确的校准技术,以确保在高通量遗传学研究中的结果可靠性。
亮点部分包括:
1. **校准误差的影响**: 该研究揭示了荟萃分析中的潜在问题,可能会导致对因果变异的错误推断,从而影响我们对疾病风险因素的理解。
2. **SLALOM的应用**: SLALOM作为一种强大的工具,可以帮助识别和处理这些误差,通过识别和排除可能受局部LD影响的异常基因座。
3. **可疑位点分析**:研究人员对荟萃分析中大量出现的可疑基因座进行了深入分析,以了解其在实际遗传背景下的表现。
4. **统计离群点检测挑战**: 研究强调了在处理遗传数据时需要谨慎对待离群点检测,因为它可能放大校准误差。
5. **国际合作**: 作者来自全球生物库元分析计划,表明了跨学科合作的重要性,以解决此类复杂遗传学问题。
Kanai等人的研究为我们理解全基因组关联研究中的校准误差提供了新视角,这对于提高未来遗传学研究的准确性和可靠性具有重要意义。为了确保遗传关联研究的可信度,有必要进一步改进和优化校准技术,以减少这类潜在误差的影响。
2020-05-15 上传
2021-05-30 上传
2021-06-29 上传
2021-03-10 上传
2021-05-29 上传
2021-02-09 上传
2021-05-11 上传
点击了解资源详情
cpongm
- 粉丝: 5
- 资源: 2万+