Bioperl课程:序列分析与数据库操作
需积分: 9 42 浏览量
更新于2024-07-17
收藏 465KB PDF 举报
"Bioperl课程 - Catherine Letondal 和 Katja Schuerer 的PDF教程"
Bioperl 是一个开源的Perl模块集合,用于生物信息学应用开发。本课程由Catherine Letondal和Katja Schuerer撰写,旨在介绍Bioperl模块,并通过实例和练习帮助学习者掌握其用法。课程内容已更新至Bioperl 1.0版,以主题形式重新组织,但主要内容保持不变。
1. **通用介绍**
- **Bioperl 文档**:Bioperl提供了详细的文档资源,帮助用户理解和使用Bioperl的各种模块。
- **通用Bioperl类**:课程涵盖了Bioperl中的基础类,这些类是构建复杂生物信息学程序的基础。
2. **序列处理**
- **Bio::SeqIO 类**:这个类用于序列输入/输出,支持多种序列格式之间的转换(如FormatConverter)。
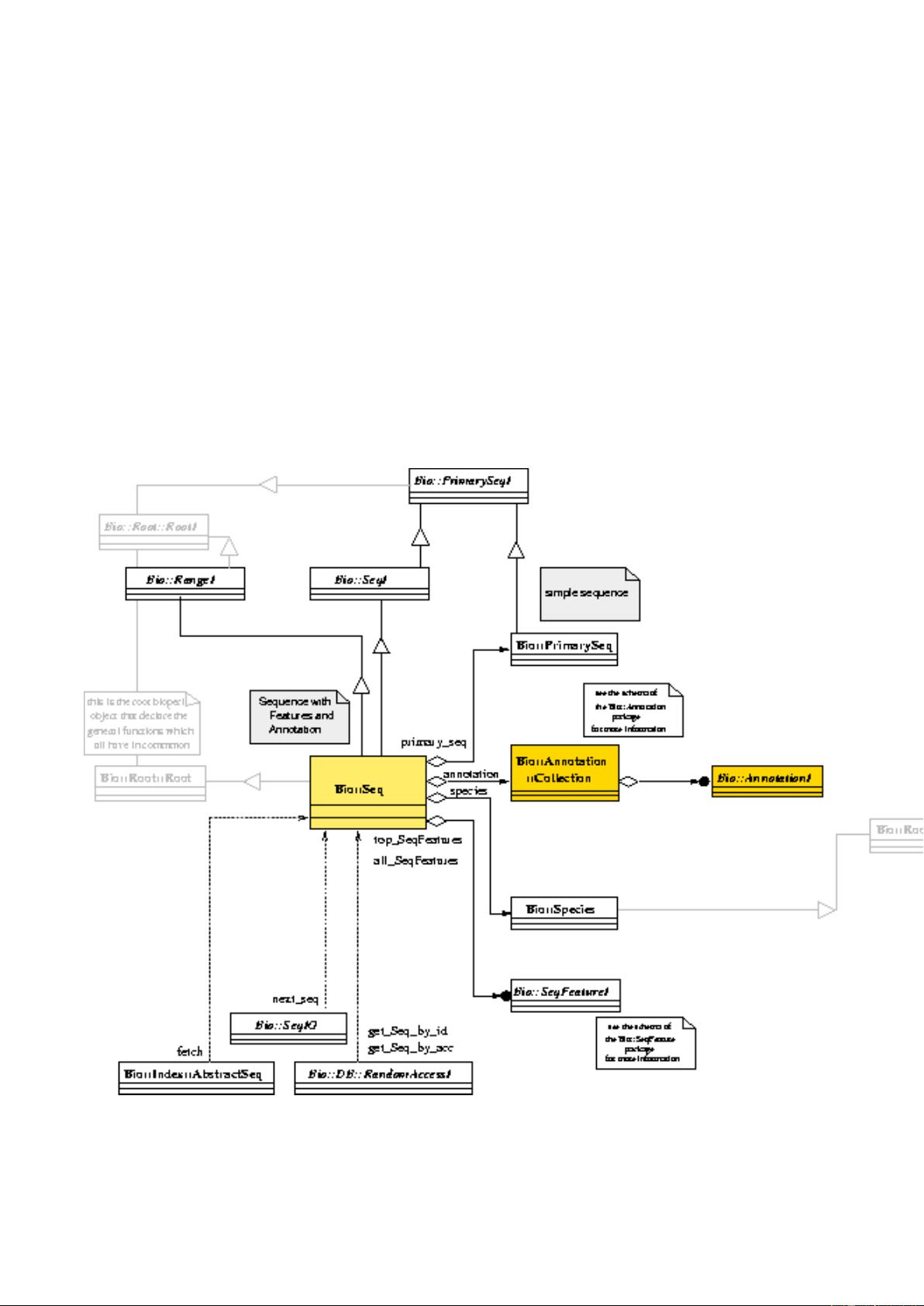
- **序列类**:包括对序列类的介绍,如何构建机制的概述,深入理解Bio::Seq类,以及Bioperl的序列类结构。
3. **序列分析工具**
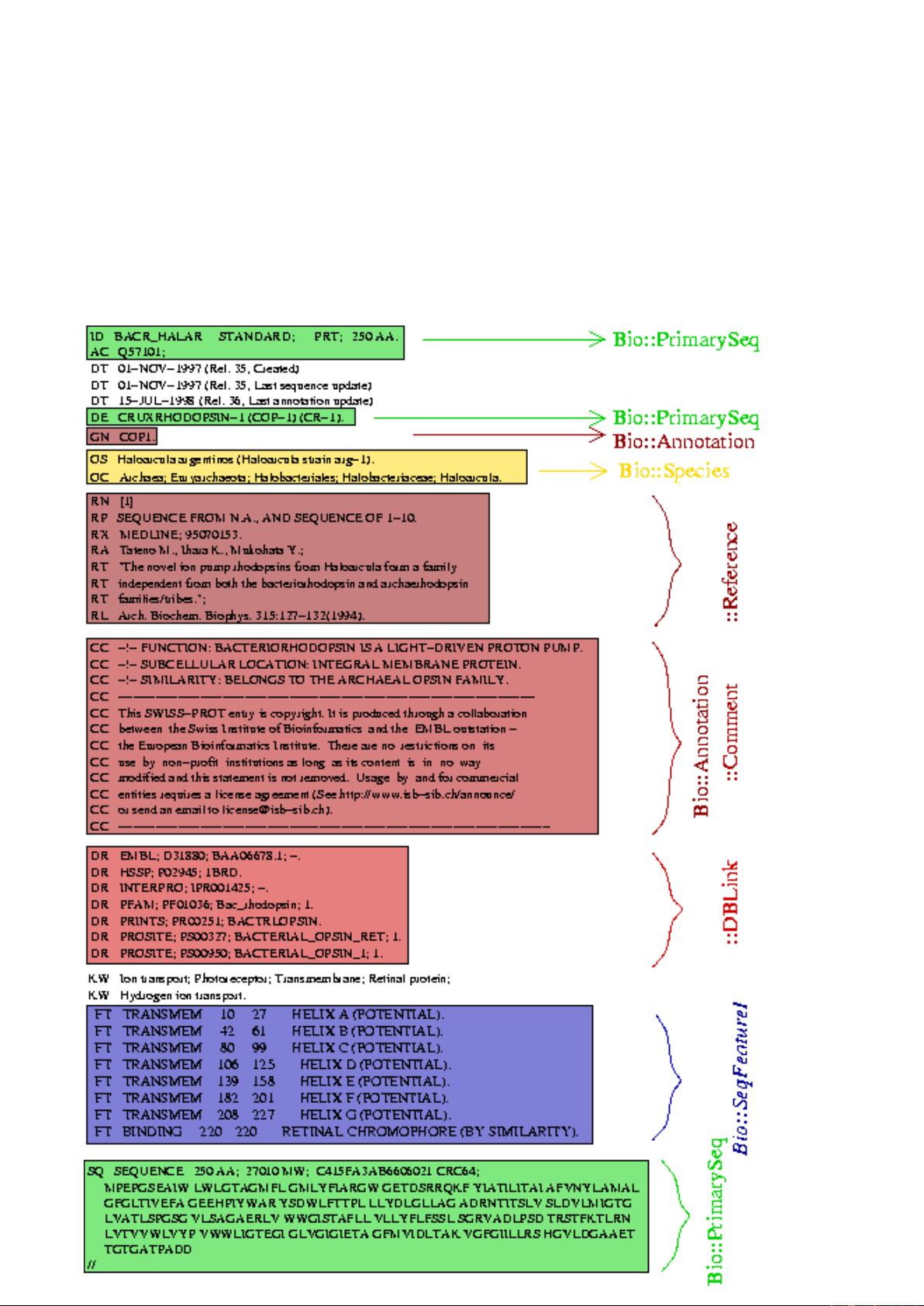
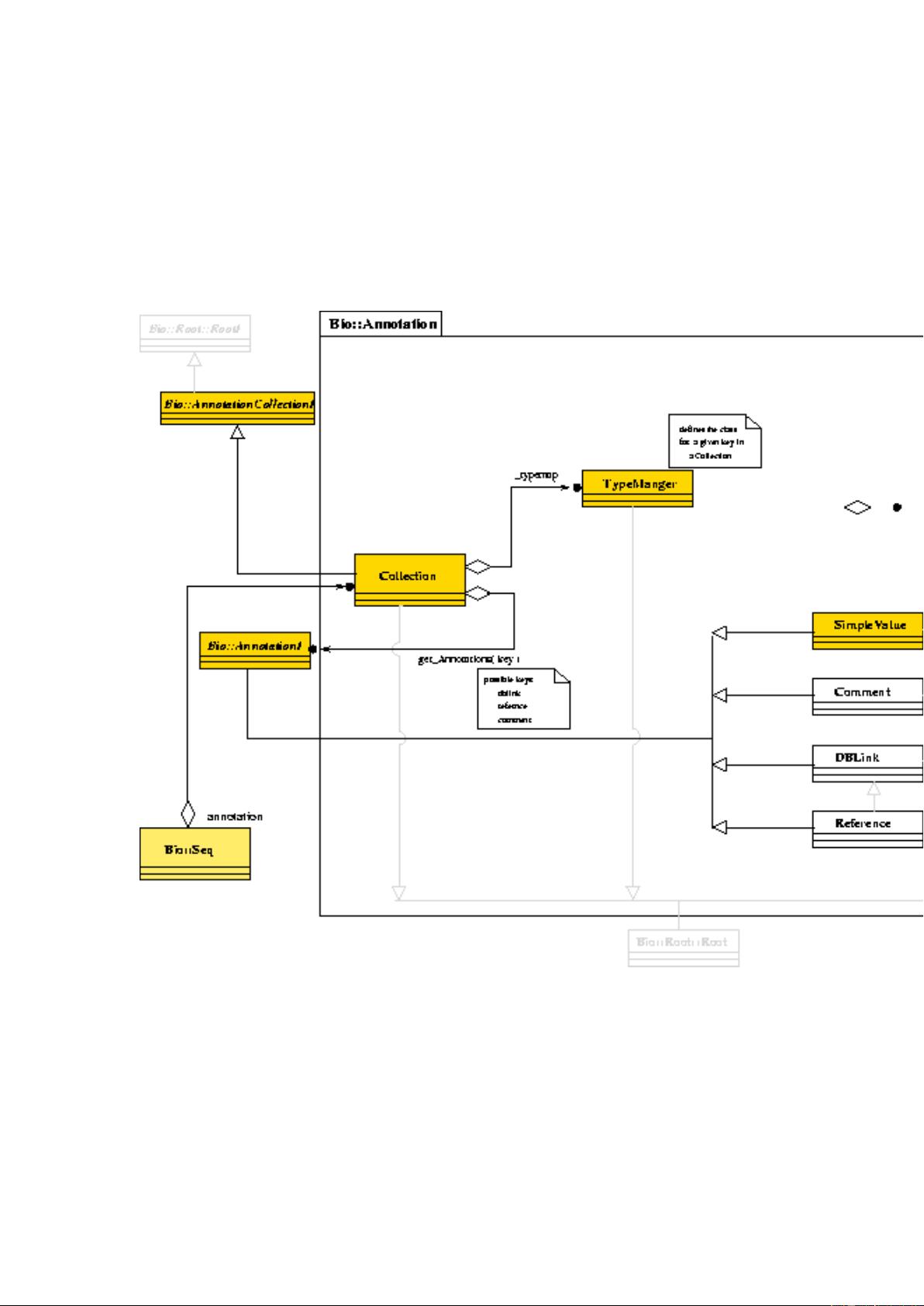
- **特征和位置类**:讨论了序列上的特征(Feature)及其位置表示。
- **编码阅读:提取CDS**:解释如何从序列中提取编码区域(Coding Sequence, CDS)。
- **标签系统**:用于存储和检索与特征相关的元数据。
- **位置类**:描述了如何表示序列上的精确位置。
- **特征的图形化视图**:展示如何可视化序列特征。
4. **比对**
- **AlignIO**:用于比对文件的输入/输出。
- **SimpleAlign**:提供简单的序列比对操作。
- **protal2dna**:将蛋白质比对转换为DNA比对的代码示例。
5. **分析**
- **BLAST**:介绍了如何使用Bioperl进行BLAST分析,包括运行BLAST、解析BLAST结果,以及BPlite家族解析器。
- **PSI-BLAST**:详述了Position-Specific Iterative BLAST的使用方法。
- **bl2seq**:用于两个序列之间的Blast比对。
- **Blast内部类结构**:探讨了Bioperl中用于BLAST分析的内部类。
6. **数据库**
- **数据库类**:介绍了访问生物信息学数据库的类。
- **使用golden访问本地数据库**:展示了如何使用特定方法连接本地数据库。
7. **Perl提醒**
- **UML**:简短介绍了统一建模语言在Perl中的应用。
- **使用Bioperl模块的Perl提示**:涵盖引用、文件句柄和流、异常处理、Getopt::Std模块、类的创建,以及BEGIN块的使用。
- **进一步理解Bioperl模块的Perl提示**:深入到模块结构和编译指令。
本课程适合那些希望使用Perl进行生物信息学编程的初学者和进阶者,通过学习,你可以掌握如何利用Bioperl高效地处理序列数据、执行比对分析和访问数据库。此外,提供的Perl编程提示将增强你对Bioperl模块的理解和使用能力。
131 浏览量
125 浏览量
117 浏览量
143 浏览量
133 浏览量
150 浏览量
345 浏览量
2008-12-16 上传
182 浏览量
drjiachen
- 粉丝: 172
- 资源: 2138
最新资源
- STM32F10xxx中文手册.zip
- LeetCode-Go:LeetCode题解
- 大学生创业者特色餐厅经营:两年三家店
- center.jquery:用可爱的动画在水平和垂直方向上居中放置任何元素。 这是一个供将来参考的jQuery插件示例
- Theme-clock:一个带有bg转换器的简单主题时钟
- generator.rar
- 多个光标:MATLAB:registered: 绘图的光标功能-matlab开发
- Zer0tolerance42.github.io:网站
- ll:缩短我的一些网站配置文件的链接
- 酒店弱电智能化系统招标文件
- soaringroad-front:个人定制化博客系统前端
- phoenix-clocks:使用 Phoenix Framework 的软实时功能显示几乎所有时区的当前时间
- AuditISX-开源
- firmware.zip
- 图书馆借书管理规划方案
- 渐入渐出动画 无闪烁 无黑底 Demo