MAPMAKER3.0基因定位教程:从数据整理到运行软件
需积分: 10 141 浏览量
更新于2024-09-10
收藏 261KB PDF 举报
"mapmaker 中文使用说明,内容详尽"
MapMaker是一款广泛应用于基因定位的软件,尤其在植物遗传学研究中非常常见。本文档将详细介绍如何使用MapMaker 3.0版本进行数据整理和软件操作。
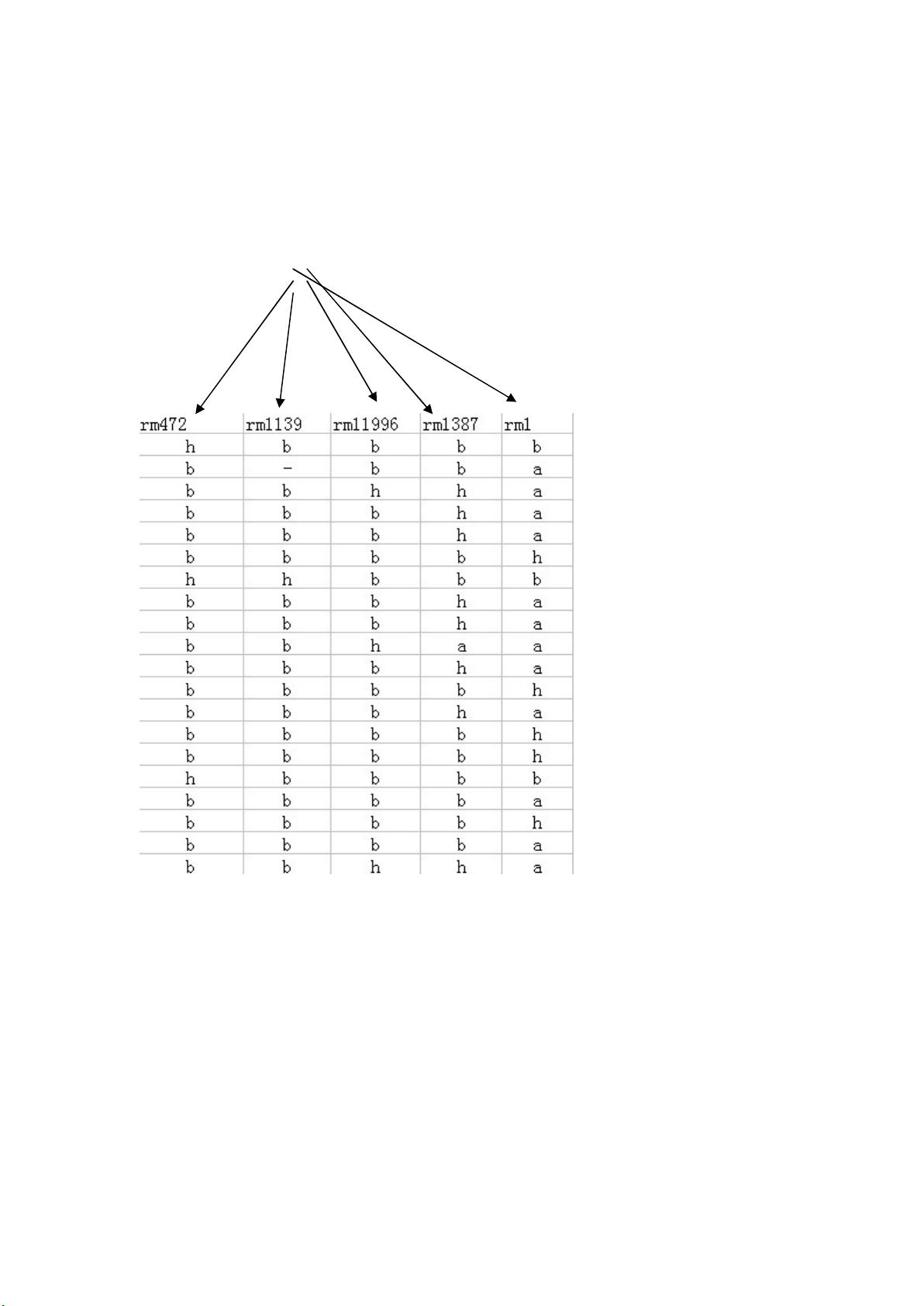
首先,数据整理是MapMaker运行前的关键步骤。在F2定位群体中,假设我们需要定位的基因为隐性,因此对不同标记的带型进行赋值。在Excel表格中,列出所有多态标记(例如:rm472、rm1139、rm11996、rm1387、rm1),并根据显性(a)、杂合(h)和隐性(b)带型分配数值。此外,添加一个特殊的“G”标记,代表目标基因,其赋值与隐性纯合体带型一致。完成整理后,将表格转置并在每个标记前加上“*”,同时在文件开头注明标记数量和定位群体株数,保存为RAW格式。
接下来,运行MapMaker 3.0软件。确保已将整理好的数据文件(例如:“dw.raw”)与软件保存在同一文件夹内。软件启动后,按照以下步骤操作:
1. 输入“pd”,然后按空格键,接着输入文件前缀名(例如:“dw”),回车。
2. 输入“photoxt”,其中“xt”是保存的文件名,回车。
3. 输入“unitcm”,设置单位为厘米,回车。
4. 输入“centfunckosambi”,选择Kosambi遗传距离计算方法,回车。
5. 输入“s{123456}”,这里的数字表示标记个数,回车。
6. 输入“group”,开始进行标记分组,回车。
每一步都将导向软件的不同界面,逐步进行基因连锁分析。MapMaker 3.0会基于输入的数据进行计算,生成连锁图谱,帮助研究人员识别和定位目标基因。
MapMaker 3.0的使用流程主要包括数据预处理和软件交互操作。正确整理数据和遵循软件指令是成功进行基因定位的基础。通过熟练掌握这些步骤,用户可以高效地利用MapMaker进行遗传图谱构建和基因定位研究。
1798 浏览量
343 浏览量
108 浏览量
434 浏览量
2024-09-21 上传
119 浏览量
487 浏览量
125 浏览量
hmilycn
- 粉丝: 0
最新资源
- C++编写的库存管理系统功能详解
- JScript与VBScript开发帮助文档指南
- Java版账务管理系统:操作简便、功能全面
- WebRTC屏幕捕获插件:tam4dummies-crx使用指南
- Matlab压缩感知图像恢复代码详解
- RCF实例:树形结构与数据CRUD操作示例
- DRF框架实现美多商城教程与代码解析
- 解决com.oracle:ojdbc14:jar:10.2.0.5.0缺失问题的方法
- 支持Win7的VC6.0绿色版安装包下载
- OpenDBDiff与SQL-DBDiff_V0.4:两款开源免费数据库对象同步工具介绍
- 鼠标驱动的动态Flash图片轮播效果
- 解决Redis连接错误并提供快速下载安装包
- JavaScript创建下拉菜单的实用教程
- 基于Asp.net和sql的酒店管理系统开发
- QQ头像制作网站教程:HTML图片动画与项目源码
- C# ASP.NET 文件与内容实时监控技术