深度学习驱动的多标签癌症通路:基因功能注释的革新方法
需积分: 9 28 浏览量
更新于2024-09-08
收藏 1.09MB PPTX 举报
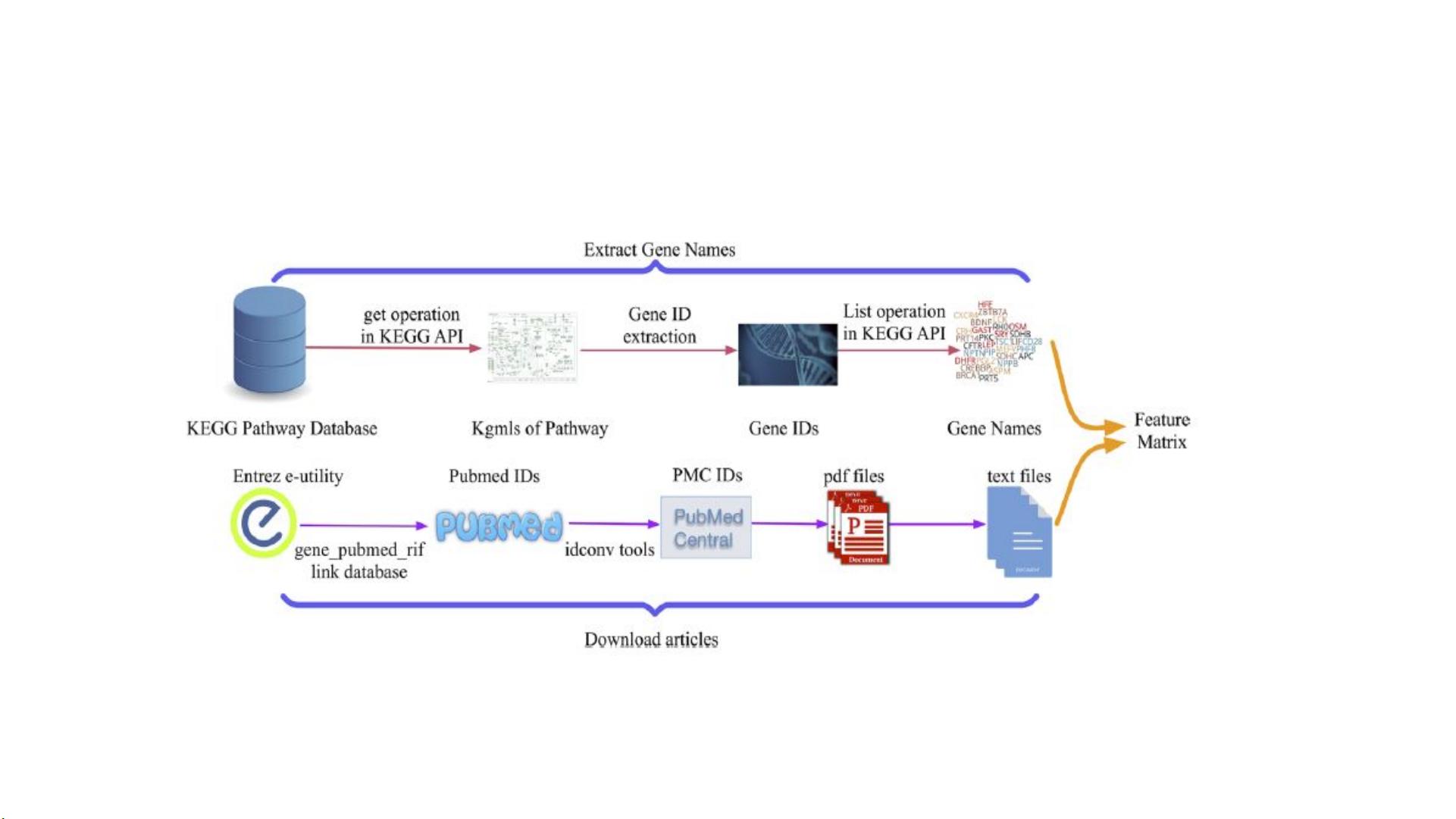
本文探讨的是"Multi-label Deep Learning for Gene Function Annotation in Cancer Pathways",即在癌症通路中利用多标签深度学习进行基因功能注释的问题。随着生物医学数据的持续增长,传统的生物定位方法因依赖资源限制而显得落后,而癌症研究中的通路分析则为揭示癌症相关生物学机制提供了有力工具。作者提出了Stacked Denoising Autoencoder Multi-Label Learning (SdaMLL) 模型,这是一个创新的深度学习模型,旨在通过捕捉输入模式的损坏部分的鲁棒性中间表示,提升基因多功能发现和通路完整性分析的准确性。SdaMLL的优势在于无需生物学家对数据进行特征工程,可以直接处理从全文生物医学出版物中获取的癌症通路数据。
特征矩阵构建过程中,研究人员收集了1144个基因的名称以及与这些基因相关的18930篇PubMedCentral文献中的信息。特征矩阵包括KEGG PATHWAY数据库中的途径图概述和基因在文章中提及的所有基因,KEGG是一个广泛使用的生物信息学数据库,由京都大学生物信息学中心创建,专注于从分子水平数据中理解生物系统的高级功能。
为了验证模型的泛化能力,采用10倍交叉验证策略,将数据集划分为10个子集,每个子集在不同的评估中轮流作为验证集。这种方法允许在有限的KEGG途径基因数据支持下,测试模型的性能。PubMedCentral (PMC) 是一个存档生物医学和生命科学文献的服务,由美国国立卫生研究院提供,用于存储电子版本的文献,它与NLM紧密合作,为研究者提供了丰富的生物医学资源。
这篇文章的主要贡献在于将深度学习应用于癌症通路的基因功能注释,展示了多标签学习模型在处理大规模生物医学数据中的潜力,同时强调了结合多种数据源和先进技术的重要性,以推动癌症研究的进步。通过SdaMLL模型和可视化工具,研究人员可以更有效地识别癌症通路中新基因的功能,并进一步加深对癌症生物学的理解。
131 浏览量
174 浏览量
335 浏览量
131 浏览量
2021-02-20 上传
203 浏览量
2021-05-24 上传
187 浏览量
点击了解资源详情
飒飒小啾啾
- 粉丝: 0
最新资源
- 网页自动刷新工具 v1.1 - 自定义时间间隔与关机
- pt-1.4协程源码深度解析
- EP4CE6E22C8芯片三相正弦波发生器设计与实现
- 高效处理超大XML文件的查看工具介绍
- 64K极限挑战:国际程序设计大赛优秀3D作品展
- ENVI软件全面应用教程指南
- 学生档案管理系统设计与开发
- 网络伪书:社区驱动的在线音乐制图平台
- Lettuce 5.0.3中文API文档完整包下载指南
- 雅虎通Yahoo! Messenger v0.8.115即时聊天功能详解
- 将Android手机转变为IP监控摄像机
- PLSQL入门教程:变量声明与程序交互
- 掌握.NET三层架构:实例学习与源码解析
- WPF中Devexpress GridControl分组功能实例分析
- H3Viewer: VS2010专用高效帮助文档查看工具
- STM32CubeMX LED与按键初始化及外部中断处理教程