Python生物信息学实践:DNA序列操作与k-mer分析
需积分: 0 191 浏览量
更新于2024-08-04
1
收藏 775KB DOCX 举报
"这篇文档是关于Python编程在生物信息学中的应用,主要涉及DNA序列处理、k-mer计数和序列比对的功能实现与测试。作者通过实例展示了如何使用Python来实现DNA序列的反向互补(Complement)和翻译(Translate)操作,以及k-mercounter功能,用于对DNA序列进行k-mer计数并比较序列间的相似性。"
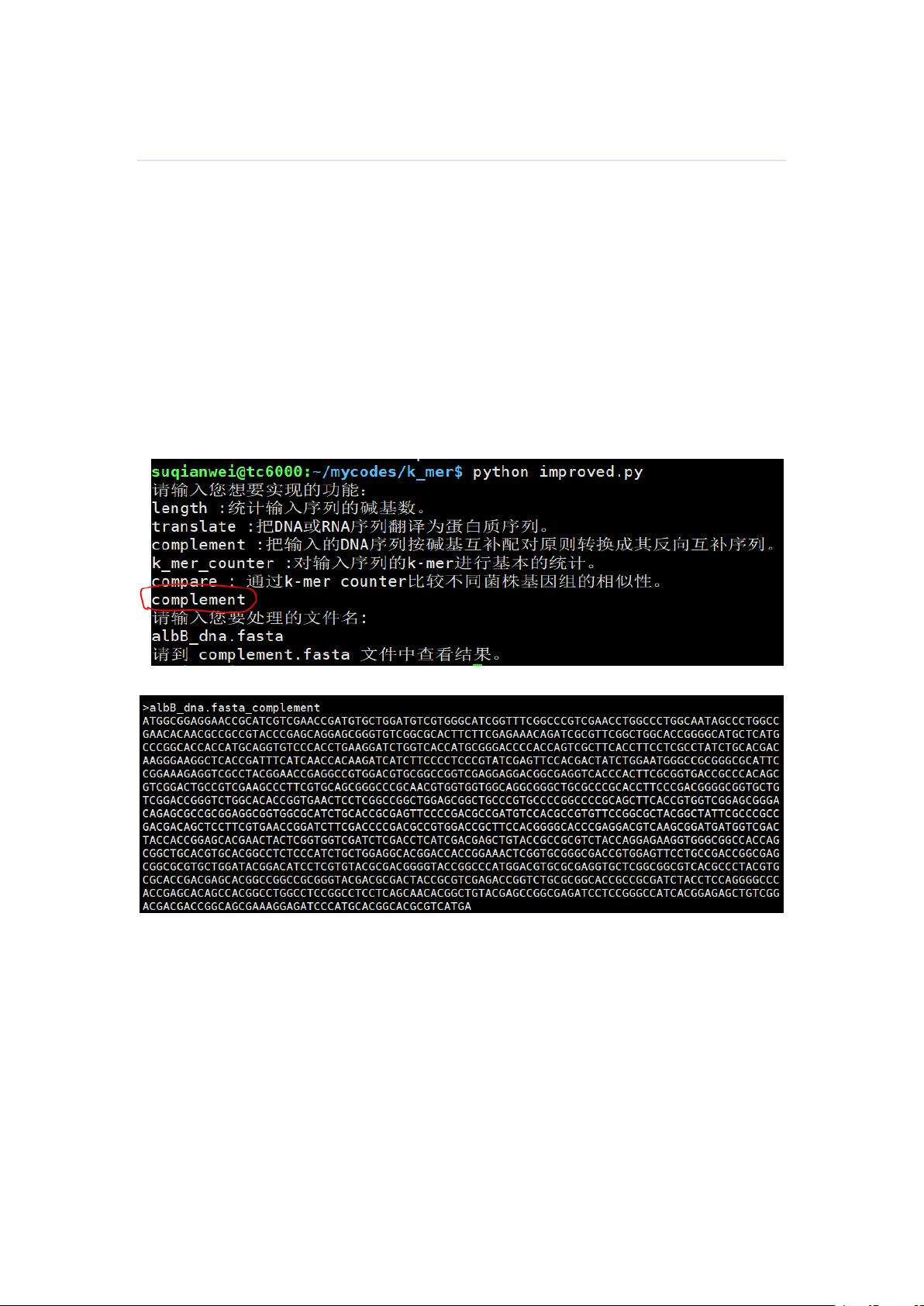
本文档首先介绍了使用Python实现的两个关键功能:Complement和Translate。Complement功能用于将DNA序列转变为反向互补序列,这是生物信息学中常用的操作,特别是在DNA配对和序列比对中。Translate功能则是将DNA序列转换为对应的蛋白质序列,这是基因表达过程的一部分,从DNA到RNA再到蛋白质的翻译。
接着,文档提到了k-mercounter功能,用于统计DNA序列中长度为k的连续子串(k-mers)的出现次数。作者测试了不同的k值对内存和计算效率的影响。当k=15时,程序能够正常运行,但随着k值增加到20,由于需要处理的组合数量剧增,导致内存错误。在k=10时,对人类基因组GRCh38.p13.fasta进行计数,程序能够在几分钟内完成,并且处理了序列中的异常字符(如N、M、R)。
此外,作者还使用k-mercounter功能对比了不同序列的相似性。通过设置k值为10,发现两个大肠杆菌序列的相似度为0.93,而caulobacterNA1000与coelicolor的相似度为0.75。这些结果可以通过热图进行可视化,以便更直观地理解序列间的关联。
文档的后续部分提到了程序的改进,包括使用argparse模块来实现交互式操作,优化处理多个文件的能力,以及添加新功能,如统计序列长度(--length)、DNA/蛋白质序列翻译(--translate)、反向互补序列(--complement)以及k-mer计数和序列相关性计算(--compare)。特别是新版本的程序现在能够处理任意数量的序列文件,增强了其在实际应用中的灵活性。
总结起来,这篇记录展示了Python在生物信息学中的强大应用,包括基础的DNA序列操作和复杂的数据分析,同时也体现了作者对程序优化和用户体验的关注。这些功能和方法对于研究基因组学、蛋白质结构以及比较基因组学等领域具有重要的价值。
4094 浏览量
8402 浏览量
点击了解资源详情
107 浏览量
2023-04-26 上传
139 浏览量
119 浏览量
2023-08-15 上传
2024-09-07 上传
Jaihwoe
- 粉丝: 21
- 资源: 350