PAUP4.0快速入门教程:系统发育分析指南
需积分: 43 147 浏览量
更新于2024-07-31
收藏 1.08MB PDF 举报
"PAUP4.0快速开始指南是一个针对初学者的教程,旨在介绍如何使用PAUP,一个用于系统发育分析的程序,支持简约法、最大似然法和距离法。该指南通过一个实际的例子数据文件进行演示,数据文件可以在指定网址获取。教程适用于Macintosh、Windows和Portable界面的用户,尽管Windows和Portable界面主要依赖命令行,Macintosh界面则包含菜单命令和命令行。在指南中,菜单选项和用户需输入的命令以不同的字体表示,以帮助用户理解和操作。启动PAUP的方式是在Mac和Windows上双击应用图标。"
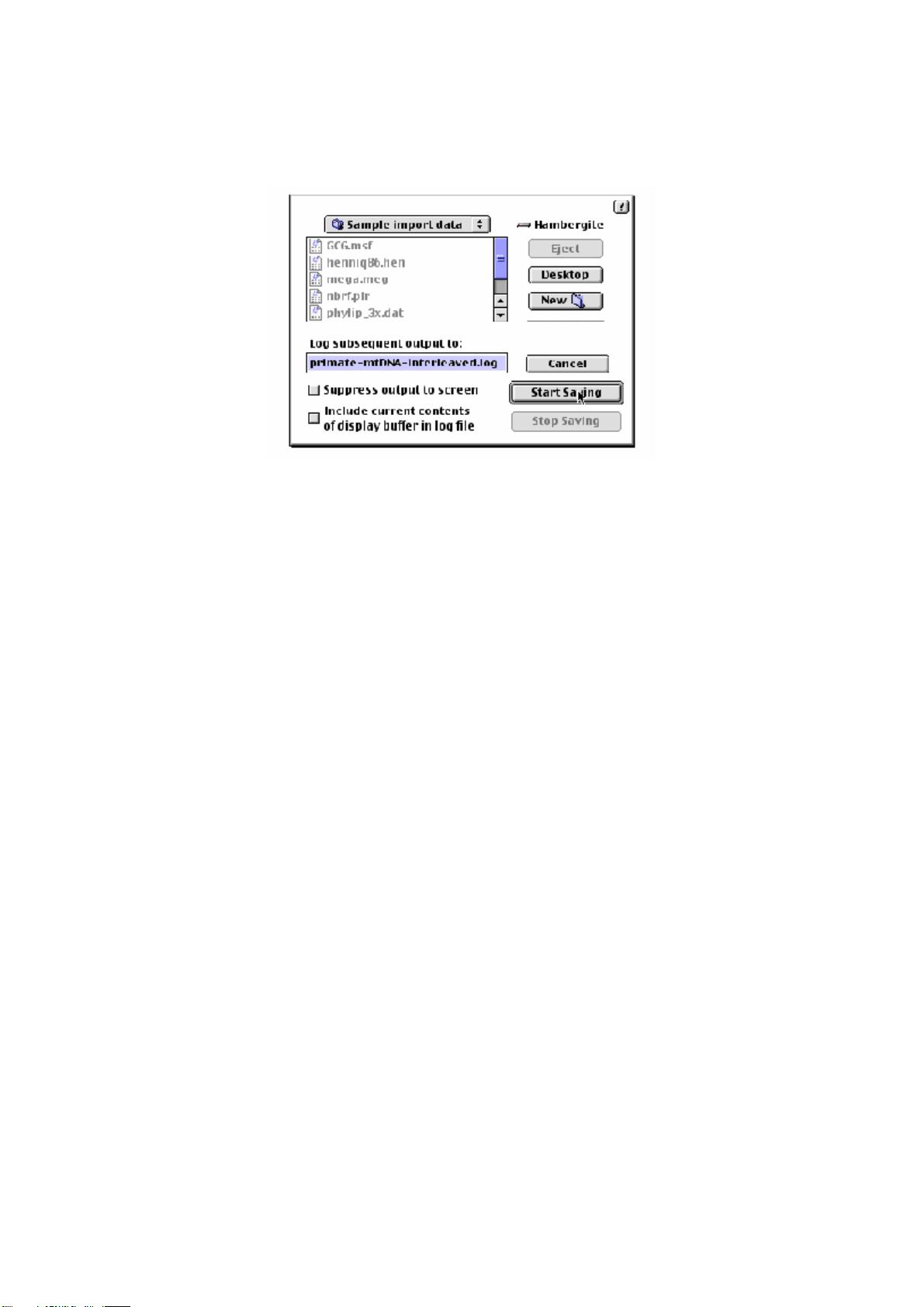
在PAUP的使用过程中,首先需要确保你有一个有效且正确格式的数据文件。PAUP可以处理包含序列数据的NEXUS文件,这是系统发育分析的标准格式。数据文件通常包含了物种的遗传序列或其他可比较的特征,这些信息将被用来构建进化树。例如,给出的示例数据文件“primate-mtDNA-interleaved.nex”包含了灵长类动物的线粒体DNA序列。
一旦PAUP启动,你可以通过菜单或命令行来操作。在Macintosh界面,用户可以点击菜单栏中的"File",然后选择"Open"来打开数据文件。在Windows和Portable界面,用户需要在命令行中输入相应的指令,如“输入: file <filename>”来加载文件。加载数据后,PAUP会进行一系列分析,包括但不限于:
1. **数据预处理**:检查数据的完整性,处理缺失数据,定义序列编码等。
2. **距离矩阵计算**:根据所选的模型计算物种间的遗传距离。
3. **进化树构建**:使用简约法、最大似然法或距离法构建初始的进化树。
4. **树的优化**:通过分支交换算法(如Fitch-Margoliash、NJ、ML等)改进树的拓扑结构,以最大化统计一致性或最小化树长度。
5. **树的检验**:进行Bootstrap或jackknife重采样以评估树的稳定性。
6. **结果输出**:保存进化树和相关统计数据,可能包括图形化的树显示。
PAUP提供了丰富的命令行选项,这使得用户可以根据研究需求自定义分析过程。例如,用户可以通过命令行指定进化模型(如GTR+I+Γ),设置搜索参数(如树的搜索次数和停止条件),或者进行更复杂的分析如混合模型分析或贝叶斯分析。
对于初次使用者,这个快速开始指南是一个很好的起点,但随着对PAUP的深入使用,建议查阅更详细的用户手册和在线资源,以便掌握更多的高级功能和技巧。PAUP的灵活性和强大的分析能力使其成为系统发育研究中不可或缺的工具。
点击了解资源详情
点击了解资源详情
点击了解资源详情
2013-07-25 上传
2010-01-12 上传
2021-03-21 上传
2022-11-06 上传
tong2200
- 粉丝: 0
- 资源: 1
最新资源
- MATLAB新功能:Multi-frame ViewRGB制作彩色图阴影
- XKCD Substitutions 3-crx插件:创新的网页文字替换工具
- Python实现8位等离子效果开源项目plasma.py解读
- 维护商店移动应用:基于PhoneGap的移动API应用
- Laravel-Admin的Redis Manager扩展使用教程
- Jekyll代理主题使用指南及文件结构解析
- cPanel中PHP多版本插件的安装与配置指南
- 深入探讨React和Typescript在Alias kopio游戏中的应用
- node.js OSC服务器实现:Gibber消息转换技术解析
- 体验最新升级版的mdbootstrap pro 6.1.0组件库
- 超市盘点过机系统实现与delphi应用
- Boogle: 探索 Python 编程的 Boggle 仿制品
- C++实现的Physics2D简易2D物理模拟
- 傅里叶级数在分数阶微分积分计算中的应用与实现
- Windows Phone与PhoneGap应用隔离存储文件访问方法
- iso8601-interval-recurrence:掌握ISO8601日期范围与重复间隔检查