"生工生物 Denovo 转录组分析结题报告:软件、实验、结果详解"
需积分: 0 106 浏览量
更新于2024-01-03
10
收藏 2.57MB PDF 举报
公司无参转录组分析结题报告
1. 名词解释
在本报告中,我们将解释以下与无参转录组分析相关的名词:
1.1 无参转录组:指的是使用高通量测序技术对生物样本中的全部转录本进行测序和分析的方法。相比有参转录组,无参转录组不需要事先提供参考基因组序列。
1.2 转录本:指的是生物基因组内的基因片段,在基因表达过程中被转录成RNA分子的序列。
1.3 测序深度:指的是对于一个生物样本进行测序的重复次数。较高的测序深度可以提供更准确和可靠的测序结果。
1.4 转录组表达量:指的是一个基因或转录本在转录组中的表达水平。转录组表达量可以通过测序数据进行定量分析得到。
2. 相关软件及数据库
在无参转录组分析过程中,我们使用了以下软件和数据库:
2.1 软件
- Trinity:用于将测序数据进行组装,得到转录本序列。
- HISAT2:用于将测序数据比对到转录本序列上。
- StringTie:用于进行基因和转录本的定量分析。
- edgeR:用于进行差异表达基因分析。
- GOseq:用于进行功能富集分析。
2.2 数据库
- NCBI数据库:用于获取基因和转录本的注释信息。
- KEGG数据库:用于进行代谢通路分析。
3. 实验流程
无参转录组分析的实验流程包括以下步骤:
3.1 样本准备:从生物样本中提取RNA,并使用高通量测序技术将RNA转化为测序文库。
3.2 测序:使用Illumina高通量测序平台对测序文库进行测序,生成原始测序数据。
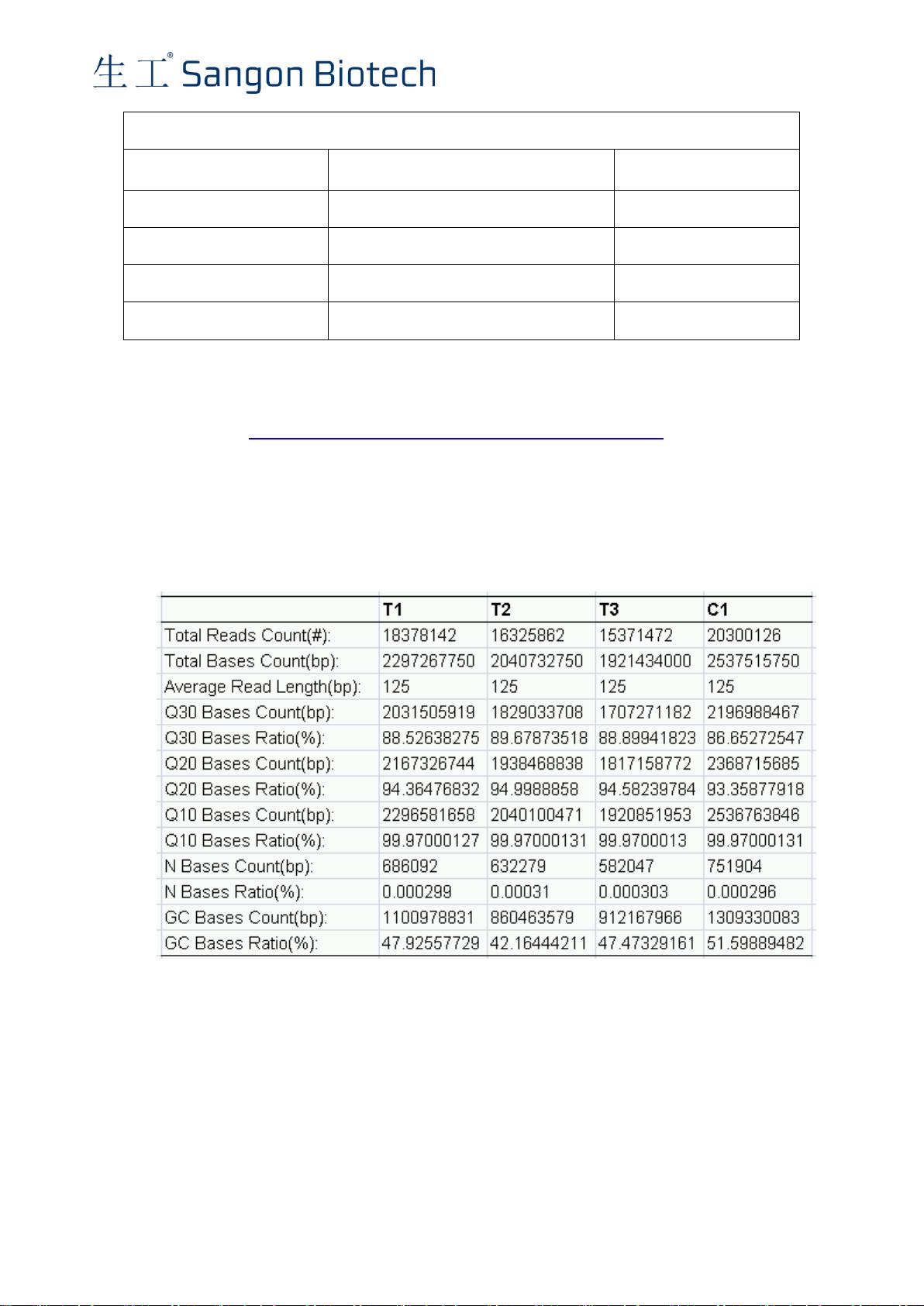
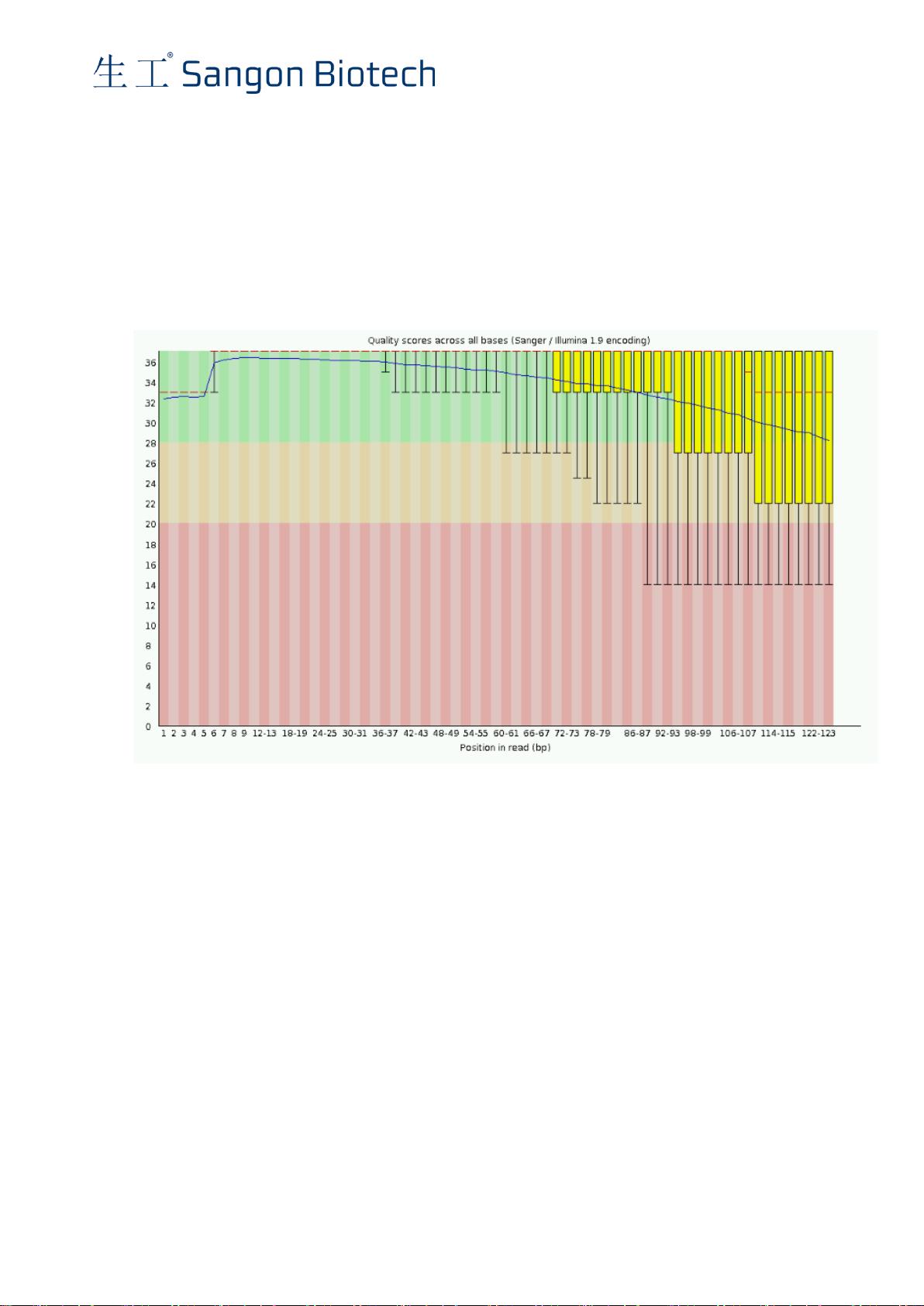
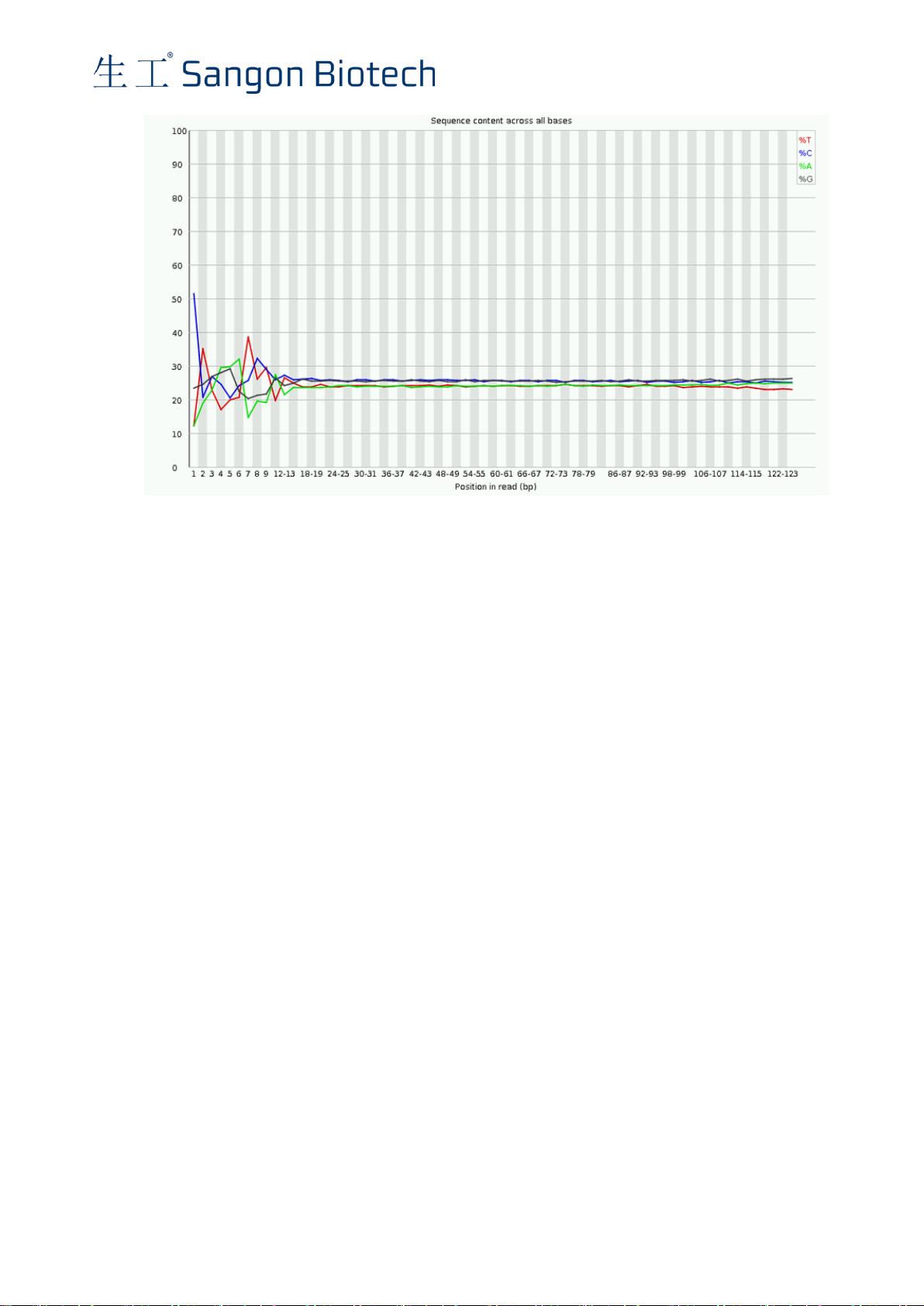
3.3 数据质控:对原始测序数据进行质量控制,包括去除低质量的测序片段和去除接头序列。
3.4 转录组组装:使用Trinity软件将质控后的测序数据进行组装,得到转录本序列。
3.5 转录本定量:使用StringTie软件对转录本进行定量,计算转录本的表达量。
4. 分析流程
无参转录组分析的分析流程包括以下步骤:
4.1 差异表达基因分析:使用edgeR软件对不同样本之间的转录本表达量进行差异分析,找出差异表达的基因。
4.2 功能富集分析:使用GOseq软件对差异表达基因进行功能富集分析,找出在特定生物学过程中富集的基因功能。
4.3 代谢通路分析:使用KEGG数据库对差异表达基因进行代谢通路分析,找出在特定代谢通路中富集的基因。
5. 分析结果展示
在本报告中,我们展示了以下无参转录组分析的结果:
- 转录本组装结果:展示了经过Trinity组装后得到的转录本序列,包括转录本长度、转录本覆盖度等信息。
- 差异表达基因分析结果:展示了在不同样本之间的差异表达基因,包括基因的表达量差异以及调控的方向。
- 功能富集分析结果:展示了差异表达基因在不同生物学过程中的功能富集情况,包括对应的GO注释和富集的P值。
- 代谢通路分析结果:展示了差异表达基因在不同代谢通路中的富集情况,包括对应的代谢通路注释和富集的P值。
综上所述,本报告详细介绍了无参转录组分析的实验和分析流程,并展示了相应的结果。这些结果可以帮助客户了解样本的转录组表达情况以及差异表达基因在功能和代谢通路上的富集情况,为进一步的研究提供重要参考。
2022-08-03 上传
2022-08-03 上传
2023-07-05 上传
2021-09-15 上传
2023-05-27 上传
2015-03-09 上传
2021-08-18 上传
2024-10-17 上传
网络小精灵
- 粉丝: 36
- 资源: 334
最新资源
- 掌握Jive for Android SDK:示例应用的使用指南
- Python中的贝叶斯建模与概率编程指南
- 自动化NBA球员统计分析与电子邮件报告工具
- 下载安卓购物经理带源代码完整项目
- 图片压缩包中的内容解密
- C++基础教程视频-数据类型与运算符详解
- 探索Java中的曼德布罗图形绘制
- VTK9.3.0 64位SDK包发布,图像处理开发利器
- 自导向运载平台的行业设计方案解读
- 自定义 Datadog 代理检查:Python 实现与应用
- 基于Python实现的商品推荐系统源码与项目说明
- PMing繁体版字体下载,设计师必备素材
- 软件工程餐厅项目存储库:Java语言实践
- 康佳LED55R6000U电视机固件升级指南
- Sublime Text状态栏插件:ShowOpenFiles功能详解
- 一站式部署thinksns社交系统,小白轻松上手