SQBC:大型密集图上的高效子图匹配方法
181 浏览量
更新于2024-07-15
收藏 481KB PDF 举报
SQBC(Subgraph Query Based on Clique feature)是一项针对大型和密集图谱的高效子图匹配方法。随着生物科学和计算机科学的最新进展,许多复杂网络,如蛋白质相互作用网络、社交网络和基因调控网络等,被建模为大型且密度较高的图结构。在这种背景下,开发出能够有效处理大规模图的子图匹配算法变得至关重要。
传统上,虽然已经提出了一些探索性的方法,但它们在面对大型和密集图时往往表现不佳,尤其是在执行效率和搜索精度之间找到平衡上。SQBC旨在解决这个问题,它结合了精心设计的 clique (完全子图) 编码与现有的 vertex (顶点) 编码策略,如文献提到的[40]中的技术。这种方法的优势在于,通过利用clique的特性,它能够在保持搜索效率的同时,更好地处理图中丰富的局部结构信息,这对于识别潜在的相关模式和相似性至关重要。
SQBC的工作流程可能包括以下几个关键步骤:
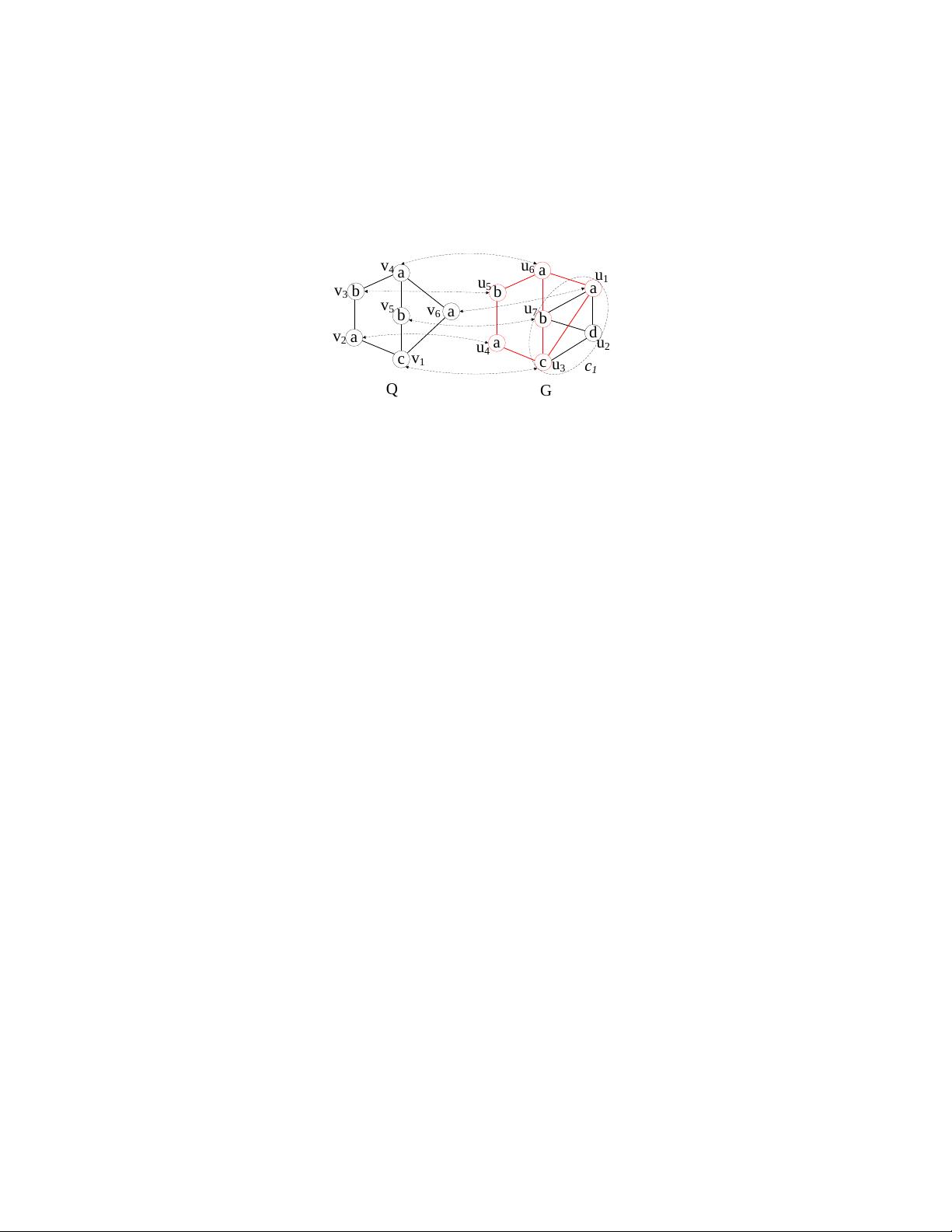
1. **Clique编码**: SQBC首先对图中的cliques进行编码,cliques作为图中的稠密区域,包含了一组顶点之间的全部连接。通过这种方式,算法可以捕捉到图中的核心聚类结构,从而减少匹配过程中的冗余计算。
2. **特征融合**: 与仅依赖于顶点编码的方法不同,SQBC将clique编码与顶点编码相结合,形成一种复合索引。这使得算法能够同时考虑全局和局部信息,提高了匹配的准确性。
3. **查询优化**: 在接收到一个子图查询时,SQBC利用预计算的clique和顶点编码快速定位可能的匹配候选。通过clique的结构,算法可以避免遍历整个图,大大减少了搜索空间。
4. **高效搜索**: 由于采用了高效的数据结构和查询策略,SQBC能够在处理大规模和密集图时保持良好的时间复杂度,即使在图中存在大量重复或相似子图的情况下,也能保持高效的匹配性能。
5. **可扩展性和适应性**: 通过设计灵活的框架,SQBC可以适应不同规模和复杂度的图,使其成为解决实际生物信息学和社交网络分析问题的理想工具。
总结来说,SQBC是一项创新的子图匹配技术,它在大型和密集图谱处理方面展现出了卓越的效率和性能,为理解和分析复杂网络提供了有力的工具。对于那些处理大规模数据集和寻求高性能查询解决方案的领域,SQBC的研究成果具有显著的实际应用价值。
点击了解资源详情
2024-12-20 上传
2024-12-20 上传
2024-12-20 上传
2024-12-20 上传
2024-12-20 上传
2024-12-20 上传
2024-12-20 上传
2024-12-20 上传
weixin_38694343
- 粉丝: 3
- 资源: 915
最新资源
- CoreOS部署神器:configdrive_creator脚本详解
- 探索CCR-Studio.github.io: JavaScript的前沿实践平台
- RapidMatter:Web企业架构设计即服务应用平台
- 电影数据整合:ETL过程与数据库加载实现
- R语言文本分析工作坊资源库详细介绍
- QML小程序实现风车旋转动画教程
- Magento小部件字段验证扩展功能实现
- Flutter入门项目:my_stock应用程序开发指南
- React项目引导:快速构建、测试与部署
- 利用物联网智能技术提升设备安全
- 软件工程师校招笔试题-编程面试大学完整学习计划
- Node.js跨平台JavaScript运行时环境介绍
- 使用护照js和Google Outh的身份验证器教程
- PHP基础教程:掌握PHP编程语言
- Wheel:Vim/Neovim高效缓冲区管理与导航插件
- 在英特尔NUC5i5RYK上安装并优化Kodi运行环境