DectICO:无对准监督宏基因组分类的特征提取与动态选择方法
89 浏览量
更新于2024-08-26
收藏 943KB PDF 举报
"DectICO是一种基于特征提取和动态选择的无对准监督宏基因组分类方法,旨在利用下一代测序技术生成的大量元基因组数据进行高效分类。该方法不依赖于已知微生物基因组,能独立区分元基因组样本中的多样成分。"
在宏基因组学领域,随着下一代测序技术的不断发展,我们能够获取到越来越多的元基因组数据,这些数据反映了不同时间和空间下的微生物群落结构。比较和分类这些具有不同微生物社区的元基因组对于理解微生物生态系统的复杂性和多样性至关重要。传统的分类方法往往依赖于序列比对,但这种方法耗时且计算量大,尤其是在处理大规模数据时。
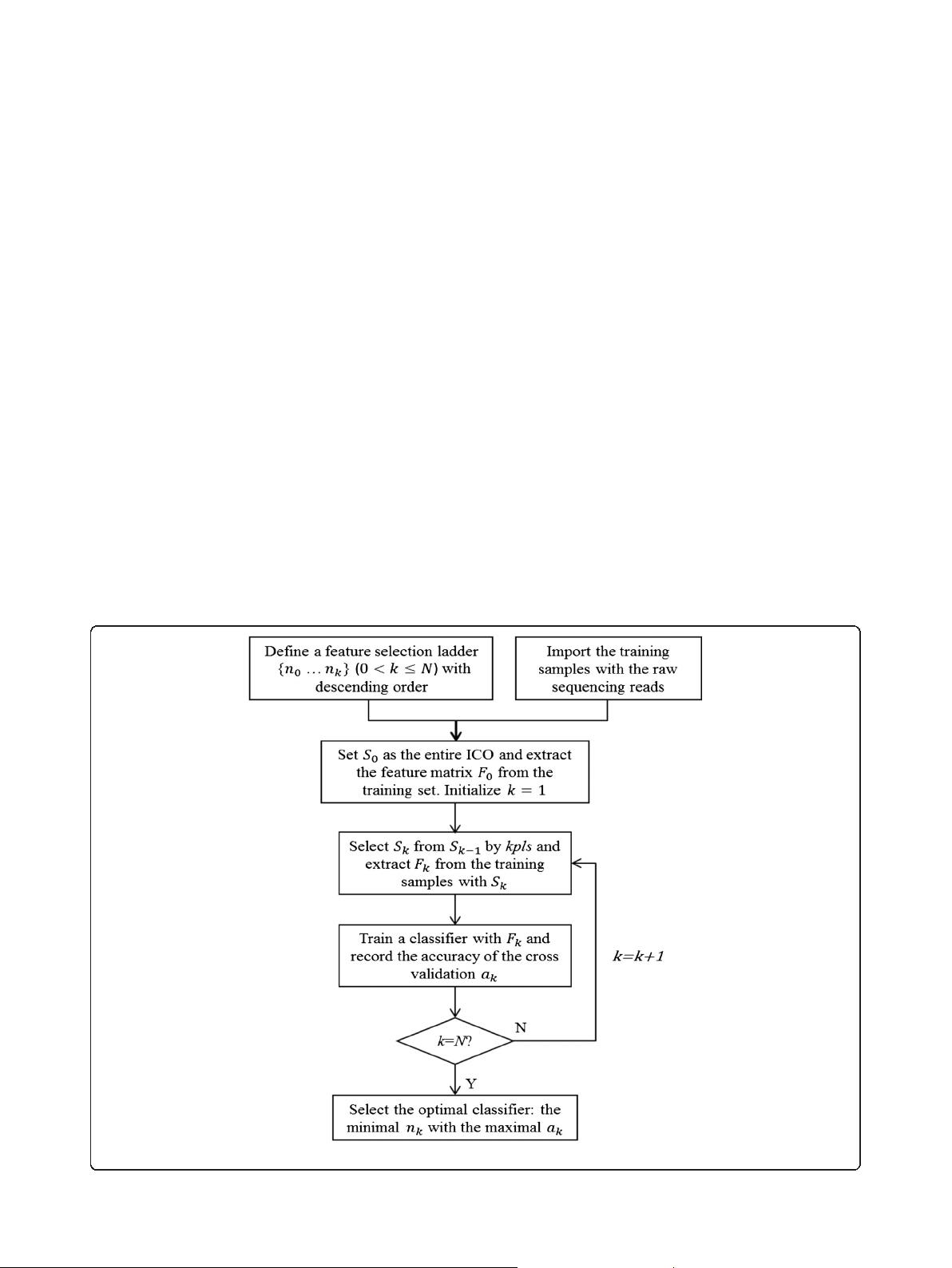
DectICO(Dectection with Intrinsically Correlated Oligonucleotides and dynamic feature Selection)是为了解决这一问题而提出的无对准监督分类方法。它主要基于寡核苷酸的内在相关性来构建特征集。寡核苷酸是DNA或RNA分子的基本单元,其特定排列可以反映基因组的特异性。DectICO通过使用核主成分分析(kernel partial least squares algorithm,KPLS)动态选择这些特征,确保选取最能区分样本的特征。
接下来,DectICO利用支持向量机(Support Vector Machine,SVM)对提取出的特征矩阵进行训练,构建分类器。SVM是一种强大的监督学习模型,适用于处理高维数据,能够有效地找到最佳决策边界,将不同类别的样本分开。
为了验证DectICO的性能,研究者在三个实际的元基因组序列数据集上进行了评估。实验结果表明,DectICO在分类准确性和效率方面表现出色,优于其他同类方法,证明了无对准策略在宏基因组分类中的优势。
总结来说,DectICO提供了一种新颖且高效的元基因组分类工具,其无对准的特性降低了对预先知识的依赖,动态特征选择策略则确保了模型的鲁棒性和准确性。这一方法对于深入研究微生物群落结构、功能以及环境响应具有重要意义,并有望在环境微生物学、医学微生物学以及生物地球化学等领域得到广泛应用。
2022-08-08 上传
2021-03-26 上传
2021-06-08 上传
2021-05-14 上传
2021-05-03 上传
2021-02-26 上传
点击了解资源详情
点击了解资源详情
点击了解资源详情
weixin_38659311
- 粉丝: 5
- 资源: 892
最新资源
- N10SG模块opencpu固件.zip
- 回收站变变变.zip易语言项目例子源码下载
- ARLAS-wui-builder:ARLAS-Wui的制造商
- ys-park-2
- electronic-ftrouter:用于运行电子的模板存储库,其中有运行路径的routex
- KottuRoti:Ant214项目游戏文件
- 前端开发css+html灯笼动画插件源代码
- pyg_lib-0.2.0+pt20-cp38-cp38-macosx_10_15_x86_64whl.zip
- tele_sign:Node.js库通过http发送消息
- CMPE:CMPE 安卓
- check-api-playground
- 判决matlab代码-self_other_moral:自我和他人道德判断的神经/行为基础项目
- 094. 2019年中国洗碗机市场年度总结报告.rar
- cornflux:用于React应用程序的调度库,可促进数据封装
- AndroidVision:在您的手机上学习图像处理
- forten:Monorepo for Overmind模块