"溯源和马尔可夫链实战:源源不断的微生物组数据分析"
需积分: 0 109 浏览量
更新于2023-12-23
收藏 1.73MB PDF 举报
36R溯源和马尔可夫链实战是一项实践性的生物信息学技术培训课程,由易生信和毕生缘共同提供。该课程讲解了如何使用SourceTracker和FEAST进行溯源分析,并介绍了马尔可夫链的基本原理和应用。本文主要对课程中涉及的样本信息表、OTU丰度表、状态转移矩阵和马尔可夫链等内容进行总结和讨论。
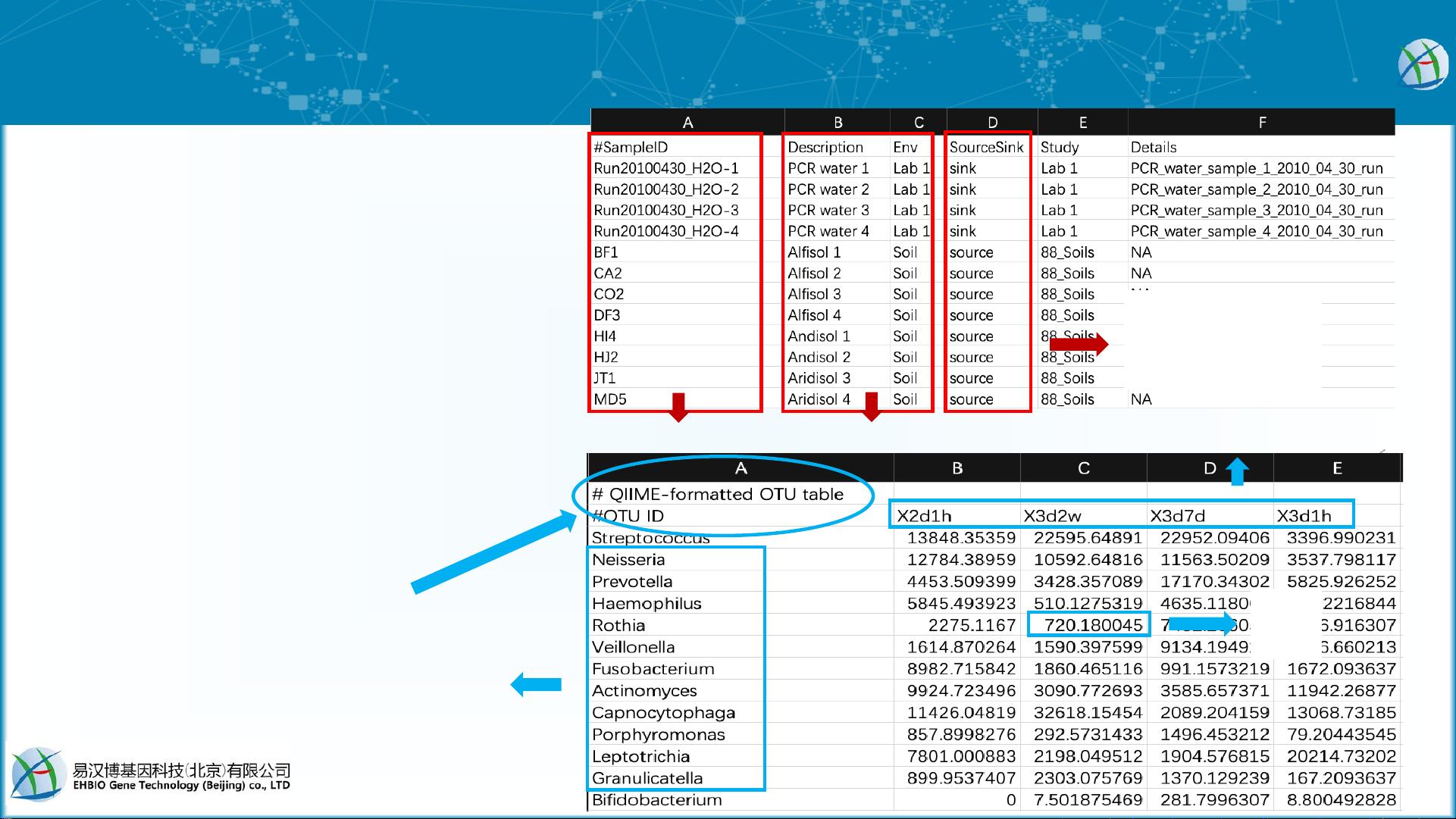
首先,样本信息表是进行溯源分析的基础数据之一。该表包括样本ID号、样本描述信息、样本所属类别等字段。在SourceTracker中,还需要包括Source和Sink的信息,用于指定每个样本的来源和溯源目标。另外,样本信息表中还需包括OTU编号或细菌分类名以及相对丰度等信息。这些信息对于实施溯源分析和构建马尔可夫链非常重要。
其次,OTU丰度表是另一个必要的输入数据。OTU是 Operational Taxonomic Units的缩写,表示具有一定相似性和聚集性的微生物分类单元。OTU丰度表记录了每个样本中不同OTU的相对丰度,通常以矩阵的形式呈现。这些数据将用于构建状态转移矩阵,描述微生物群落在不同样本之间的转移关系。
在马尔可夫链的应用中,状态转移矩阵起着至关重要的作用。状态转移矩阵描述了不同微生物状态(或类别)之间的转移概率,是马尔可夫链模型的核心。通过状态转移矩阵,我们可以模拟微生物群落在不同时间点或条件下的演变规律,帮助理解微生物组的动态变化过程。
SourceTracker和FEAST是两种常用的溯源分析工具。它们基于微生物群落的组成特征,通过模式识别和概率统计方法,尝试推断不同样本之间的关联和转移关系。在实际应用中,我们需要将样本信息表和OTU丰度表按照规定的格式输入到这些工具中,以进行溯源分析。同时,我们还可以使用R语言等编程语言,自行编写脚本来实现这些分析过程。
总的来说,36R溯源和马尔可夫链实战课程深入介绍了溯源分析和马尔可夫链在微生物组学中的应用。通过学习这些内容,我们可以更好地理解和利用微生物群落数据,揭示微生物间的相互作用和动态变化规律。这对于生物医药领域的疾病预防、治疗和健康管理具有重要意义,也有助于推动微生物组学技术的进一步发展和应用。
2022-08-08 上传
2017-12-28 上传
2018-09-26 上传
2017-01-15 上传
2024-06-11 上传
2024-02-03 上传
2023-12-03 上传
2023-04-30 上传
田仲政
- 粉丝: 20
- 资源: 332
最新资源
- NIST REFPROP问题反馈与解决方案存储库
- 掌握LeetCode习题的系统开源答案
- ctop:实现汉字按首字母拼音分类排序的PHP工具
- 微信小程序课程学习——投资融资类产品说明
- Matlab犯罪模拟器开发:探索《当蛮力失败》犯罪惩罚模型
- Java网上招聘系统实战项目源码及部署教程
- OneSky APIPHP5库:PHP5.1及以上版本的API集成
- 实时监控MySQL导入进度的bash脚本技巧
- 使用MATLAB开发交流电压脉冲生成控制系统
- ESP32安全OTA更新:原生API与WebSocket加密传输
- Sonic-Sharp: 基于《刺猬索尼克》的开源C#游戏引擎
- Java文章发布系统源码及部署教程
- CQUPT Python课程代码资源完整分享
- 易语言实现获取目录尺寸的Scripting.FileSystemObject对象方法
- Excel宾果卡生成器:自定义和打印多张卡片
- 使用HALCON实现图像二维码自动读取与解码