SPM8安装与fMRI预处理步骤详解
"这篇文档详细介绍了SPM8的安装步骤以及如何进行fMRI预处理的基本操作,主要针对Matlab2012a环境。作者柳琳来自西安电子科技大学睡眠与神经影像研究室,文档适用于组内人员和合作伙伴交流学习。"
在神经科学领域,SPM(Statistical Parametric Mapping)是一个广泛使用的软件工具,用于分析功能性磁共振成像(fMRI)数据。SPM8是其一个版本,专门设计来处理和分析大脑的血氧水平依赖(BOLD)信号,这些信号反映了大脑的活动变化。
**SPM8的安装与运行**
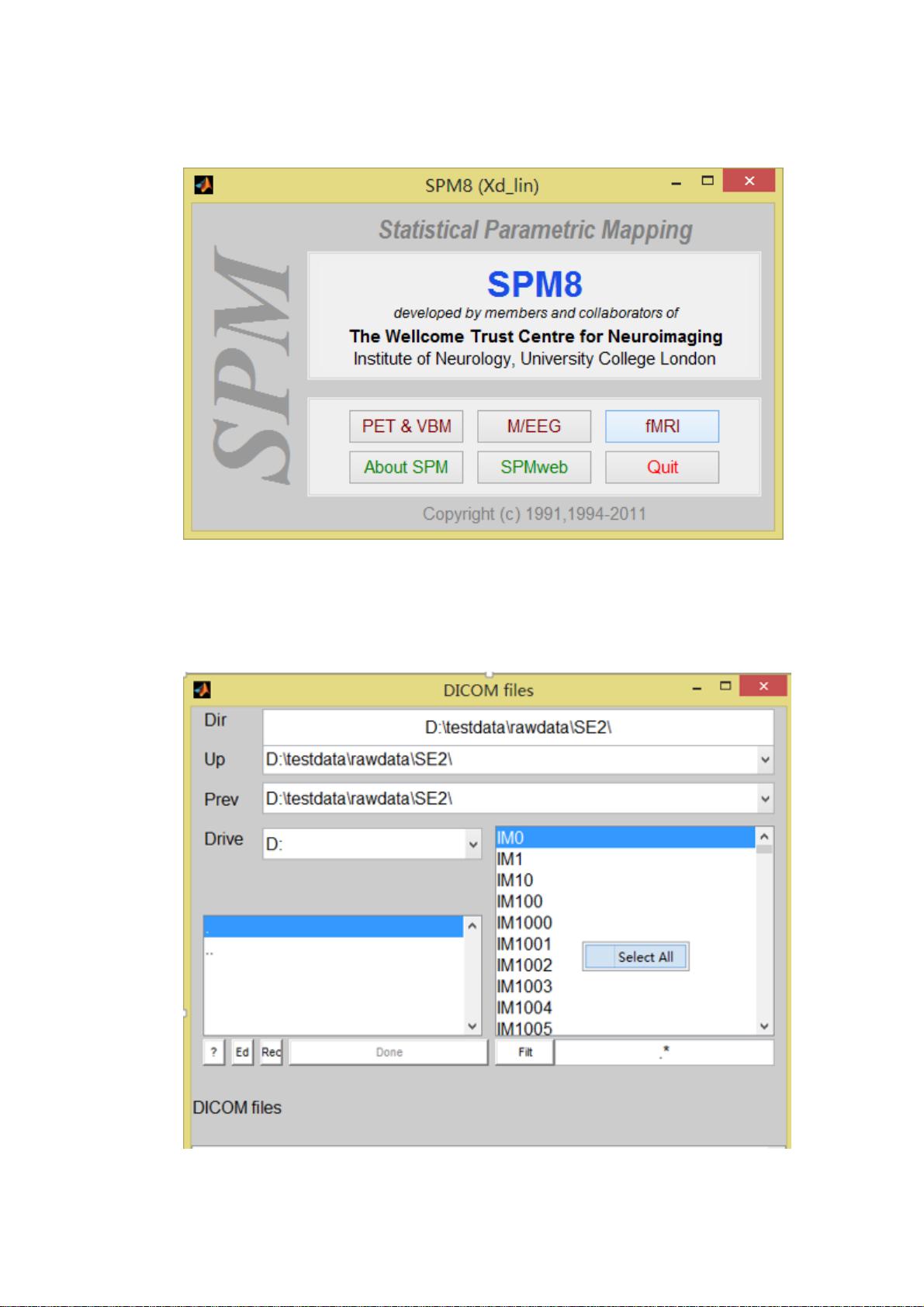
1. **安装SPM8**:首先,打开Matlab,通过File菜单选择SetPath,然后添加SPM8的文件夹到搜索路径。保存设置后,即可在Command Window中输入`spm`启动SPM8。
2. **卸载SPM5**:如果之前安装了SPM5,需要先卸载,方法同上,只需找到spm5文件夹并选择Remove,然后Save。
**数据准备**
fMRI数据分析的第一步通常是将原始数据转化为SPM可读的格式。以下是一个简单的流程:
1. **数据拷贝**:将原始的DICOM格式数据复制到非系统盘的英文命名文件夹中,以避免因中文路径导致的错误。
2. **启动SPM8**:在Matlab中启动SPM8,选择fMRI选项。
3. **数据导入**:使用DICOM Import功能,全选原始数据,点击Done。
4. **格式转换**:指定输出文件夹(英文路径),选择“.\”选项并点击Done,将数据转换为.img和.hdr格式,这两个文件分别存储图像数据和矩阵数据。
**多Session数据处理**
对于包含多个Session的数据,每个Session都需要单独进行上述转换过程。SPM8也提供了Batch选项,支持批量处理,这在处理大量数据时非常有用。
**fMRI预处理流程**
fMRI预处理通常包括以下几个步骤:
1. **头动校正**:消除由于受试者头部移动产生的伪影。
2. **空间标准化**:将个体数据转换到标准空间(如MNI模板)以便比较。
3. **高斯滤波**:降低噪声并平滑数据。
4. **时间序列分析**:进行模型建立,包括回归分析等。
5. **统计分析**:计算t值或F值图,进行阈值化处理,生成激活图。
在SPM8中,这些预处理步骤可以通过设定工作流并依次执行。每个步骤都有相应的参数可以调整,以适应不同的研究需求。
SPM8提供了一个强大而灵活的平台,用于处理和分析fMRI数据,从数据导入、预处理到统计分析,涵盖了整个fMRI研究的核心环节。正确理解和掌握SPM8的使用,对于理解大脑功能和进行神经科学研究至关重要。
2019-08-13 上传
2018-10-20 上传
2019-08-13 上传
2024-10-27 上传
2024-10-27 上传
2024-11-04 上传
2024-10-27 上传
2024-11-04 上传
2024-11-10 上传
LaVine01
- 粉丝: 1
- 资源: 7
最新资源
- XML文档对象模型(XML DOM)研究与应用
- DWR中文教程适合初学开发人员的最佳文档
- 新版设计模式手册[C#].pdf
- Professional JavaScript For Web Developers 2nd edition
- ibatis开发指南(含基础、高级部分)
- Beginning ASP.NET E Commerce In C Sharp From Novice To Professional
- Learning the vi and Vim Editors 7th Edition Jul 2008
- 网络工程的验收与鉴定.doc
- CSS.Mastery.Advanced.Web.Standards.Solutions.pdf
- AD与DA转换的pdf详细文档
- extjs详细教程-中文版
- 電腦做什麼事 0 序章 關於電腦
- 英语学习英语的资料,不是图片,视频
- Web_Service开发指南
- c#的习题,绝对实用,不下后悔
- MCTS70-640SelfPacedTrainingKit.pdf