DNA_ETL与元基索引ETL:编码原理与Tinshell应用
需积分: 0 159 浏览量
更新于2024-08-04
收藏 343KB DOCX 举报
第十一章深入探讨了DNA_ETL(Data Normalization and Extraction Language)与元基索引ETL(Enhanced Tableau Extract, Load, Transform)在中国软件开发中的应用。DNA_ETL的设计灵感源自德塔数据库的语言编译机,其编码方式允许开发者自由设计,包括中文描述,这增加了灵活性(pages 413, 834-835)。编码行能够整合到ETL流程中的节点中,实现单行执行,提升效率(pages 782, 784)。
PLETL语言作为DNA_ETL的扩展,不仅继承了德塔数据库的编译机特性,还引入了TCP和正则表达式等网络协议功能,以及多语种命令支持,使其具备更广泛的适用性(pages 377, 786-790)。Tinshell是PLETL语言下的核心组件,它基于德塔数据库编译机进行了优化,主要用于处理脚本的编译和执行,专注于输入和输出计算(pages 786-835, 782)。
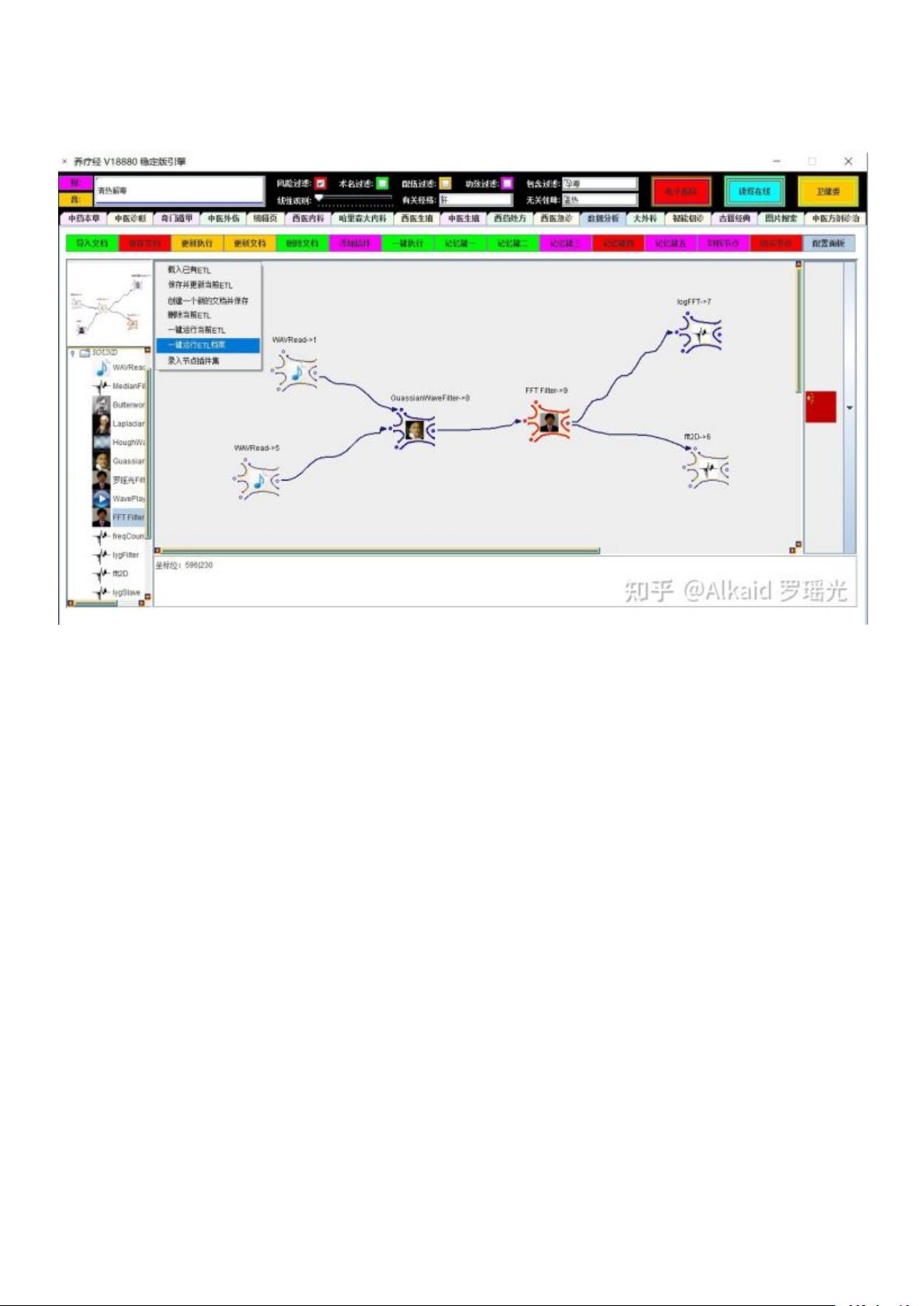
养疗经DNA元基版本的Tinshell在集成测试中表现出色,特别是在Alkaid罗瑶光的视频中展示的应用场景。这个章节也回顾了编译机的发展历程,德塔编译机起源于德塔socket流可编程数据库系统中的PL/SQL编译器(page 377)。随着技术的演进,德塔编译机独立出来,专门用于脚本编码,进一步拓展了与ETL和TCP的集成,最终在肽化索引(可能指代某种数据结构优化)后,成为模拟神经元ETL节点网络计算的核心(pages 783, 784)。
OSGI插件的设计初衷是为了方便节点导入,类似KNIME工具,但最初并未实现深层次集成,仅停留在继承层面。作者在2017年的经历促使他深入研究了类加载器技术(Class<?> myclass = loader.loadClass),这在后续的开发中可能扮演了重要角色(page 291)。
这一章节详细讲解了DNA_ETL和相关技术在IT领域的创新应用,强调了它们如何通过不断优化和扩展,提升数据处理的效率和灵活性,同时也揭示了开发者在面临技术挑战时的思考和解决方案。
2022-05-08 上传
2022-07-25 上传
2021-01-21 上传
2022-08-03 上传
销号le
- 粉丝: 32
- 资源: 289
最新资源
- SSM动力电池数据管理系统源码及数据库详解
- R语言桑基图绘制与SCI图输入文件代码分析
- Linux下Sakagari Hurricane翻译工作:cpktools的使用教程
- prettybench: 让 Go 基准测试结果更易读
- Python官方文档查询库,提升开发效率与时间节约
- 基于Django的Python就业系统毕设源码
- 高并发下的SpringBoot与Nginx+Redis会话共享解决方案
- 构建问答游戏:Node.js与Express.js实战教程
- MATLAB在旅行商问题中的应用与优化方法研究
- OMAPL138 DSP平台UPP接口编程实践
- 杰克逊维尔非营利地基工程的VMS项目介绍
- 宠物猫企业网站模板PHP源码下载
- 52简易计算器源码解析与下载指南
- 探索Node.js v6.2.1 - 事件驱动的高性能Web服务器环境
- 找回WinSCP密码的神器:winscppasswd工具介绍
- xctools:解析Xcode命令行工具输出的Ruby库