多重检验与微阵列数据分析:FDR控制与原假设估计的改进方法
版权申诉
72 浏览量
更新于2024-07-02
收藏 798KB PDF 举报
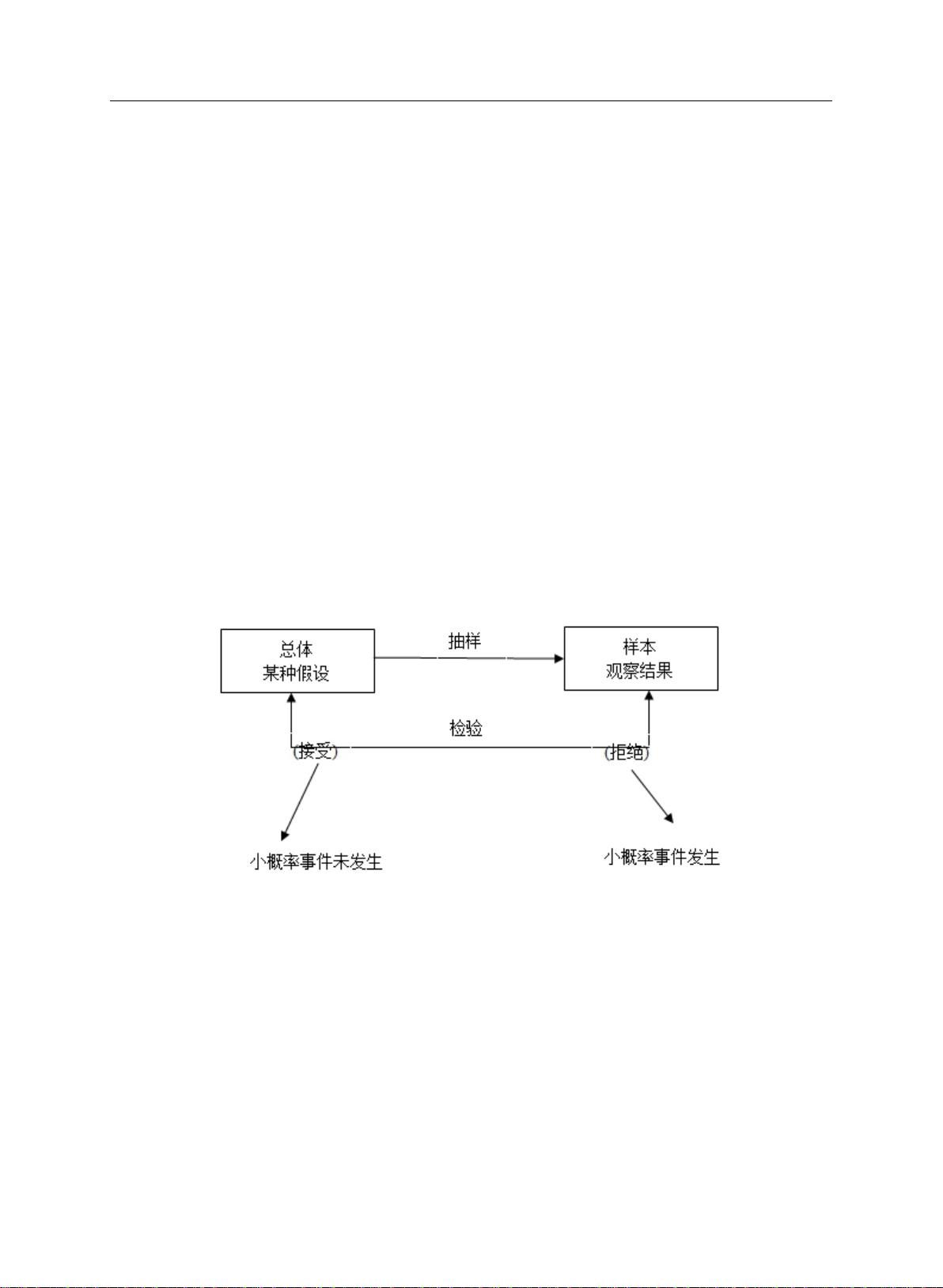
多重检验技术是生物信息学和基因组学等领域中不可或缺的数据分析工具,特别是在微阵列数据分析中发挥着关键作用。该论文深入探讨了多重检验中两个核心问题:错误发现率(False Discovery Rate, FDR)的控制和正确原假设比例(true null hypothesis proportion, m0)的估计。多重检验的目标是在众多独立测试中,既要控制因过度解读而导致的第一类错误(Type I error),即误判非差异基因为差异,又要提高显著性水平,从而找出真正有意义的差异基因。
论文首先介绍了多重检验的基本理论,特别强调了控制第一类错误的重要性,通常通过调整Family Wise Error Rate (FWER)和FDR来实现。FWER虽然传统上被广泛采用,但过于保守;而Benjamini-Hochberg (1995)提出的FDR控制方法提供了一种更为灵活且效果更好的策略。文章重点研究了四种能够有效控制FDR的算法,包括Bonferroni方法,通过对模拟数据的对比分析,展示了q值方法在控制FDR的同时保持较高检验功效的优势。
其次,论文针对估计原假设比例m0的问题进行了深入研究。通过模拟实验,文中对几种常见的估计方法进行了比较,如Jiang&Doerge(2008)的均值法,以及作者对其进行了改进,使用三次样条法替代bootstrap法,以提高估计精度。实验结果显示,改进后的样条平滑估计方法在估计m0时比李伟(2014)的方法更为精确。
论文还通过实际案例,如Hendenfalk(2001)的乳腺癌数据和FengPan等人(2009)的B细胞数据,验证了改进的估计方法在微阵列数据分析中的性能。相比于Hochberg&Benjamini(2000)、Storey&Tibshirani(2002)和Langaas等人(2005)提出的降密度算法,作者的方法能够发现更多或更少数量的差异基因,同时保持了与李伟算法相当的功效,这证明了改进的均值估计法的有效性。
总结来说,这篇论文深入探讨了多重检验技术在微阵列数据分析中的应用,重点在于如何有效控制FDR和准确估计m0,从而提高基因差异筛选的精确性和效率。通过实际案例和模拟实验,作者提出了改进的估计方法,对于生物信息学研究者而言,这些研究成果为微阵列数据分析提供了实用的工具和技术指导。
2023-12-23 上传
2022-07-02 上传
2010-11-13 上传
2021-07-26 上传
2021-07-26 上传
2022-07-02 上传
2021-07-26 上传
programyp
- 粉丝: 89
- 资源: 9323
最新资源
- Android圆角进度条控件的设计与应用
- mui框架实现带侧边栏的响应式布局
- Android仿知乎横线直线进度条实现教程
- SSM选课系统实现:Spring+SpringMVC+MyBatis源码剖析
- 使用JavaScript开发的流星待办事项应用
- Google Code Jam 2015竞赛回顾与Java编程实践
- Angular 2与NW.js集成:通过Webpack和Gulp构建环境详解
- OneDayTripPlanner:数字化城市旅游活动规划助手
- TinySTM 轻量级原子操作库的详细介绍与安装指南
- 模拟PHP序列化:JavaScript实现序列化与反序列化技术
- ***进销存系统全面功能介绍与开发指南
- 掌握Clojure命名空间的正确重新加载技巧
- 免费获取VMD模态分解Matlab源代码与案例数据
- BuglyEasyToUnity最新更新优化:简化Unity开发者接入流程
- Android学生俱乐部项目任务2解析与实践
- 掌握Elixir语言构建高效分布式网络爬虫