理解BLAST算法:数据库中的序列搜索与比对
版权申诉
24 浏览量
更新于2024-07-03
收藏 4.16MB PPT 举报
"该资源为一个关于数据库搜索的PPT,主要探讨了数据库搜索在生物信息学中的应用,特别是BLAST算法的原理和用途。"
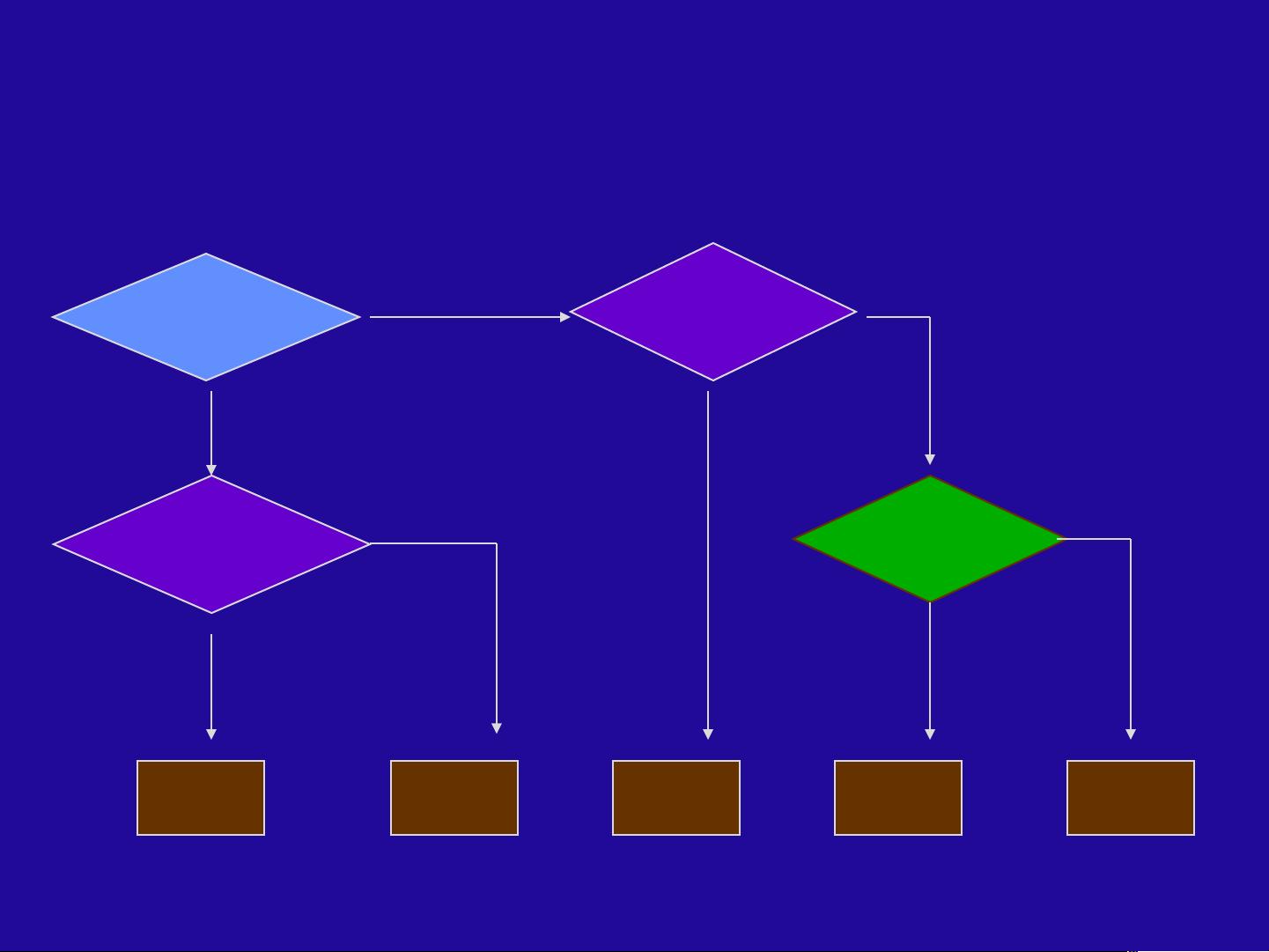
数据库搜索是生物信息学领域中一个至关重要的技术,它主要用于查找和分析生物序列数据。在描述中提到的关键字查询和目标搜索是数据库搜索的基本方式,通过输入特定的关键词或目标序列,可以在庞大的生物序列数据库中找到相关的匹配项。
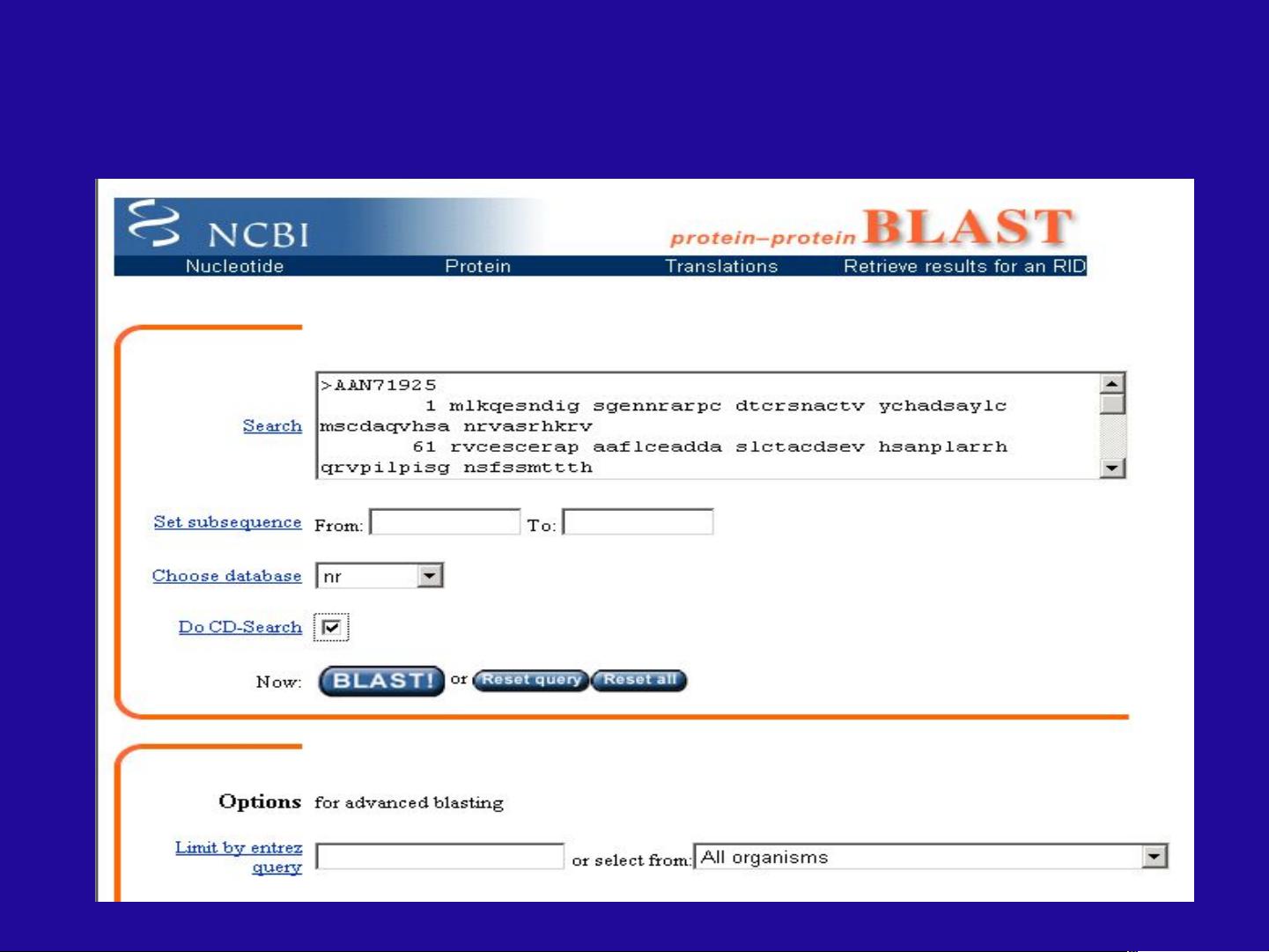
BLAST,全称为Basic Local Alignment Search Tool,是一种快速的序列比对工具,由Altschul等人在1990年和1997年提出。它主要用来判断序列是否为新序列,预测序列的功能,进行同源性鉴定,以及在基因电子克隆中发挥作用。BLAST算法的核心是序列片段对的概念,即两个序列中长度相等且能完全匹配的子序列。无空位的BLAST比对寻找超过特定阈值的匹配片段,然后延伸这些片段对,形成高分值片段对(HSPs)。
无空位的BLAST算法在最初阶段只能处理没有空位(即插入或缺失)的比对。然而,为了捕捉到更具有生物学意义的比对,BLAST被改进为允许空位的比对。这一改进的算法首先找到最佳的HSP,然后使用动态规划方法在其两端延伸,允许插入空位,从而提高了搜索效率和结果的生物学相关性。
BLAST的运行机制包括将查询序列分割成多个小片段,筛选数据库中包含这些片段的序列,然后延伸匹配序列并计算分数,通常会根据期望值(E值)和比特分数(Hit Score)来评估比对的显著性。期望值E值表示在随机情况下获得当前或更好匹配结果的概率,比特分数则是一种标准化的分数,用于不同长度和复杂度的序列之间的比较。
BLAST算法是生物信息学中的一个关键工具,它的高效性和准确性使得科学家们能够在大量序列数据中快速找到相关性,从而推动了基因组学和蛋白质组学的研究进展。通过对BLAST的理解和应用,研究人员可以更好地理解生物序列间的相似性和差异性,从而揭示潜在的生物学功能和进化关系。
点击了解资源详情
点击了解资源详情
点击了解资源详情