2011年Gromacs分子模拟讲座:原理与应用详解
需积分: 9 27 浏览量
更新于2024-07-15
收藏 2.38MB PDF 举报
Gromacs-lec-2011-11-session1.pdf是一份关于Gromacs程序的详细介绍文档,由清华大学机械系的李启楷教授在2011年的高性能计算培训中分享。Gromacs是一款专用于分子生物学和生物化学领域的分子动力学模拟工具,其核心优势包括:
1. **基础原理与应用**:该讲座首先概述了Gromacs的基本原理,它是如何将物理、化学和生物学理论链接起来,通过模拟难以在实验中观察的现象,如蛋白质折叠和纳米尺度过程,来理解这些复杂系统的行为。
2. **软件特性**:
- **编程语言与并行处理**:Gromacs主要用C语言编写,同时结合MPI库和线程进行并行化处理,使其能够高效地运行在分布式内存集群和共享内存计算机上。
- **内置功能**:提供多种内置的力场和工具,支持各种类型的分子模拟研究。
- **免费下载**:最新版本(4.5.5)是开源的,可以从gromacs.org网站免费获取。
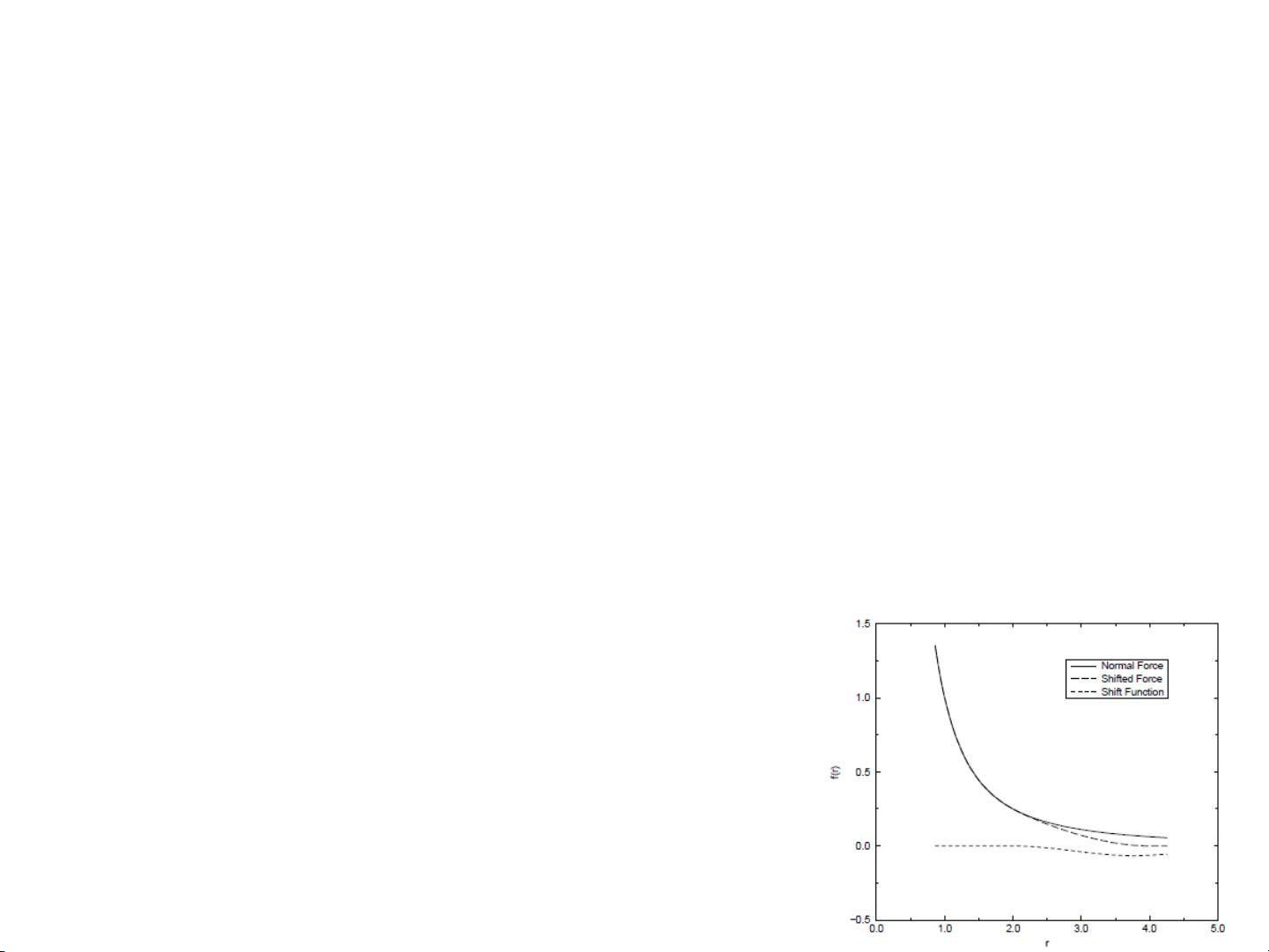
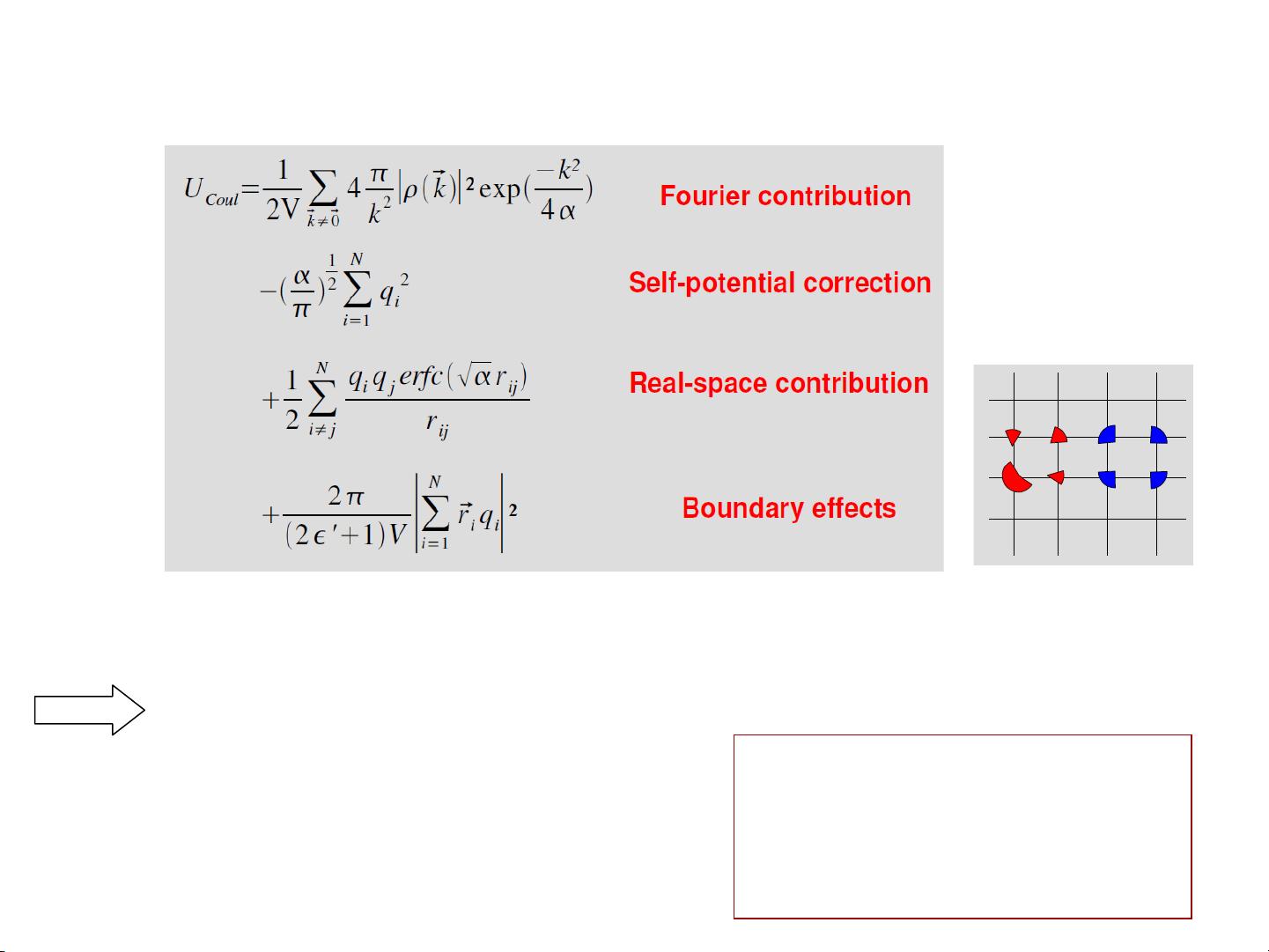
3. **模拟目的**:MD(分子动力学)模拟之所以重要,是因为它基于牛顿运动定律,通过微小时间步长的积分来计算粒子的轨迹,模拟系统随时间的动态变化。这使得研究人员能够计算关键的热力学量,如自由能和结合能,从而深入了解分子间相互作用和系统行为。
4. **模拟方法**:利用牛顿方程,特别是动量定理,通过计算每个粒子受到的力(F),根据牛顿第二定律(F = ma),确定加速度,然后通过微分来估计位置变化。这个过程被重复执行,生成系统的动力学轨迹,从而揭示分子层面的动态行为。
文件内容涵盖了Gromacs在分子模拟中的基本概念、技术细节以及其在科学研究中的实际应用,对想要学习或使用Gromacs进行科研的人员来说,这是一个宝贵的资源。通过这份讲座,参与者可以掌握如何设置和运行Gromacs模拟,以及如何解读和分析模拟结果,从而推动生物学和化学领域的理论进步。
2020-07-14 上传
2021-04-22 上传
2021-05-03 上传
2024-03-28 上传
2024-03-28 上传
2020-07-14 上传
2024-03-28 上传
2020-12-28 上传
2021-04-26 上传
D2472260125
- 粉丝: 1
- 资源: 35
最新资源
- MATLAB新功能:Multi-frame ViewRGB制作彩色图阴影
- XKCD Substitutions 3-crx插件:创新的网页文字替换工具
- Python实现8位等离子效果开源项目plasma.py解读
- 维护商店移动应用:基于PhoneGap的移动API应用
- Laravel-Admin的Redis Manager扩展使用教程
- Jekyll代理主题使用指南及文件结构解析
- cPanel中PHP多版本插件的安装与配置指南
- 深入探讨React和Typescript在Alias kopio游戏中的应用
- node.js OSC服务器实现:Gibber消息转换技术解析
- 体验最新升级版的mdbootstrap pro 6.1.0组件库
- 超市盘点过机系统实现与delphi应用
- Boogle: 探索 Python 编程的 Boggle 仿制品
- C++实现的Physics2D简易2D物理模拟
- 傅里叶级数在分数阶微分积分计算中的应用与实现
- Windows Phone与PhoneGap应用隔离存储文件访问方法
- iso8601-interval-recurrence:掌握ISO8601日期范围与重复间隔检查