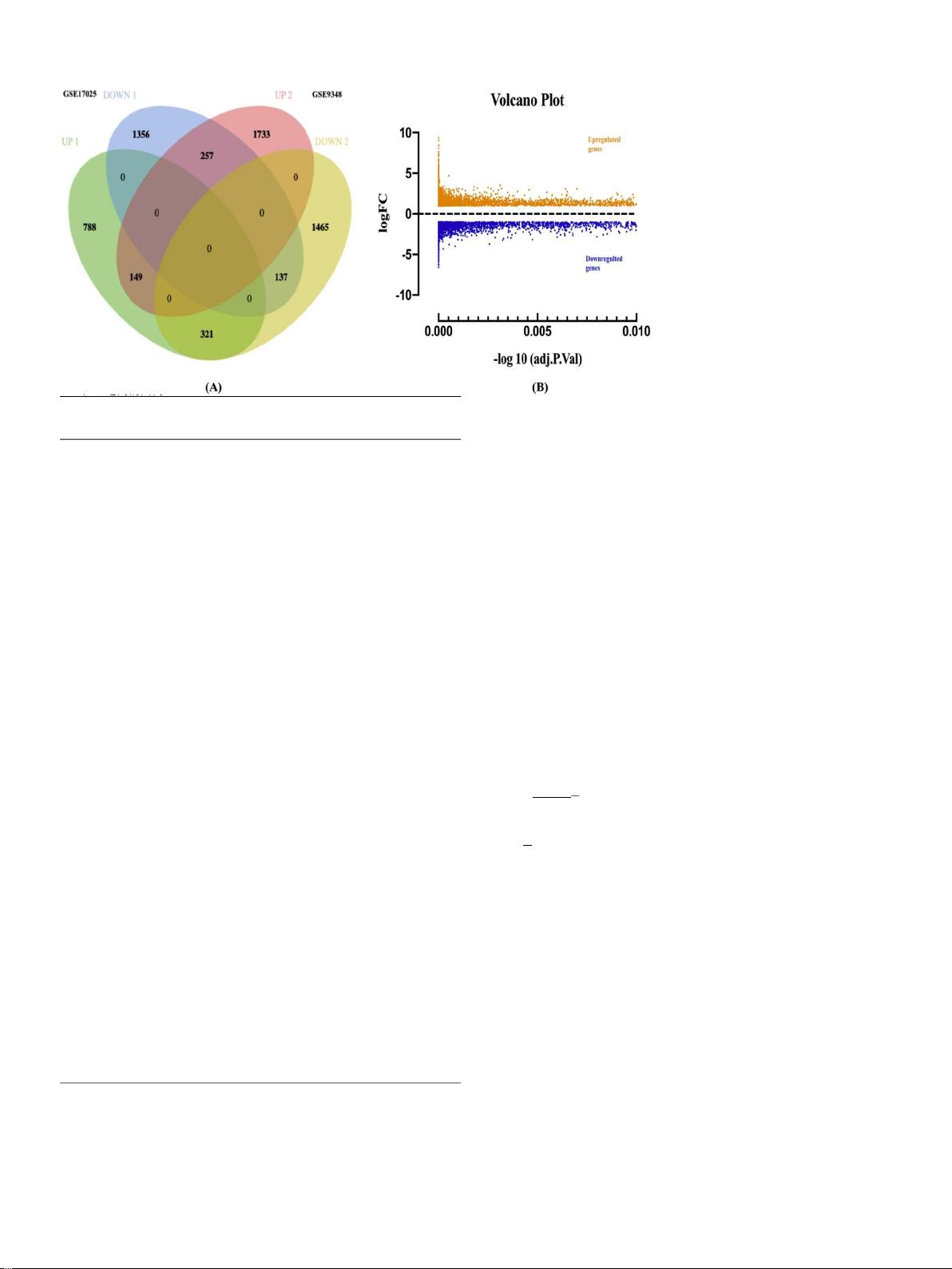
图三.
两个数据 集的差异 表达基因 。A. DEG
的维恩图。分别对来自EC和CRC数据集的上
调和下调GSE 17025数据集包含3008个DEG
(1258个 上调和 1750个下调 ), GSE 9348
数 据 集 包 含 4062 个 DEG ( 2139 个 上 调 和
1923个下调)。
B.
总DEG的火山图(来自两
个数据集)。棕色的点代表上调基因,蓝色
的点代表下调基因。(For对本图中颜色图例
的解释,读者可参考本文的网络版。)
(ii)它们具有相同的平台,(iii)它们具有至少20个样本,分为肿瘤
和正常样本,以及(iv)关于这些数据集的研究以英语公开可用。两
个数据集(表1),
EC的GSE 17025 [22]和CRC的GSE 9348 [23]符合所有标准,从基因表
达综合库(GEO)中检索[24],
149 CAPN13 HK2 IGK HAPLN1 EPHA4 FOXA 1 CTLA 4
OAS 1 MALL MICAL2 CXCL13 PAX6 MARCKS CHST 6
EZR FYB GDE 1 DNAJB 4 CYAT 1 MAL SPINK5 CA2
ETS1 SSFA2 TMEM37 MICALL2 CCDC168 ADAMDEC
1 SLAMF7 TFCP 2L1 SGPP2 DST HIST1H2BC
MS4A4A CPT1A BCL2L11 GSTA 1 PTPRF TMPRSS2
KCNJ2 GJB 2 HIST 1H3F VDR NEDD4 L CAMMMT
MARVELD 3 CTSS CLDN7 CYBB HIST1H4H PTPN 22
APLP2 STAM2 ME2 PRKCB TCP11L1 SCYL2 NHSL1
PLOD2 PAPSS2 PCCB CD24 CD164 RNASE1 AP1AR
RHBDL2 MESP1 MESP2 CCNYL1 GK TJP3 SLC27A2
MPDU 1 FLII RIOK3 FOXN4 MGAT4A LACTB AK9
CALML4 LGALS 8 CKMT 1A TLCD2 RNF187 OASL
RAB 27A RAP1A SLC41A2 HIST1H3G QSOX 1 PSEN1
IL18 NEURL1B RIMS 3 EGLN 1 GOT1 CFIGLAR LC1
PRR5L CCNG2 UNC5B TC2N CORO2A CLN8 CD28
SLC17A5 BPNT1 RBM47 PPP2R3A SYNPO CCDC88C
CDCP1 PDE8A SNX13 SLC36A1 SLC22A23 SIPA1L2
CALM3 STS IVNS1ABP FAM126B ATP6V0D1 PPID
GMFB SOCS6 HMOX1 CKAP2 KLF3 GSR FCRL 3
CCNJL ABHD17C FOSL2 AK2 FAM162A CDC42 GNAQ
ATL 3 ATP6V1D SCP 2 GAREM 1 CTNND 1 SLC2A5
UPP1 WDR 1 FLNB GHITM
公开可用的功能基因组学数据储存库,其支持符合MIAME的数据提
交,NCBI(https://www.ncbi.nlm. nih.gov/geo/)。
2.3.
数据集的综合分析
CRC数据集包含年龄和种族匹配的患者(n 70)和健康对照(n 12)
的全基因组表达谱 使用AffymetriX U133 Plus 2阵列。所有患者均为早
期结直肠癌。EC数据集包含使用AffymetriX平台的I期EC患者(n 91)和
对照(n 12)样品的基因表达谱。在获得数据集后,我们进行了Z分数归
一化方法,这是一种广泛使用的技术,用于解决使用不同实验设置进行
微阵列数据比较的问题,以转换疾病和对照状态的每个基因表达数据。
为了标准化基因表达矩阵,我们使用平均值和标准差。
假设m是每个基因的值,n是样本。 因此
基因表达值可以用Xmn表示,并且可以是反式的。
ADAM10 BSG
RPS21 AKT2 TGIF1 SMYD5 IRAK1BP1 ACD NAP1L1
UBE2I RFXAP RBPMS LYRM4 FAM92A1 EXOSC 8
SPIN3 LOC730268 MAGED 2 LAMP2 ZAK ZNF 599
JUN ARGLU 1 WDR91 LPCAT 2 LRRC14 SNX21 CPLX
1 LSM 5 PDCD 5 CREBZF CEP290 SPICE 1 TTC 17
PCGF 3
TPR MXRA7 UCKL1 ZMYM5 NR4A2 ZCCHC7
CAMSAP2 OLA1 AGFG 1 SLC2A8 IRS2 NSUN5P2
C12orf29 EIF3H NENF CLEC11A SNORD77 BICD 1
KIZ CDCA 7 KIAA1217 PAGR 1 CEP83 COL5A1 MYC
YTHDC1 GPALPP 1 C12orf66 AMOTL 2 HABP4
CACNA 1D EGR 2 OBSCN ZNF783 EPDR 1 WISP1
CTSK FSTL 3 COL27A1 GFBP 7 LINC00663 CDK 6
EDNRA PABPC1L TSEN54 TMEM 120B GNB5
DCUN1D5 EGR1 ZEB1 CCDC57 SHROOM4 JUNB
SFXN3 BOK OGFOD1 LOC101927668 WNT2 PRRX 1
LEF1 LOC101 927811 TMEM170B LOC 100272217
ACKR3 FBN1 IRS1 RUSC2 PES 1 CAMTA 1 NUB1
ITGBL 1 ETS2 CYR61 GRK3 NR4B1@1 C9orf72 NAT6
TIMP3 ZNF703 KAT2A IL20RA EGR3 SCML1 NEBL
FOXA2 STMN3 LSAMP WT1 SLC22A3 CDH11 SIK1
LIMS4 SCARNA 2 DACH 1 AZGP 1 ETV5 CTGF KCNH8
MSX2 DUSP2 TRO SFRP 4 FOS FOSB
这里,
A
和
σ
A
分别表示平均值和标准差该方程用于比较样品的基因表
达值在标准化之后,通过设定以下截止值来过滤基因:
校正p值<0.01,|log
2
FC| 1、统计学意义上的意义--
倾斜DEG。我们还用Limma软件包对获得的DEG进行了
[25]在R/Bioconductor软件(http://www.bioconductor.org)中进行
确认。
2.4.
DEG
的富集分析
我们使用用于注释、可视化和集成发现的数据库(DAVID v6.8,
www.example.com)对CRC和EC之间的相互DEG进行功能和途径富集
分析http://david.abcc。 ncifcrf.gov/)[26]。通过执行这些基因的过
度表达分析,功能概述DEG及其相关途径进行了阐述。我们在DAVID
中进行了基因本体(GO)分析,