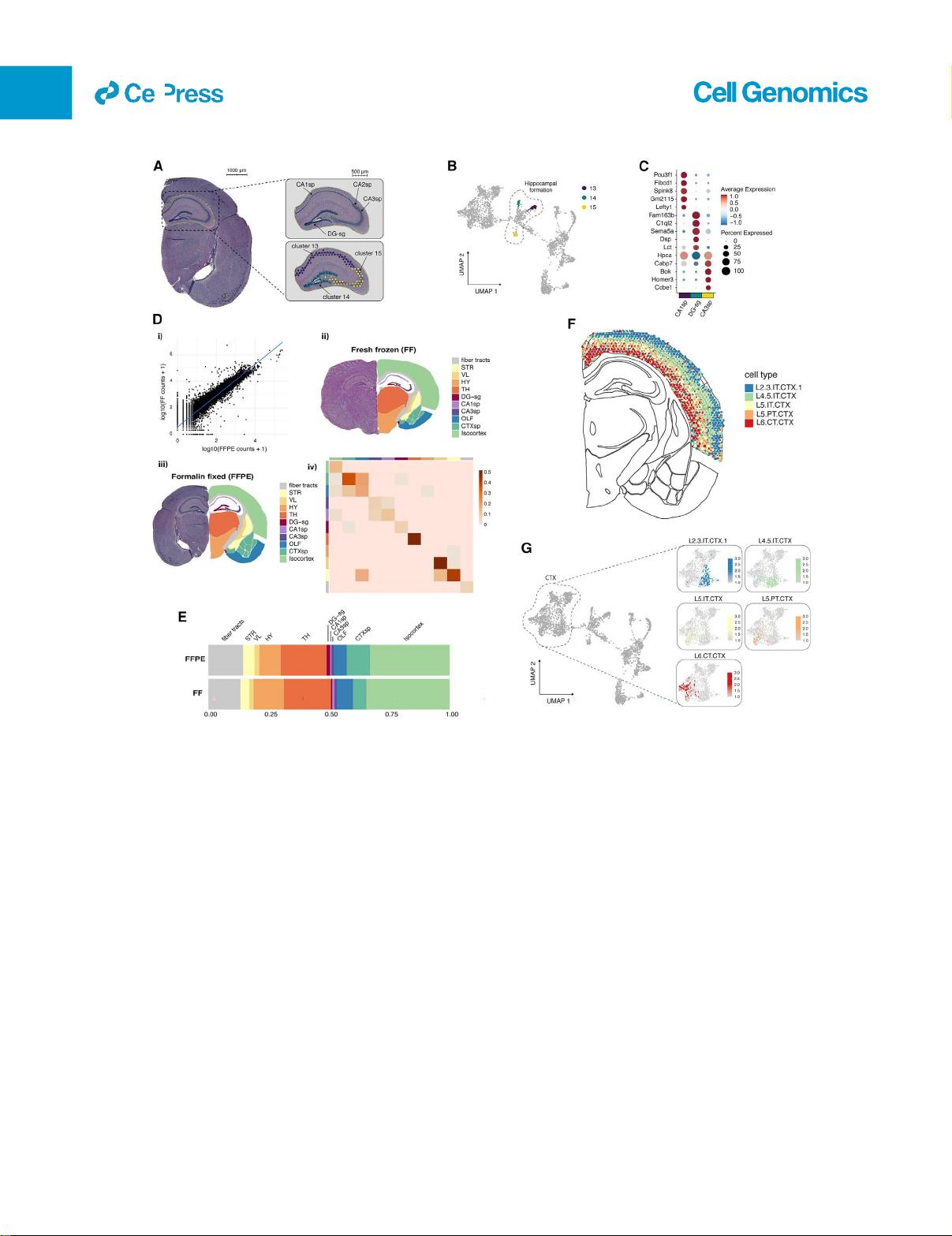
允许在被空间分析的组织中无偏的mRNA聚(A)捕获。基于测
序的分析的开创性方法之一是空间转录组学,
4
当应用于新鲜冷
冻(FF)组织切片以探索和分析器官发育和疾病中的转录组学
景观时,该方法已反复证明其价值。
5
FFPE样品
9
、
10
的基于灵敏测序的空间转录组学方法的开发受到
福尔马林诱导的mRNA分子交联和降解所引起的技术挑战的阻
碍。已经开发了全基因组定量策略以应用于大量FFPE样品,
11
,
12
其通常涉及核糖体消耗或使用寡核苷酸探针杂交的靶向捕获以
富集片段化mRNA;然而,目前没有可用于FFPE组织样品的无在
这项研究中,我们提出了一种基于商业平台的方案,适用于从
FFPE组织切片中恢复空间分辨的mRNA谱。我们应用该方案来
表征来自小鼠脑、人类类器官、临床癌症样品和来自感染严重急
性呼吸综合征(SARS)-CoV-2的患者的肺组织的FFPE组织我
们的结果表明,所提出的协议是通用的,可以恢复全基因组信息
从不同的组织类型保存在不同的条件下。FFPE一直是生物库中
临床样本储存的金标准,我们预计我们的发现将促进对存档生物
标本的研究,以更好地表征疾病进展、免疫反应和器官发育中关
键事件的分子基础。
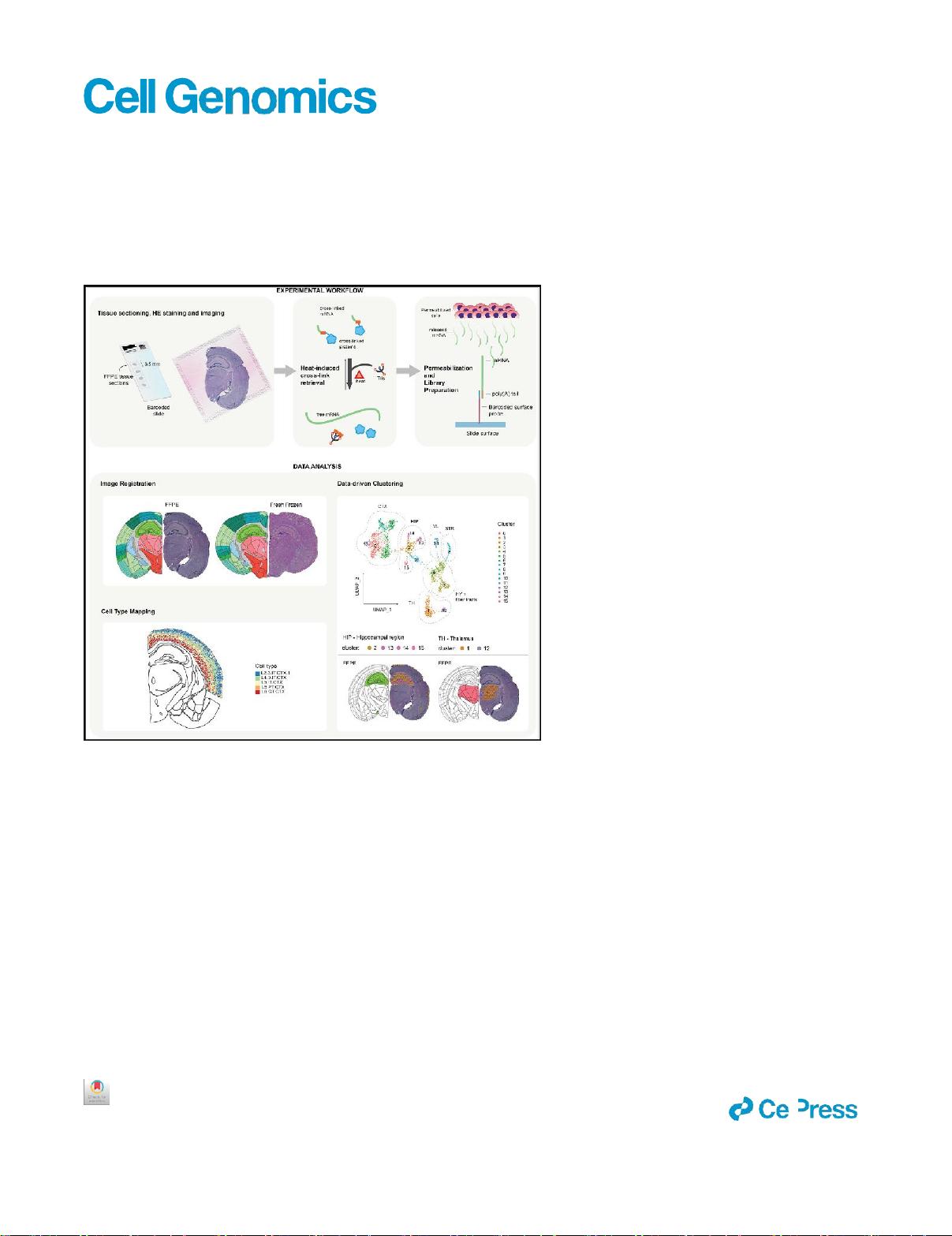
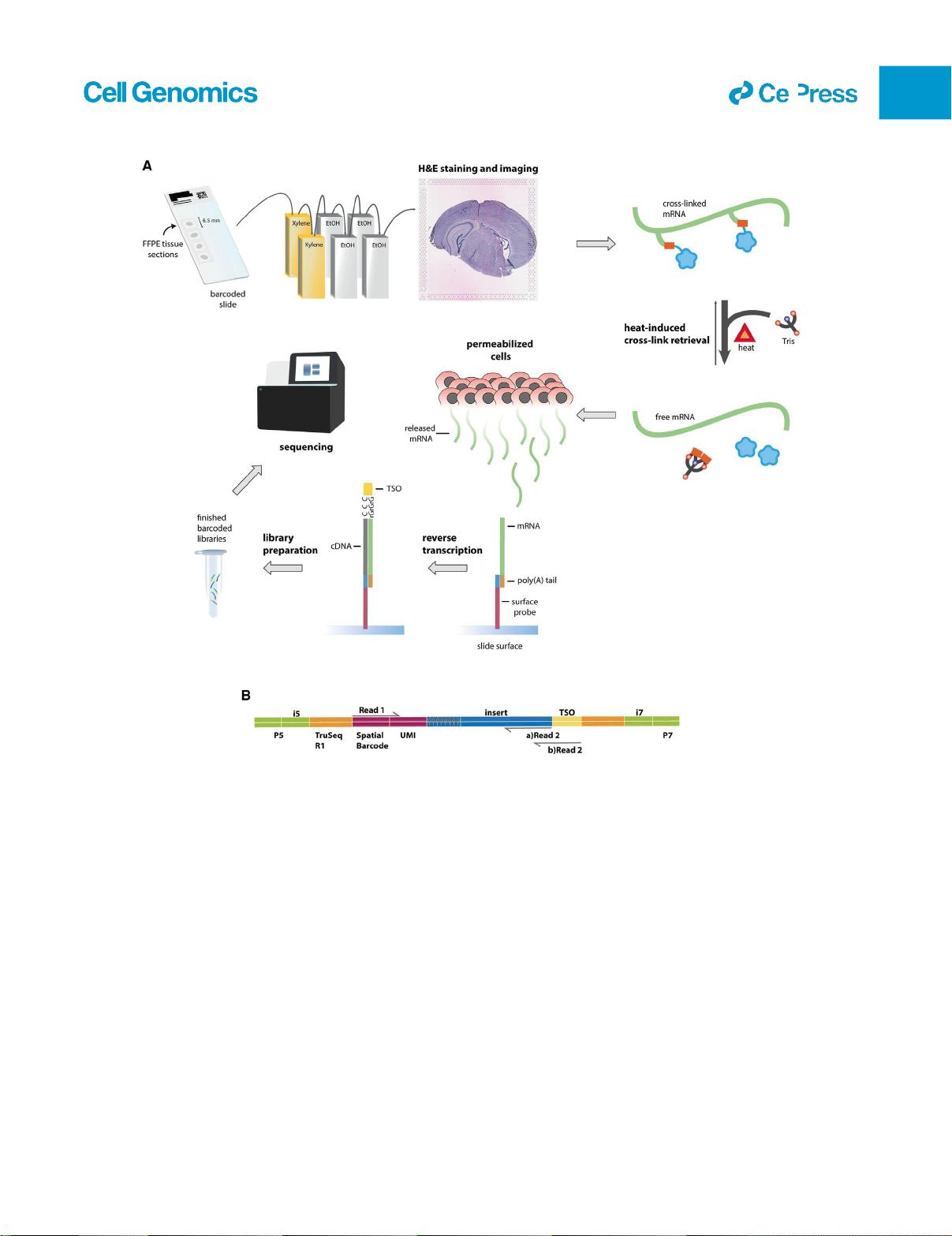
设计
通过
原位
去除石蜡和交联,将组织切片置于条形码载玻片上,实
现用于空间FFPE分析的固定mRNA的回收(图1)。用于FFPE
样品的方案在几个方面与已建立的FF工作流程
13
不同,以允许交
联逆转以及尺寸选择和片段化过程的变化。引入某些步骤以适应
FFPE样品的特殊性,而其他变化与数据产量优化有关(参见
STAR方法)。
首先,将漂浮的FFPE组织切片附着到空间条形码化的载玻片
上,在烘箱中干燥,然后通过在二甲苯和乙醇中连续浸渍脱蜡考
虑到FFPE组织已经用交联剂固定了较长时间(约24 h),我们
推断形态将得到保留,因此省略了固定步骤。组织切片用苏木精
和伊红(HE)染色并在高分辨率显微镜下成像。接下来,将组
织切片用胶原酶预透化,然后进行交联逆转。胶原酶被包括在早
期的空间转录组学方案中,以促进组织-
细胞去除和mRNA捕获,因为已知其有助于细胞外基质结构的破
坏。
14
通过在70 ℃下用pH 8.0的Tris-EDTA缓冲液热诱导修复
15
的pH
8.0先前已经报道了防止不希望的副反应,例如pH依赖性RNA水
解。
[16]
此外,有人提出了三羟甲基氨基甲烷和甲醛分子之间的淬
灭机制。
2
通过研究Tris-EDTA(TE)缓冲液组成和pH的变化,
使用储存在6
℃
的结肠癌FFPE组织优化去交联(图S1)。我们发
现,没有一种组合物在荧光标记的cDNA信号中呈现任何剧烈变
化,并得出结论,所有组合物都产生可接受的信号。通过额外的
组织优化(TO)实验评价了我们认为具有稍好的cDNA信号的两
种条件(图S2A)。接下来,为了获得定量数据,使用这些条件
制备cDNA文库。再次,测序数据没有显示出任何重大差异,其
中似乎最佳的组成是TE缓冲液pH 8.0(表S1)。
交联逆转后,组织切片进行酶促透化。建议根据组织类型优化
透化时间,因为可能影响mRNA可及性的特定特征在组织间存在
差异。这通常通过TO测定来确定,如其他地方所述。在我们的
实验中,我们另外评估了PBS作为可能的解交联剂(图S2B)。
在小鼠脑组织中,在30 min的透化持续时间下确定最佳信号(图
S2B),PBS的性能不优于TE缓冲液pH 8.0。
接下来,如STAR方法中所述,用表S2中给出的实验条件和质
量度量预测序来自不同组织的测序文库。
基于
TSO
的质量控制(
QC
)测定
在这项工作中,我们开发了一种测定法,用于在文库制备之前评
估样品的非交联聚腺苷酸化RNA分子的空间可及性(图S3)。
在 我 们 的 方 法 设 计 过 程 的 开 始 , 我 们 依 赖 于 RNA 完 整 性 数
(RIN)和分布值200(DV200)计算总RNA提取,以衡量FFPE
块是否有足够的RNA质量进行分析。然而,这些指标对测序数据
质量的预测能力低于预期。鉴定了一些差异:一组临床癌肉瘤样
品具有相对低的RIN值(观察到的:2.3我们研究的第一个FFPE
小鼠脑样品,我们从中获得了高转录组覆盖率用于我们的分析,
具有65%的DV200值然而,具有高达68%的DV200的FFPE块
(其后来显示已固定极长时间(5天,通常为24小时))产生低
复杂性的文库(表S3)。这些质量测量基于核糖体RNA完整性
或总RNA,并且不考虑甲醛处理引起的mRNA的聚(A)尾的可
能损失。此外,这些都是散装质量