MSSU-Net:多尺度监督算法提升肾脏与肿瘤CT分割精度
PDF格式 | 1.58MB |
更新于2025-01-16
| 48 浏览量 | 举报
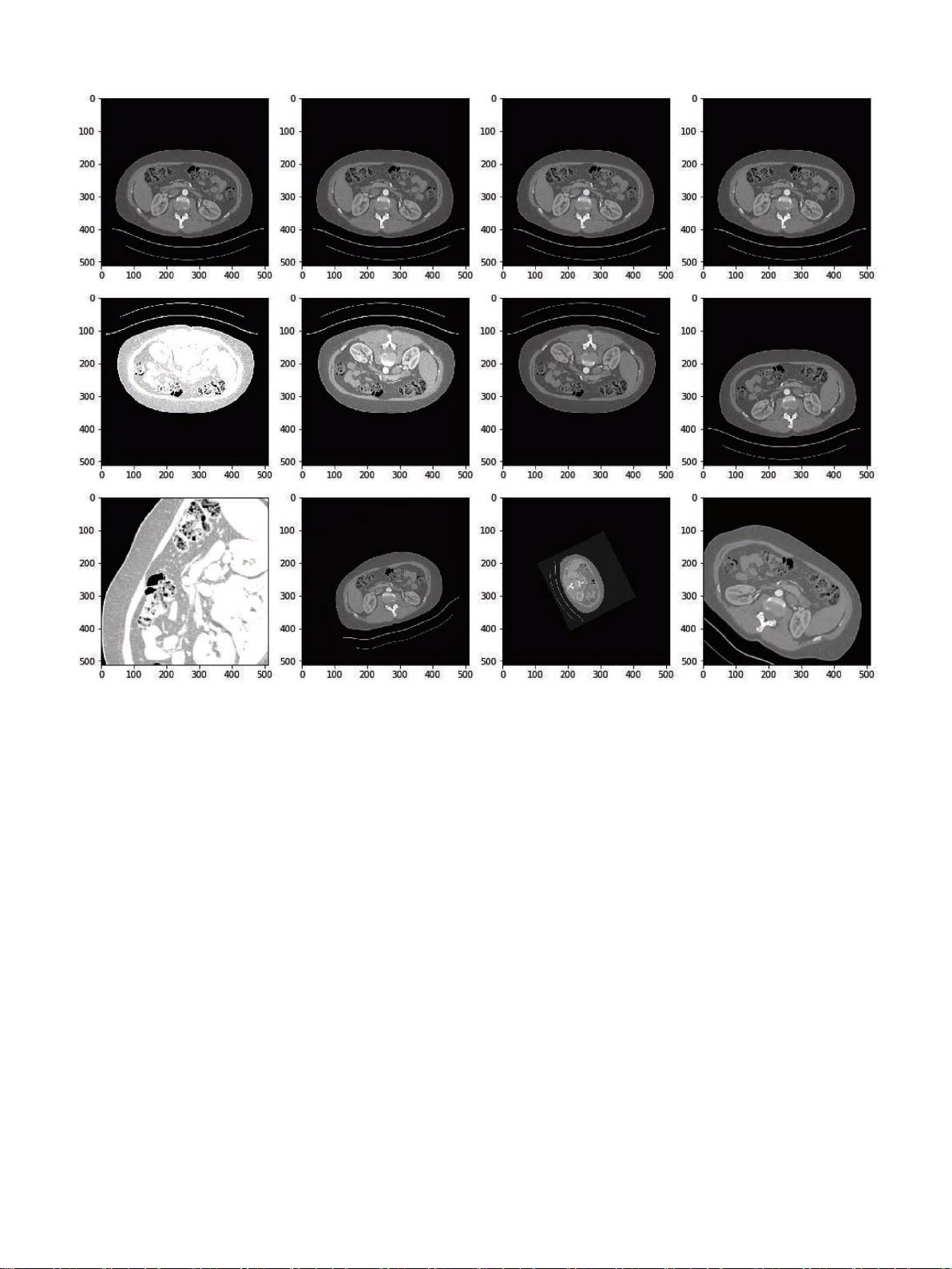
MSS U-Net是一项创新的深度学习算法,专为医学图像分析设计,特别是在肾脏和肿瘤的CT图像分割领域展现出卓越性能。该研究发表在《医学信息学解锁19》(2020)上,关注的是多尺度监督在3D U-Net架构中的应用,即MSSU-Net。传统的3D U-Net是一种基于深度学习的卷积神经网络,广泛用于医疗影像分析,其核心是U型结构,能够有效地捕捉上下文信息。
赵文帅等人提出的MSSU-Net旨在解决肾脏和肾脏肿瘤分割的精度问题,这是一个在放射组学分析中至关重要的任务,因为它能帮助医生制定精确的手术计划。传统的手动分割方法依赖于临床医生的视觉判断,耗时且主观性较大。MSSU-Net通过结合深度监督和指数对数损失函数,优化了3D U-Net的训练过程,提高了模型的稳定性和效率。
在设计上,MSSU-Net引入了多尺度特征提取,它能够捕获不同尺度上的细节信息,这对于肾脏和肿瘤这类大小、形状各异的目标具有重要意义。此外,文章还提出了一种连接组件为基础的后处理策略,进一步提升了分割结果的准确性和完整性。通过与现有的分割技术进行比较,MSSU-Net在肾脏和肿瘤的Dice系数上分别达到了0.969和0.805的高精度,这在KiTS19挑战赛的数据集上得到了验证。
值得注意的是,虽然早期的研究倾向于使用无监督方法,如阈值、区域生长、聚类等,但MSSU-Net凭借深度学习的优势,提供了端到端的解决方案,避免了传统方法中的一些限制。随着3D卷积神经网络在医学领域的广泛应用,MSSU-Net展示了深度学习在自动肾脏和肿瘤分割中的潜力,有望推动医学影像分析的进一步发展和临床实践的改进。
相关推荐






cpongm
- 粉丝: 6
最新资源
- Ruby语言集成Mandrill API的gem开发
- 开源嵌入式qt软键盘SYSZUXpinyin可移植源代码
- Kinect2.0实现高清面部特征精确对齐技术
- React与GitHub Jobs API整合的就业搜索应用
- MATLAB傅里叶变换函数应用实例分析
- 探索鼠标悬停特效的实现与应用
- 工行捷德U盾64位驱动程序安装指南
- Apache与Tomcat整合集群配置教程
- 成为JavaScript英雄:掌握be-the-hero-master技巧
- 深入实践Java编程珠玑:第13章源代码解析
- Proficy Maintenance Gateway软件:实时维护策略助力业务变革
- HTML5图片上传与编辑控件的实现
- RTDS环境下电网STATCOM模型的应用与分析
- 掌握Matlab下偏微分方程的有限元方法解析
- Aop原理与示例程序解读
- projete大语言项目登陆页面设计与实现