开放获取
文章
对韩国生物库数据中72,298个个体的
76个性状进行全基因组研究
Kisung Nam
,
1
Jangho Kim
,
1
和
Seunggeun Lee
1,2,
*
1
首尔国立大学数据科学研究生院,首尔08826,大韩
2
电极导线触点
* 通讯地址:https://doi.org/10.1016/j.xgen.2022.100189lee7801@snu.ac.kr
总结
缺乏对不同祖先群体的全基因组关联研究(
GWAS
),导致遗传学发现和多基因评分不足我们对韩国生物
库数据中的
76
种表型进行了
GWAS
,即韩国基因组和流行病学研究(
KoGES
)(
n = 72
,
298
)。我们的
分析发现了
2,242
个相关基因座,包括
122
个新的关联,其中许多在日本生物银行(
BBJ
)
GWAS
中得到了
复制。我们还应用了几种最新的方法进行遗传关联检验,以增加功效,发现在简单的病例对照
GWAS
中未
发现的其他关联。我们评估了遗传多效性以研究与多性状相关的基因。在对
KoGES
和
BBJ
之间的
32
种表型
进行荟萃分析后,我们进一步确定了
379
种新的关联,并通过使用荟萃分析结果证明了多基因风险评分的预
测性能
76
个
KoGES GWAS
表型的汇总统计是公开的,有助于更好地理解东亚人群的遗传结构。
介绍
基于人群的生物库,如UK Biobank
2
,
3
和FinnGen,
4
促进了数万
甚至数百万样本中大量性状的全基因组关联研究(GWAS)这些
丰富的资源有助于确定许多遗传关联,并阐明复杂性状的遗传组
成
5
、
6
利用分析结果,建立了基于基因组的预测模型,并成功地
识别了具有高疾病风险的个体尽管取得了成功,但GWAS目前的
一个主要限制是相对缺乏非欧洲样本。
7
由于欧洲人的罕见变异
在其他祖先群体中可能具有较高的次要等位基因频率(MAF),
因此缺乏非欧洲样本可能会限制进一步的发现。此外,如果在临
床实践中使用遗传发现仅限于欧洲血统的个人,可能会导致
8
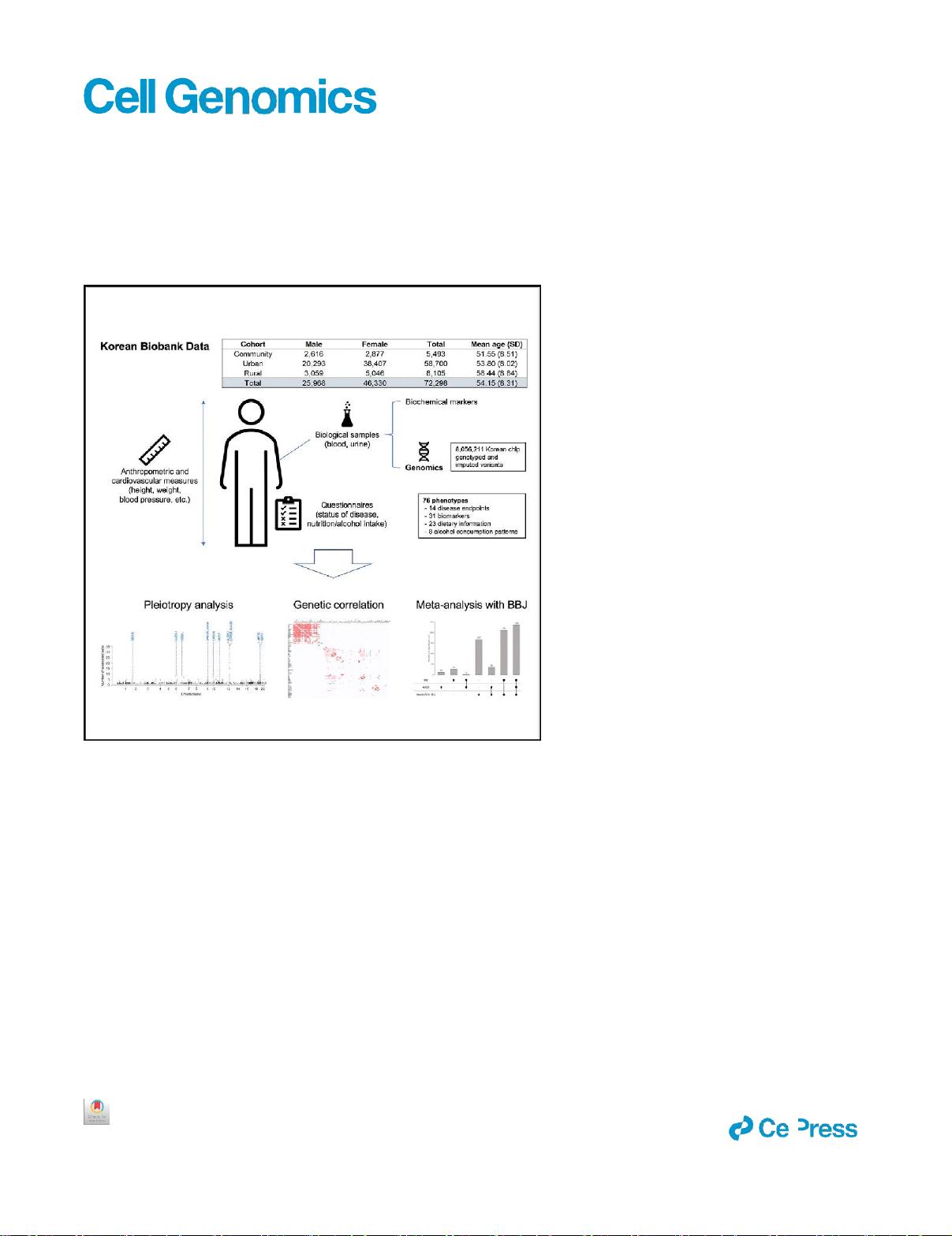
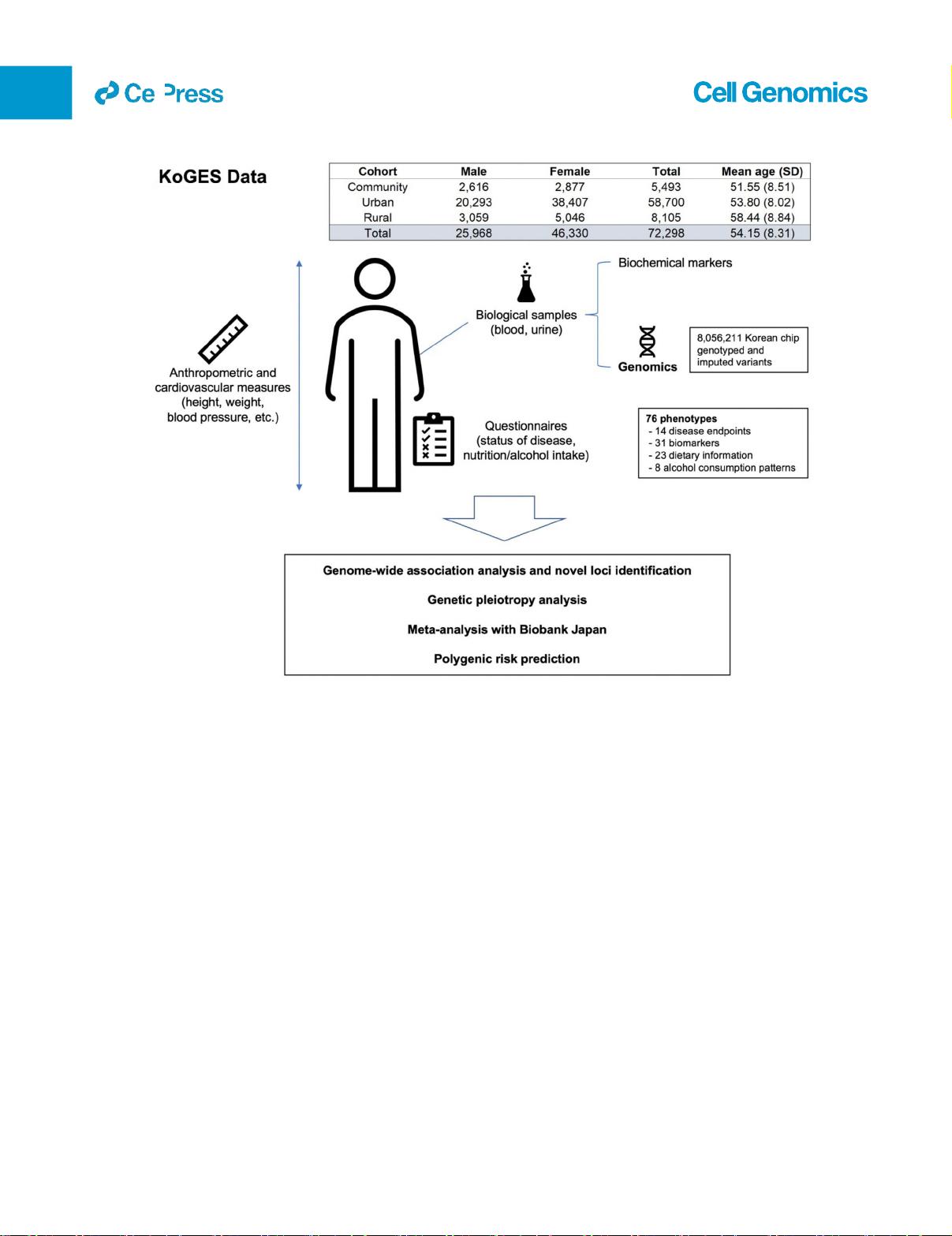
我们报告了来自韩国基因组和流行病学研究(KoGES)的
72,298名韩国人的76种表型的GWAS以前使用KoGES数据进
行 了 几 次 GWAS , 包 括 用 于 人 体测 量 特 征 和 一 些 代 谢 物 的
GWAS。
9
最近,通过分析大量的表型,包括对日本生物银行
(BBJ)
12
-14和台湾生物银行15数据的全表型分析,已经做出了
重大努力来对东亚人的遗传关联进行通过增加东亚GWAS样本的
样本量和人口多样性通过分析14个二元疾病终点、31个生物标
志物、23个饮食信息和8个饮酒量
表型,我们在全基因组显著性水平上鉴定了47种表型的2,242
个相关基因座(P 53 10
- 8
)。为了充分利用KoGES数据中的信
息,除了连续、二元和分类表型的混合效应模型外,我们还应用
了最新的生存分析方法GWAS
16
和纳入家族病史的方法
4
,并确定
了19个额外的显著相关性。在关联中,122例为新关联,在BBJ
中,超过70%的具有相应表型和遗传变异的新许多新的基因座在
欧洲人中具有非常低的MAF,这表明通过利用来自不同祖先群体
的样本可以增加功率为了找到东亚特有的遗传关联,我们使用
KoGES和BBJ(n = 251,000)GWAS结果对32个性状我们发
现了25个性状的379个新基因座,大多数是临床生物标志物,其
中85%的基因座在个体研究中未被发现我们还使用荟萃分析的
GWAS摘要构建了多基因风险评分(PRS),并表明与仅从BBJ
GWAS训练的PRS相比,使用荟萃分析的KoGES和BBJ训练的
PRS在预测UK Biobank中东亚样本的性状值时可以具有20%的
R2
,这表明我们的分析结果的一个潜在效用我们公开提供所有
GWAS汇总统计数据,以扩大对
东亚人群的遗传基础
结果
KoGES
KoGES是韩国国家生物库的一部分,是一项前瞻性队列研究,
CellGenomics 2,100189,October 12,2022
<
$2022作者。1
这是一篇基于CC BY-NC-ND许可证的开放获取文章(http://creativecommons.org/licenses/by-nc-nd/4.0/)。