生物信息学序列分析新境界:CRF模型在基因预测中的应用
发布时间: 2024-08-21 01:55:28 阅读量: 96 订阅数: 46 

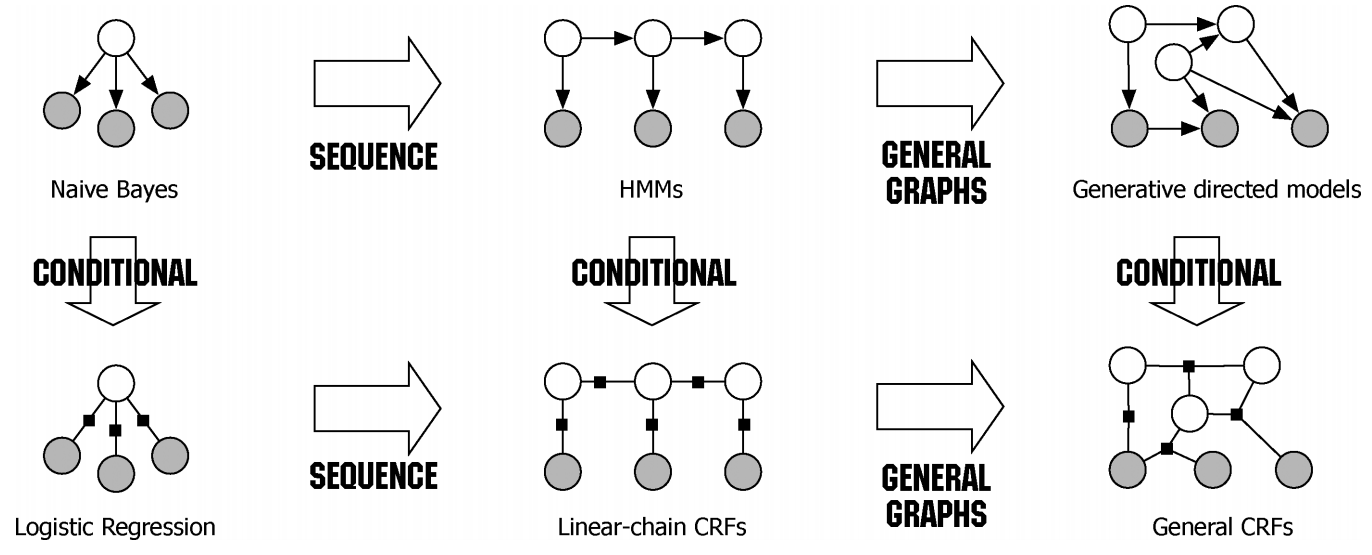
# 1. 序列分析基础与CRF模型简介
序列分析在生物信息学中至关重要,用于分析DNA、RNA和蛋白质序列。条件随机场(CRF)模型是一种强大的序列分析技术,它将序列建模为一组相互依赖的随机变量,并根据观察到的序列预测隐藏状态。
CRF模型的优势在于它可以捕获序列中局部和全局特征之间的复杂依赖关系。它在基因预测中得到了广泛的应用,因为基因序列具有高度结构化和上下文依赖性的特点。CRF模型能够有效地识别基因的起始和终止位置,并预测基因的类型和功能。
# 2. CRF模型在基因预测中的理论基础
### 2.1 条件随机场模型原理
#### 2.1.1 模型定义和基本概念
条件随机场(CRF)是一种概率图模型,用于对序列数据进行建模和预测。它假设序列中的每个元素(如基因序列中的碱基)的标签(如基因或非基因)取决于其相邻元素的标签。
CRF模型由以下元素定义:
- **无向图 G**:表示序列中的元素之间的依赖关系。
- **随机变量 Y**:表示序列中每个元素的标签。
- **特征函数 F**:将序列和标签映射到实数。
- **势函数 ψ**:将特征函数映射到实数。
#### 2.1.2 模型训练和预测算法
CRF模型的训练和预测通常使用以下算法:
- **训练:**
- 使用最大似然估计(MLE)或变分推断优化模型参数。
- 优化目标函数,该函数衡量模型对训练数据的似然性。
- **预测:**
- 使用维特比算法或置信传播算法查找序列中最可能的标签序列。
- 维特比算法是一种动态规划算法,用于在给定观察序列的情况下找到最可能的隐含状态序列。
### 2.2 CRF模型在基因预测中的应用场景
#### 2.2.1 基因预测的挑战和难点
基因预测是一个具有挑战性的任务,因为:
- 基因序列中存在噪声和不确定性。
- 基因边界可能模糊或重叠。
- 不同物种的基因结构差异很大。
#### 2.2.2 CRF模型的优势和适用性
CRF模型非常适合基因预测,因为它:
- 可以捕获序列中元素之间的依赖关系。
- 可以处理噪声和不确定性。
- 可以通过特征工程和超参数调优进行定制。
- 具有良好的泛化能力,可以在不同物种的基因序列上进行预测。
# 3. CRF模型在基因预测中的实践应用
### 3.1 基因序列数据的预处理
#### 3.1.1 序列质量控制和过滤
在基因预测之前,对原始基因序列数据进行质量控制和过滤至关重要。这包括:
- **去除低质量碱基:**使用质量分数阈值去除质量低于特定水平的碱基。
- **去除重复序列:**使用比对工具识别和去除重复序列,因为它们可能干扰预测。
- **去除污染序列:**使用比对工具识别和去除来自其他物种或实验污染的序列。
#### 3.1.2 特征提取和工程
特征提取是将原始基因序列数据转换为可用于机器学习模型的特征向量的过程。对于基因预测,常用的特征包括:
- **碱基序列:**序列中碱基的顺序。
- **碱基频率:**序列中不同碱基的频率。
- **二元组和三元组:**序列中相邻碱基对或三元组的频率。
- **开读框:**潜在蛋白质编码区域的序列片段。
- **同源性:**与已知基因序列的相似性。
特征工程涉及对提取的特征进行转换和组合,以增强模型的性能。这可能包括:
- **归一化:**将特征值缩放或转换到相同范围内。
- **特征选择:**选择与预测目标最相关的特征子集。
- **特征组合:**创建新的特征,例如特征交互或转换。
### 3.2 CRF模型的训练和调优
#### 3.2.1 训练数据集的构建和划分
训练数据集是用于训练CRF模型的基因序列数据集合。它应包含代表目标基因组多样性的样本。训练数据集通常被划分为训练集、验证集和测试集:
- **训练集:**用于训练模型的参数。
- **验证集:**用于调整模型超参数和防止过拟合。
- **测试集:**用于评估训练后的模型性能。
#### 3.2.2 模型超参数的优化和选择
CRF模型的超参数控制模型的行为,例如正则化参数和特征权重。超参数的优化对于模型性能至
0
0





