【生态数据分析全流程】:从PC-ORD数据清洗到结果输出
发布时间: 2025-01-06 13:16:50 阅读量: 10 订阅数: 10 

# 摘要
生态数据分析是理解和解释生态系统动态的关键步骤,本文系统地介绍了生态数据的处理与分析方法。首先,阐述了生态数据的特点和分析的重要性,并介绍了PC-ORD软件在数据导入与前期处理中的作用。随后,深入探讨了数据处理中的缺失值处理、异常值检测以及数据标准化和变换等关键步骤。统计分析部分详细介绍了描述性统计、推断性统计以及多变量分析技术的应用。高级主题章节进一步探讨了空间分析、时间序列分析以及生态模型的构建和预测。最后,本文还讨论了数据可视化技术、结果的解释与应用,并提供了撰写和发布分析报告的指导。本文为生态研究者提供了一套完整的数据分析流程和工具,帮助他们更有效地进行生态学研究和数据分析。
# 关键字
生态数据;PC-ORD软件;数据缺失值;异常值检测;多变量分析;数据可视化;GIS;时间序列分析;生态模型;统计解释
参考资源链接:[PC-ORD生态数据分析全面教程:多变量方法详解](https://wenku.csdn.net/doc/35hh814b0v?spm=1055.2635.3001.10343)
# 1. 生态数据的特点与分析意义
## 1.1 生态数据的定义与特性
生态数据是记录自然界生物及其环境相互作用的数值信息。它通常具有高维度、非线性、时序性和空间依赖性等特点。生态数据的获取往往依赖于长期的野外观察和实验,以及通过现代遥感技术、GIS(地理信息系统)等手段采集的环境数据。理解生态数据的这些特点对于后续的数据处理和分析至关重要。
## 1.2 生态数据分析的意义
生态数据分析可以帮助科学家们解释复杂的生物与环境关系,预测生态系统的变化趋势,以及指导自然资源的可持续管理。通过对生态数据的深入分析,可以揭示物种多样性、生态过程、群落结构及其对环境变化的响应,为生态保护和生态服务功能的评估提供科学依据。
## 1.3 分析方法与工具的选择
在分析生态数据时,选择合适的方法和工具至关重要。生态学研究者常使用统计分析软件,如PC-ORD、R语言、SPSS等,对数据进行描述性统计、假设检验、多变量分析等操作。而随着机器学习和大数据技术的发展,更多高级技术如神经网络、深度学习也被应用于生态数据挖掘和模式识别中。选择正确的分析工具和技术路径,可以提高分析效率,确保分析结果的准确性和可靠性。
# 2. PC-ORD软件简介与数据导入
## 2.1 PC-ORD软件功能概述
### 2.1.1 软件界面与主要功能模块介绍
PC-ORD是一款专业的生态数据分析软件,由MjM Software设计。软件界面直观,模块化操作方便快捷,适用于生态学家、环境科学家以及生物多样性研究人员进行数据处理和统计分析。PC-ORD的主要功能模块包括数据管理、多样性分析、相似性和排序、以及群落分析等。
在软件界面的左侧,是软件的"项目"部分,用户可以在此创建和管理项目。右侧则是"项目视窗",显示当前项目的概览和各个模块。在"数据视窗"中,用户可以输入和编辑生态数据表格。"图表视窗"用于绘制和查看数据分析的图形结果,如排序图和分类树。
此外,PC-ORD提供了多个模块用于特定类型的分析,例如:
- 多样性分析模块用于计算物种丰富度、多样性指数等;
- 排序模块用于非度量多维尺度分析(NMDS)和主坐标分析(PCoA);
- 群落分析模块用于TWINSPAN和指示种分析等。
### 2.1.2 数据导入的基本流程
要将生态数据导入PC-ORD中,首先需要准备数据文件。推荐使用制表符分隔的文本文件(.txt),或者CSV文件格式,因为这些格式兼容性好,可以方便地被PC-ORD读取。数据文件通常包含物种出现的样方数据,包括样方标识、物种标识以及相应的数值数据。
基本的数据导入步骤如下:
1. 打开PC-ORD,选择"File"(文件)菜单中的"Open"(打开)选项,选择需要导入的数据文件。
2. 在打开的数据导入向导中,选择适当的"File type"(文件类型),并指定数据文件的分隔符,例如制表符、逗号等。
3. 检查导入预览中的数据,确认各个字段(列)的标题是否正确,以及数据是否被正确识别。
4. 如果数据中包含物种名称和样方名称,需要在下一步设置这些列为"Codes"(代码列),其他为"Values"(值列)。
5. 按照提示完成导入过程,并确认数据已正确导入项目中。
导入数据后,可以通过"Data Editor"(数据编辑器)查看和编辑数据,确保数据准确无误。
## 2.2 数据集的前期准备
### 2.2.1 数据格式的确认与转换
生态数据常常来源于不同的研究和测量,因此格式可能会有所差异。在导入PC-ORD之前,确保数据格式是统一的,至少应包括样方标识、物种标识和相应的数值数据。数据格式的确认包括检查是否每行代表一个样方,每列代表一个物种的出现情况,以及检查是否有缺失值。
在一些情况下,可能需要将数据转换成PC-ORD可以接受的格式。例如,将Excel文件转换成CSV格式。这可以通过以下步骤完成:
1. 在Excel中打开要转换的数据文件。
2. 选择"另存为"选项,并从下拉菜单中选择"CSV (逗号分隔) (*.csv)"。
3. 在保存对话框中,确保"Unicode (UTF-8)"编码被选中,并取消勾选"包含文字导入向导"。
4. 点击"保存"按钮完成转换。
如果需要处理更复杂的格式转换问题,可以使用R语言、Python等编程语言中的数据处理库,如`pandas`或`dplyr`等,进行数据预处理和格式转换。
### 2.2.2 数据来源的描述与记录
在进行数据分析之前,记录数据来源是非常重要的。这不仅有助于研究的透明度,还便于其他研究者对数据集进行验证或重复分析。数据来源应包括以下信息:
- 数据收集的地理位置、时间、环境条件等;
- 数据的收集方法和设备,例如样方的大小、采样深度等;
- 数据的提供者和采集人信息;
- 如有文献引用,提供相关的参考文献信息。
在PC-ORD中,建议创建一个包含这些信息的文本文件,并将其保存在项目文件夹中。在分析报告中,这些信息应详细记录,以便读者能够追溯和理解数据的背景。
## 2.3 数据清洗的策略与步骤
### 2.3.1 数据清洗的必要性
数据清洗是数据分析前的一个重要步骤,特别是在生态学研究中,数据往往庞大且复杂。未经清洗的数据中可能包含错误、异常值或缺失值,这些都会影响数据分析的准确性和结果的可靠性。通过数据清洗可以保证数据的质量,提高分析和解释数据的效率。
数据清洗的必要性具体体现在:
- 提高数据质量:通过清洗数据中的错误和异常值,提高数据的整体质量;
- 提升分析准确性:准确的分析依赖于清洁的数据集,错误的数据会导致错误的分析结果;
- 加快分析处理速度:数据清洗可以减少在后续分析中的数据问题处理时间。
### 2.3.2 数据清洗的标准操作流程
在PC-ORD中,数据清洗通常包括以下标准操作流程:
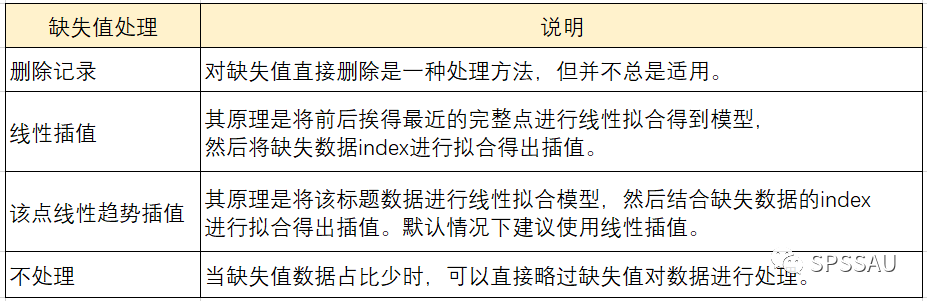
1. **识别和处理缺失值**:生态数据可能因测量或记录错误而产生缺失值。PC-ORD允许用户通过"Data Editor"直接查看和修改数据。缺失值可以用0、NA或者特定的标记表示,应根据具体情况和研究目的决定是否需要填充或删除这些值。
示例代码块:
```R
# 使用R语言识别和处理数据集中的缺失值
data <- read.csv("ecology_data.csv") # 读取数据文件
na_count <- sapply(data, function(y) sum(length(which(is.na(y)))))
print(na_count) # 打印出每列的缺失值数量
# 假设我们选择填充缺失值为该列的平均值
data[is.na(data)] <- mean(data, na.rm = TRUE) # 填充缺失值
```
2. **检测和修正错误值**:错误值可能是数据输入错误或异常的结果。在数据清洗过程中,应检查数值范围和逻辑一致性,例如物种数量不应为负数。
示例代码块:
```R
# 检查并修正物种数量为负数的错误值
data <- read.csv("ecology_data.csv") # 读取数据文件
# 检测物种数量小于0的记录,并进行修正
data[data < 0] <- NA # 将错误值设置为缺失
# 再次进行数据清洗,例如填充这些缺失值为平均值
data[is.na(data)] <- mean(data, na.rm = TRUE)
```
3. **识别和处理异常值**:异常值可能是测量误差或极端变量,可能对分析结果造成误导。可以使用统计方法或可视化手段来识别异常值,并决定是否需要将其从数据集中移除或进行调整。
4. **标准化数据格式**:不同的研究可能采用不同的数据表示方式,例如物种名称。统一数
0
0






