【UCINET 6网络比较】:多网络数据比较分析的专家级教程
发布时间: 2024-12-16 18:30:26 阅读量: 2 订阅数: 5 


医学分割数据集肾结石分割数据集labelme格式359张1类别.zip
参考资源链接:[UCINET 6 for Windows中文手册:详解与资源指南](https://wenku.csdn.net/doc/7enj0faejo?spm=1055.2635.3001.10343)
# 1. UCINET 6网络分析基础
## 1.1 网络分析简介
网络分析是一种使用图论和数学方法来研究网络结构和属性的技术。在社会学、生物学、信息科学等多个领域,网络分析帮助我们了解实体间的关系与复杂系统的运作机制。UCINET(University of California at Irvine's Network, 简称UCINET)作为专业的社会网络分析软件,提供了丰富的工具和算法用于处理网络数据。
## 1.2 UCINET 6的安装与基本界面
在介绍UCINET 6的网络分析功能之前,首先需要确保软件的正确安装。可以从官方网站下载软件安装包并遵循安装向导进行安装。安装完成后,启动UCINET,用户会看到一个简洁的主界面,界面中包含了多种网络分析的功能入口,如网络数据输入、网络图绘制、中心性分析等。
```mermaid
graph LR
A[启动UCINET 6] --> B[查看主界面]
B --> C[选择分析功能]
```
## 1.3 网络分析的基本概念
在进行网络分析之前,需要理解几个核心概念。节点(node)代表网络中的个体,边(edge)表示节点间的关联。节点的度(degree)是与其直接相连的边的数量,它反映了节点的活跃程度。一个网络可以是无向的,表示关系不区分方向;也可以是有向的,即关系有明确的方向性。
通过UCINET 6的界面和基本概念的讲解,为后续更深入的网络分析打下了坚实的基础。在后续章节中,我们将详细探讨如何导入数据、预处理数据、进行网络比较,以及如何通过案例来实践我们的网络分析技能。
# 2. 多网络数据的导入与预处理
数据的导入与预处理是进行复杂网络分析前的必要步骤。本章节将介绍如何有效地导入多源网络数据,并进行必要的预处理操作,以保证后续分析的准确性与有效性。
## 2.1 数据导入技巧
### 2.1.1 支持的数据格式和导入方法
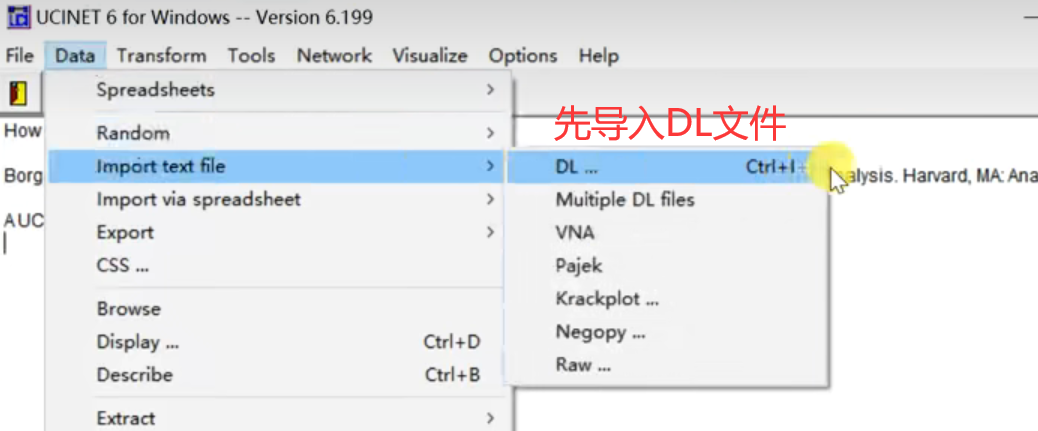
UCINET 支持多种数据格式,如 UCINET DL、Pajek、UCINET Full、GraphML 等。在导入数据时,正确选择和理解各种格式是第一步。
1. **UCINET DL 格式** - 这是 UCINET 专用的简单文本格式,包含网络节点、边和属性等信息。
2. **Pajek 格式** - 适用于大规模网络,允许存储额外的属性和分层结构。
3. **UCINET Full 格式** - 包含节点、边、属性数据以及网络的元数据。
4. **GraphML 格式** - 是 XML 格式的网络数据交换格式,适合描述复杂网络。
导入数据时,可以通过界面选择“File -> Import -> Networks”,然后选择合适的格式进行导入。此外,UCINET 还支持直接从 Excel 文件导入数据,但需要按照其数据模板进行格式化。
### 2.1.2 数据格式转换和验证
不同格式之间的转换通常需要使用专门的工具或脚本进行。以下是一个简单的数据格式转换和验证的示例代码块:
```R
# R 语言代码示例,用于转换数据格式
library(igraph)
# 从 Pajek 格式转换为 GraphML
pajek_data <- read.graph(file="pajek_file.net", format="pajek")
write.graph(pajek_data, file="graphml_output.xml", format="graphml")
# 验证 GraphML 数据
graphml_data <- read.graph(file="graphml_output.xml", format="graphml")
if (is.igraph(graphml_data)) {
print("数据格式转换成功,可以导入 UCINET。")
} else {
print("数据格式转换失败,请检查错误。")
}
```
在此代码段中,我们首先读取了 Pajek 格式的网络数据,然后将其转换为 GraphML 格式,并进行了验证,以确保转换后的数据没有丢失或错误。
## 2.2 数据清洗与预处理
### 2.2.1 缺失值处理
网络数据集中的缺失值是一个常见的问题。正确的缺失值处理方式会直接影响分析的结果。
使用 R 语言进行缺失值处理的常见方法如下:
```R
# R 语言处理缺失值
data <- read.graph(file="input_data.graphml", format="graphml")
# 删除含有缺失值的节点或边
clean_data <- delete.edges(data, which(is.na(E(data)$weight)))
clean_data <- delete.vertices(clean_data, which(is.na(V(clean_data)$label)))
# 如果缺失值不多,也可以选择填充(例如,用平均值填充)
fill_data <- data
E(fill_data)$weight[is.na(E(fill_data)$weight)] <- mean(E(fill_data)$weight, na.rm=TRUE)
V(fill_data)$label[is.na(V(fill_data)$label)] <- mean(V(fill_data)$label, na.rm=TRUE)
```
在上面的代码中,`delete.edges` 和 `delete.vertices` 函数用于删除那些含有缺失值的边和节点。如果数据集中的缺失值不多,可以使用 `mean` 函数计算无缺失值的平均值,然后用此平均值填充缺失值。
### 2.2.2 异常值处理与标准化
异常值可能会对网络分析产生误导性的影响,因此需要进行处理。标准化是为了消除由于量纲不同带来的影响,使得不同指标之间能够相互比较。
一个处理异常值和标准化数据的示例代码如下:
```R
# 处理异常值和标准化
# 使用 Z-score 方法进行标准化
network_data <- read.graph(file="clean_data.graphml", format="graphml")
std_network <- as.matrix(igraph::as_adjacency_matrix(network_data, type="both"))
for(i in 1:nrow(std_network)) {
std_network[i,] <- (std_network[i,] - mean(std_network[i,])) / sd(std_network[i,])
}
# 创建新的标准化网络对象
std_network_
```
0
0





