NSGA-II多目标优化算法在自然语言处理中的应用:优化语言模型,提升语言理解
发布时间: 2024-08-20 00:23:00 阅读量: 24 订阅数: 29 

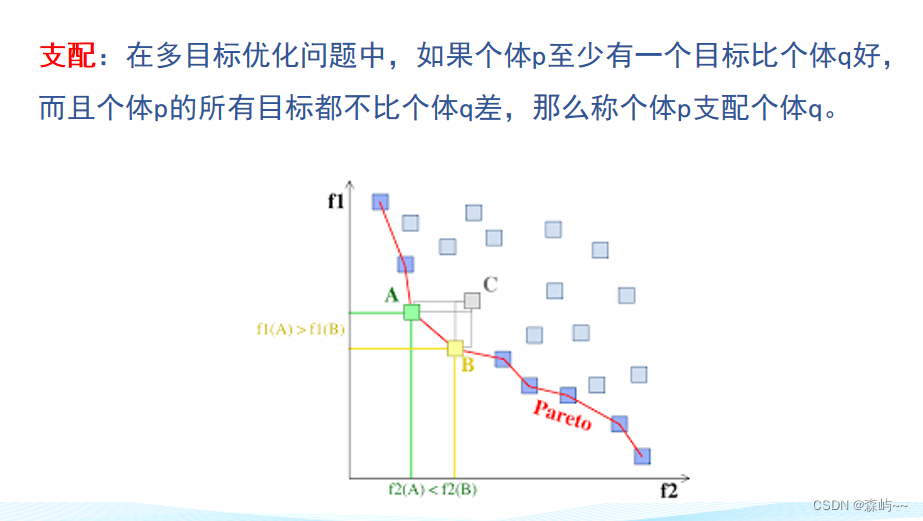
# 1. NSGA-II算法概述**
NSGA-II(非支配排序遗传算法 II)是一种多目标优化算法,用于解决具有多个相互冲突的目标的优化问题。该算法基于以下关键概念:
- **非支配排序:**将个体按其支配关系进行排序,其中一个个体支配另一个个体,如果它在所有目标上都优于后者,或者至少在一个目标上优于后者且在其他目标上不劣。
- **拥挤距离:**衡量个体在目标空间中与其他个体的拥挤程度,拥挤距离较大的个体更有可能被选择。
- **快速非支配排序:**一种快速计算非支配排序和拥挤距离的算法,提高了算法的效率。
# 2. NSGA-II算法在语言模型优化中的应用
### 2.1 语言模型的理论基础
#### 2.1.1 语言模型的定义和类型
语言模型是概率分布,描述了给定序列中单词出现的概率。语言模型用于预测下一个单词、生成文本和翻译语言。语言模型的类型包括:
- **n元语法模型:**基于前n个单词预测下一个单词的概率。
- **神经语言模型:**使用神经网络学习语言的分布,可以捕获更复杂的语言模式。
- **混合语言模型:**结合不同类型的语言模型以提高性能。
#### 2.1.2 语言模型的评估指标
语言模型的性能通常使用以下指标评估:
- **困惑度:**衡量模型预测单词序列的难度,困惑度越低越好。
- **对数似然:**衡量模型对给定单词序列的拟合程度,对数似然越高越好。
- **BLEU分数:**衡量机器翻译输出与参考翻译之间的相似度。
### 2.2 NSGA-II算法优化语言模型
NSGA-II(非支配排序遗传算法 II)是一种多目标优化算法,可以同时优化多个目标函数。在语言模型优化中,NSGA-II用于优化困惑度和对数似然等目标。
#### 2.2.1 编码方案和目标函数设计
**编码方案:**通常使用实数编码,每个基因代表语言模型的参数,如单词嵌入或神经网络权重。
**目标函数:**目标函数通常包括:
- **困惑度:**最小化困惑度以提高模型的预测能力。
- **对数似然:**最大化对数似然以提高模型对语言数据的拟合程度。
- **多样性:**最大化种群多样性以避免陷入局部最优。
#### 2.2.2 进化过程和参数设置
NSGA-II的进化过程包括:
1. **初始化种群:**随机生成一组语言模型参数。
2. **评估种群:**计算每个模型的困惑度、对数似然和多样性。
3. **非支配排序:**根据目标函数将模型分为不同的非支配等级。
4. **拥挤距离计算:**计算每个模型在非支配等级中的拥挤距离。
5. **选择:**根据非支配等级和拥挤距离选择模型进行交叉和变异。
6. **交叉和变异:**使用交叉和变异算子生成新的模型。
7. **替换:**将新的模型添加到种群中,并删除最差的模型。
**参数设置:**NSGA-II算法的参数设置包括种群大小、交叉概率、变异概率和进化代数。这些参数需要根据具体问题进行调整。
```python
import numpy as np
import random
class NSGAII:
def __init__(self, population_size, crossover_probability, mutation_probability, num_generations):
self.population_size = population_size
self.crossover_probability = crossover_probability
self.mutation_probability = mutation_probability
self.num_generations = num_generations
def initialize_population(self):
population = []
for i in range(self.population_size):
individual = np.random.rand(10) # 10-dimensional individual
population.append(individual)
return population
def evaluate_population(self, population):
for individual in population:
# Calculate perplexity and log-likelihood
perplexity = ...
log_likelihood = ...
# Calculate diversity
diversity = ...
return population
def non_dominated_sorting(self, population):
# Sort the population into non-dominated fronts
fronts = []
while population:
front = []
for individual in population:
if individual.dominated_count == 0:
front.append(individual)
population = [individual for individual in population if individual not in front]
fronts.append(front)
return fronts
def crowding_distance_assignment(self, front):
# Calculate the crowding distance for each individual in the front
for individual in front:
individual.crowding_distance = ...
return front
def selection(self, population):
# Select individuals for crossover and mutation
selected_individuals = []
while len(selected_individuals) < self.population_size:
# Select an individual based on non-dominated rank and crowding distance
i
```
0
0





