Python生物信息学实战指南:从零开始分析基因序列,揭秘生物信息学数据处理利器
发布时间: 2024-06-20 02:11:02 阅读量: 304 订阅数: 33 


生物信息python中文版.pdf
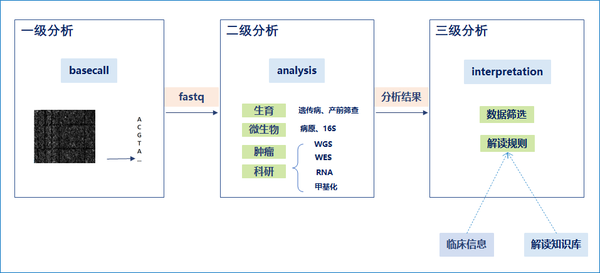
# 1. Python生物信息学概述
Python生物信息学是一门利用Python编程语言来解决生物信息学问题的学科。它结合了Python强大的数据处理能力和生物信息学领域特有的算法和工具,为生物信息学研究提供了高效、灵活的解决方案。
Python生物信息学在基因组学、蛋白质组学、代谢组学等多个生物信息学领域都有广泛的应用。它可以帮助研究人员读取、处理、分析和可视化生物数据,从而获得生物学见解,促进药物发现、疾病诊断和治疗等领域的进展。
# 2. Python生物信息学基础
### 2.1 Python序列处理库
#### 2.1.1 Biopython简介
Biopython是一个用于生物信息学计算的Python库。它提供了一组模块和类,用于处理各种生物信息学数据类型,包括序列、序列比对、基因组注释和蛋白质结构。
#### 2.1.2 Biopython序列处理模块
Biopython提供了几个模块用于序列处理,包括:
* **Seq:** 用于表示和操作序列对象。
* **SeqRecord:** 用于表示带有附加信息的序列对象,例如注释和质量分数。
* **SeqFeature:** 用于表示序列中的特征,例如基因、外显子和内含子。
* **Align:** 用于表示和操作序列比对。
### 2.2 Python数据处理技术
#### 2.2.1 NumPy简介
NumPy是一个用于科学计算的Python库。它提供了一个多维数组对象,以及用于数组操作和数学运算的函数。
#### 2.2.2 Pandas简介
Pandas是一个用于数据分析和操作的Python库。它提供了一个数据结构称为DataFrame,它是一个带有行和列的表格状数据结构。Pandas还提供了用于数据清洗、转换和可视化的函数。
#### 2.2.3 代码示例
```python
# 使用NumPy创建多维数组
import numpy as np
array = np.array([[1, 2, 3], [4, 5, 6]])
print(array)
# 使用Pandas创建DataFrame
import pandas as pd
data = {'Name': ['John', 'Jane', 'Jack'], 'Age': [20, 25, 30]}
df = pd.DataFrame(data)
print(df)
```
**逻辑分析:**
* NumPy的`array`函数创建了一个多维数组,其中数据按行和列组织。
* Pandas的`DataFrame`函数创建一个DataFrame,其中数据存储在带有列名的行和列中。
**参数说明:**
* **NumPy.array():**
* **array:**要创建的数组。
* **Pandas.DataFrame():**
* **data:**要创建DataFrame的数据。
# 3.1 基因序列读取和预处理
#### 3.1.1 序列文件格式
基因序列通常存储在文本文件中,其中每行代表一条序列。这些文件通常使用特定的格式,如 FASTA、FASTQ 和 GenBank。
- **FASTA 格式:**以 ">" 符号开头,后跟序列标识符和可选描述。序列本身从下一行开始,直到遇到另一个 ">" 符号。
```
>sequence_id
ATCGATCGATCGATCG
```
- **FASTQ 格式:**类似于 FASTA,但每行包含四个字段,分别表示序列标识符、序列、质量分数和可选描述。
```
@sequence_id
ATCGATCGATCGATCG
+sequence_id
!**&**"*+,-./0123456789:;<=>?@ABCDEFGHI
```
- **GenBank 格式:**一种更复杂的格式,包含序列信息、注释和元数据。它通常用于存储来自 GenBank 数据库的序列。
#### 3.1.2 序列读取和转换
Python 提供了多种库来读取和转换序列文件。
- **Biopython:**一个广泛使用的生物信息学库,提供 `Bio.SeqIO` 模块用于读取和写入各种序列格式。
```python
from Bio import SeqIO
# 读取 FASTA 文件
for record in SeqIO.parse("sequence.fasta", "fasta"):
print(record.id, record.seq)
# 写入 FASTQ 文件
SeqIO.write(record, "sequence.fastq", "fastq")
```
- **SeqUtils:**一个较小的库,提供类似的功能,并专注于处理 FASTQ 文件。
```python
import seqUtils
# 读取 FASTQ 文件
sequences = seqUtils.readFasta("sequence.fasta")
# 写入 FASTA 文件
seqUtils.writeFastq(sequences, "sequence.fastq")
```
- **Pandas:**一个数据处理库,可以将序列文件读入 DataFrame 中,以便进行进一步的处理。
```python
import pandas as pd
# 读取 FASTA 文件
df = pd.read_csv("sequence.fasta", sep="\n", header=None)
df.columns = ["id", "sequence"]
```
# 4.1 基因组注释和功能分析
### 4.1.1 基因组注释数据库
基因组注释是将基因组序列与生物学功能信息相关联的过程。注释数据库包含大量已知基因、蛋白质和功能信息,为基因组序列提供参考和注释。常用的基因组注释数据库包括:
- **GenBank:**由美国国家生物技术信息中心(NCBI)维护,包含来自不同生物体的序列和注释信息。
- **EMBL-EBI:**欧洲生物信息学研究所维护,提供序列、注释和生物信息学工具。
- **UniProt:**由欧洲生物信息学研究所和瑞士生物信息学研究所联合维护,包含蛋白质序列、功能和注释信息。
### 4.1.2 基因功能预测
基因功能预测是根据序列信息推测基因功能的过程。常用的方法包括:
- **同源性搜索:**通过将序列与已知功能的序列进行比较,预测其功能。
- **基因本体(GO)注释:**使用受控词汇表对基因功能进行分类和注释。
- **机器学习:**利用机器学习算法,根据序列特征预测基因功能。
```python
# 使用 Biopython 访问 GenBank 记录
from Bio import Entrez
# 设置电子邮件地址以遵守 NCBI 使用条款
Entrez.email = "your_email@example.com"
# 搜索 GenBank 记录
handle = Entrez.esearch(db="nucleotide", term="Homo sapiens")
record = Entrez.read(handle)
# 获取记录的详细信息
for item in record["IdList"]:
handle = Entrez.efetch(db="nucleotide", id=item, rettype="gb", retmode="text")
sequence_record = Entrez.read(handle)
print(sequence_record["GBSeq_definition"])
# 逻辑分析:
此代码使用 Biopython 库访问 GenBank 数据库,搜索人类序列并检索记录的详细信息,包括序列定义。
```
## 4.2 生物信息学数据可视化
### 4.2.1 Matplotlib 简介
Matplotlib 是一个用于创建交互式和静态可视化的 Python 库。它提供了一组广泛的绘图类型和功能,包括折线图、散点图、直方图和饼图。
```python
# 使用 Matplotlib 绘制折线图
import matplotlib.pyplot as plt
# 生成数据
x = [1, 2, 3, 4, 5]
y = [2, 4, 6, 8, 10]
# 绘制折线图
plt.plot(x, y)
plt.xlabel("X-axis")
plt.ylabel("Y-axis")
plt.title("折线图")
plt.show()
# 逻辑分析:
此代码使用 Matplotlib 绘制一个简单的折线图,其中 x 轴表示独立变量,y 轴表示因变量。
```
### 4.2.2 Seaborn 简介
Seaborn 是一个基于 Matplotlib 构建的高级数据可视化库。它提供了一个更高级别的 API,简化了复杂可视化的创建,例如分布图、热图和聚类图。
```python
# 使用 Seaborn 绘制分布图
import seaborn as sns
# 生成数据
data = [1, 2, 3, 4, 5, 6, 7, 8, 9, 10]
# 绘制分布图
sns.distplot(data)
plt.xlabel("Value")
plt.ylabel("Frequency")
plt.title("分布图")
plt.show()
# 逻辑分析:
此代码使用 Seaborn 绘制一个分布图,显示给定数据值的分布。
```
# 5. Python生物信息学案例研究
### 5.1 基因表达分析
#### 5.1.1 RNA-Seq数据处理
**RNA-Seq数据格式**
RNA-Seq数据通常以FASTQ格式存储,其中包含序列和质量分数信息。FASTQ文件由四行组成:
```
@HWI-ST120324:1:1:1001:1000
+HWI-ST120324:1:1:1001:1000
!''*((((***+))%%%++**-+>>>**++-CC>
```
* 第一行是序列标识符,包含仪器名称、运行号、通道号、序列号和读数号。
* 第二行是序列,由碱基字母(A、C、G、T)组成。
* 第三行是分隔符(+)。
* 第四行是质量分数,表示每个碱基被正确测序的可能性。
**RNA-Seq数据预处理**
RNA-Seq数据预处理涉及以下步骤:
1. **质量过滤:**去除低质量的读数,例如质量分数低于特定阈值的读数。
2. **修剪:**去除序列两端的低质量碱基。
3. **适配器修剪:**去除测序过程中引入的适配器序列。
4. **比对:**将预处理后的读数比对到参考基因组。
5. **计数:**计算每个基因的读数计数。
#### 5.1.2 基因表达差异分析
**差异表达基因的鉴定**
差异表达基因的鉴定涉及以下步骤:
1. **标准化:**对不同样本的读数计数进行标准化,以消除技术变异的影响。
2. **差异分析:**使用统计方法(例如t检验或DESeq2)来识别在不同样本组之间表达差异的基因。
3. **多重检验校正:**校正多重比较的影响,以降低假阳性率。
**差异表达基因的注释**
差异表达基因的注释涉及以下步骤:
1. **基因本体(GO)富集分析:**确定差异表达基因是否富集于特定生物学过程、细胞组分或分子功能。
2. **通路富集分析:**确定差异表达基因是否富集于特定代谢或信号通路。
3. **网络分析:**构建差异表达基因之间的相互作用网络,以了解它们在生物学过程中的关系。
### 5.2 蛋白质组学分析
#### 5.2.1 蛋白质组学数据处理
**蛋白质组学数据格式**
蛋白质组学数据通常以MGF或mzML格式存储,其中包含质谱数据和元数据。MGF文件包含峰值列表,每个峰值由质量荷质比(m/z)、强度和保留时间组成。mzML文件是MGF文件的扩展,包含更多元数据,例如仪器设置和样品信息。
**蛋白质组学数据预处理**
蛋白质组学数据预处理涉及以下步骤:
1. **峰值拾取:**从质谱数据中识别峰值。
2. **峰值对齐:**将不同样本中的峰值对齐到相同的m/z值。
3. **定量:**计算每个峰值的强度。
4. **蛋白质识别:**将峰值匹配到已知的蛋白质序列。
#### 5.2.2 蛋白质组学差异分析
**差异表达蛋白质的鉴定**
差异表达蛋白质的鉴定涉及以下步骤:
1. **标准化:**对不同样本的峰值强度进行标准化,以消除技术变异的影响。
2. **差异分析:**使用统计方法(例如t检验或MSstats)来识别在不同样本组之间表达差异的蛋白质。
3. **多重检验校正:**校正多重比较的影响,以降低假阳性率。
**差异表达蛋白质的注释**
差异表达蛋白质的注释涉及以下步骤:
1. **GO富集分析:**确定差异表达蛋白质是否富集于特定生物学过程、细胞组分或分子功能。
2. **通路富集分析:**确定差异表达蛋白质是否富集于特定代谢或信号通路。
3. **网络分析:**构建差异表达蛋白质之间的相互作用网络,以了解它们在生物学过程中的关系。
# 6. Python生物信息学未来发展
### 6.1 生物信息学云计算
**6.1.1 云计算平台简介**
云计算是一种按需分配的计算资源,包括服务器、存储、数据库、网络、软件、分析和人工智能。它允许用户在无需管理或拥有自己的基础设施的情况下访问这些资源。
**6.1.2 生物信息学云计算应用**
云计算在生物信息学中有着广泛的应用,包括:
- **大数据分析:**云计算平台可以提供大规模计算能力,用于处理和分析海量生物信息学数据。
- **数据存储和管理:**云存储服务提供安全、可扩展且低成本的数据存储和管理解决方案。
- **工作流自动化:**云计算平台可以自动化生物信息学工作流,例如序列比对、注释和可视化。
- **协作和共享:**云平台促进生物信息学家之间的协作和数据共享。
### 6.2 生物信息学人工智能
**6.2.1 人工智能技术简介**
人工智能(AI)是一门计算机科学领域,它让计算机能够执行通常需要人类智能的任务,例如学习、解决问题和决策。
**6.2.2 生物信息学人工智能应用**
AI在生物信息学中有着巨大的潜力,包括:
- **基因组分析:**AI算法可以用于识别基因组中的模式、预测基因功能和诊断疾病。
- **药物发现:**AI可以加速药物发现过程,通过预测药物与靶蛋白的相互作用和优化药物设计。
- **精准医学:**AI可以帮助医生根据患者的基因组和健康数据定制治疗方案。
- **生物医学图像分析:**AI算法可以用于分析医学图像,例如X射线和MRI扫描,以检测疾病和评估治疗效果。
0
0





