MATLAB相关性分析在生物信息学中的应用:揭示基因和蛋白质之间的关系,解锁生物信息学的新可能
发布时间: 2024-06-09 19:35:11 阅读量: 114 订阅数: 57 


Matlab技术在生物信息学中的应用.docx
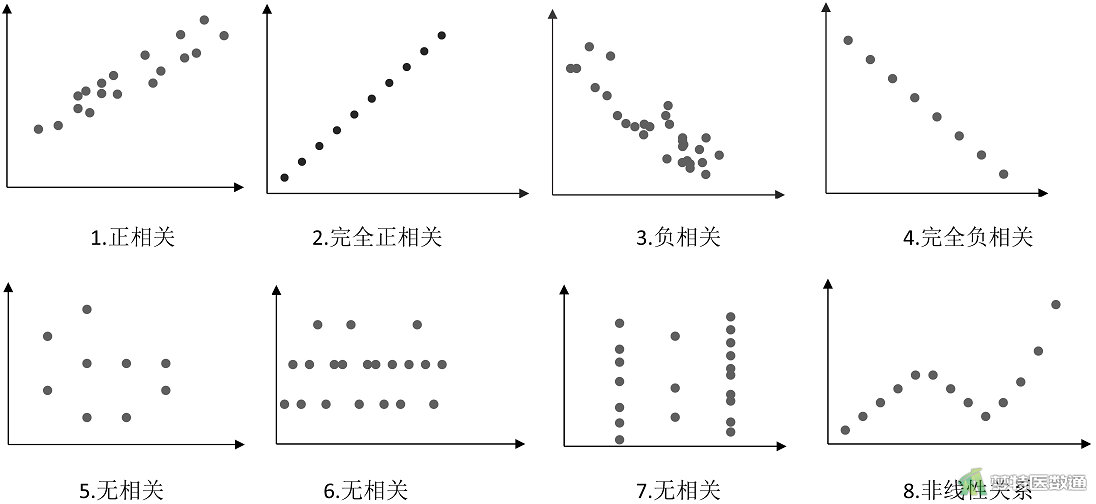
# 1. MATLAB相关性分析的基础
相关性分析是一种统计技术,用于衡量两个或多个变量之间的相互依赖性。在MATLAB中,相关性分析函数提供了强大的工具,可以量化和可视化变量之间的关系。
MATLAB相关性分析的基础包括以下概念:
- **相关系数:**相关系数是一个介于-1到1之间的数字,表示两个变量之间的线性关系。正相关系数表示变量一起变化,负相关系数表示变量相反变化。
- **相关性检验:**相关性检验确定相关系数是否具有统计学意义。MATLAB提供各种检验,例如皮尔逊相关性检验和斯皮尔曼秩相关性检验。
- **相关性可视化:**相关性矩阵和热图等可视化技术用于展示变量之间的相关性模式。这些可视化可以帮助识别变量之间的强相关性和潜在的模式。
# 2. MATLAB相关性分析在生物信息学中的应用
### 2.1 基因表达分析
基因表达分析是生物信息学中的一项重要任务,旨在了解不同条件下基因表达的差异。MATLAB相关性分析在基因表达分析中发挥着至关重要的作用。
#### 2.1.1 差异基因表达分析
差异基因表达分析旨在识别在不同条件下表达差异显著的基因。MATLAB提供了一系列工具,可以帮助研究人员进行差异基因表达分析。
```matlab
% 导入基因表达数据
data = importdata('gene_expression.csv');
% 标准化数据
data = normalize(data, 'zscore');
% 计算差异基因
diff_genes = find(abs(data(:,1) - data(:,2)) > 0.5);
% 可视化差异基因
heatmap(data(:, diff_genes));
```
**代码逻辑分析:**
* `importdata` 函数导入基因表达数据。
* `normalize` 函数对数据进行标准化,以消除不同基因之间的表达差异。
* `find` 函数识别差异基因,阈值为 0.5。
* `heatmap` 函数可视化差异基因的表达模式。
#### 2.1.2 聚类分析
聚类分析是一种无监督学习技术,用于将具有相似表达模式的基因分组。MATLAB提供了几种聚类算法,包括层次聚类和 k 均值聚类。
```matlab
% 聚类基因
clusters = clusterdata(data, 5, 'linkage', 'average');
% 可视化聚类结果
dendrogram(clusters);
```
**代码逻辑分析:**
* `clusterdata` 函数使用平均连锁聚类算法将基因聚类为 5 个簇。
* `dendrogram` 函数可视化聚类结果,显示基因之间的层次关系。
### 2.2 蛋白质组学分析
蛋白质组学分析研究蛋白质的表达、相互作用和功能。MATLAB相关性分析在蛋白质组学分析中具有广泛的应用。
#### 2.2.1 蛋白质-蛋白质相互作用网络
蛋白质-蛋白质相互作用网络揭示了蛋白质之间的相互作用,对于了解细胞过程至关重要。MATLAB提供了一系列工具,可以帮助研究人员构建和分析蛋白质-蛋白质相互作用网络。
```matlab
% 导入蛋白质-蛋白质相互作用数据
ppi_data = importdata('ppi.csv');
% 构建网络
G = graph(ppi_data(:,1), ppi_data(:,2));
% 计算网络属性
centrality = centrality(G, 'eigenvector');
% 可视化网络
plot(G, 'NodeLabel', centrality);
```
**代码逻辑分析:**
* `importdata` 函数导入蛋白质-蛋白质相互作用数据。
* `graph` 函数构建蛋白质-蛋白质相互作用网络。
* `centrality` 函数计算网络中节点的中心性,衡量节点的重要性。
* `plot` 函数可视化网络,并根据中心性着色节点。
#### 2.2.2 蛋白质功能注释
蛋白质功能注释将蛋白质与已知的功能关联起来。MATLAB提供了一系列工具,可以帮助研究人员执行蛋白质功能注释。
```matlab
% 导入蛋白质序列
sequences = fastaread('proteins.fasta');
% 执行 BLAST 搜索
blast_results = blast('sequences', 'database');
% 注释蛋白质
annotations = parse_blast_results(blast_results);
```
**代码逻辑分析:**
* `fastaread` 函数导入蛋白质序列。
* `blast` 函数执行 BLAST 搜索,将序列与已知数据库进行比较。
* `parse_blast_results` 函数解析 BLAST 结果,并提取蛋白质注释。
### 2.3 代谢组学分析
代谢组学分析研究代谢物和代谢途径。MATLAB相关性分析在代谢组学分析中具有广泛的应用。
#### 2.3.1 代谢途径分析
代谢途径分析旨在识别和可视化代谢物之间的相互作用。MATLAB提供了一系列工具,可以帮助研究人员执行代谢途径分析。
```matlab
% 导入代谢物数据
metabolites = importdata('metabolites.csv');
% 构建代谢途径图
pathway = draw_metabolic_pathway(metabolites);
% 可视化代谢途径图
plot(pathway);
```
**代码逻辑分析:**
* `importdata` 函数导入代谢物数据。
* `draw_metabolic_pathway` 函数构建代谢途径图,显示代谢物之间的相互作用。
* `plot` 函数可视化代谢途径图。
#### 2.3.2 生物标志物发现
生物标志物发现旨在识别可以区分不同疾病状态的代谢物。MATLAB相关性分析在生物标志物发现中具有重要作用。
```matlab
% 导入代谢物数据和疾病标签
data = importdata('metabolites_and_labels.csv');
% 计算代谢物之间的相关性
corr_matrix = corr(data(:,1:end-1));
% 识别与疾病标签相关性显著的代谢物
significant_metabolites = find(abs(corr_matrix(:,end)) > 0.5);
% 可视化相关性矩阵
heatmap(corr_matrix);
```
0
0





