图神经网络在药物发现中的应用:加速研发,提升药物有效性
发布时间: 2024-08-22 09:46:02 阅读量: 62 订阅数: 34 

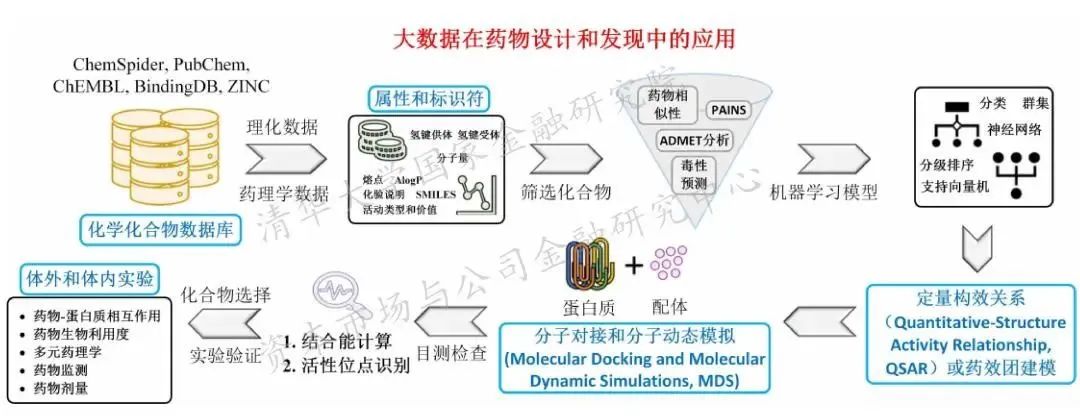
# 1. 图神经网络简介
图神经网络(GNN)是一种用于处理图结构数据的深度学习模型。与传统的深度学习模型不同,GNN 可以直接在图结构上进行操作,提取图中节点和边的特征信息,从而实现对图结构数据的建模和分析。
GNN 的基本原理是将图中的节点和边表示为向量,并通过消息传递机制在节点和边之间传递信息。通过多次的消息传递,GNN 可以聚合图中节点和边的特征信息,并更新节点的表示。这种机制使得 GNN 能够捕获图结构中的局部和全局信息,并实现对图结构数据的有效建模。
# 2. 图神经网络在药物发现中的理论基础
### 2.1 图神经网络的原理和架构
**2.1.1 图神经网络的数学基础**
图神经网络(GNN)是一种基于图结构数据的深度学习模型。它将分子表示为图,其中节点代表原子,边代表原子之间的键。GNN通过在图上执行消息传递操作来学习分子的特征表示。
消息传递操作的数学基础是图卷积运算。图卷积运算将每个节点的特征与相邻节点的特征聚合,并更新节点的特征表示。这个过程可以重复进行多层,从而学习到更高级别的分子特征。
**2.1.2 常见的图神经网络模型**
* **图卷积网络(GCN):**GCN是最基本的GNN模型,它使用图卷积运算来更新节点的特征。
* **门控图卷积网络(Gated GCN):**Gated GCN在GCN的基础上增加了门控机制,可以更好地控制信息流。
* **图注意力网络(GAT):**GAT使用注意力机制来赋予不同相邻节点不同的权重,从而学习到更重要的特征。
* **消息传递神经网络(MPNN):**MPNN是一种更通用的GNN模型,它允许消息传递操作具有任意数量的步骤。
### 2.2 图神经网络在药物发现中的优势
**2.2.1 对分子结构的建模能力**
GNN可以有效地对分子结构进行建模,因为它可以捕捉分子中原子之间的拓扑关系和化学键信息。这使得GNN能够学习到分子的结构特征,如分子骨架、官能团和立体构型。
**2.2.2 预测分子性质和活性**
GNN可以预测分子的各种性质和活性,如溶解度、毒性、生物活性等。通过学习分子的结构特征,GNN可以建立分子性质和活性之间的关系,从而进行预测。
**代码示例:**
```python
import torch
from torch_geometric.nn import GCNConv
# 定义分子图
graph = torch.nn.Data(x=torch.rand(num_nodes, num_features),
edge_index=torch.tensor([[0, 1], [1, 2]]))
# 定义图卷积网络
model = GCNConv(num_features, num_classes)
# 前向传播
out = model(graph.x, graph.edge_index)
```
**代码逻辑分析:**
* `GCNConv`类定义了一个图卷积层,它使用图卷积运算来更新节点的特征。
* `num_features`和`num_classes`分别表示输入特征的维度和输出类的数量。
* `graph.x`和`graph.edge_index`分别表示分子图的节点特征和边索引。
* `model(graph.x, graph.edge_index)`执行前向传播,计算更新后的节点特征。
# 3.1 药物靶点识别
**3.1.1 靶点预测算法**
图神经网络在药物靶点识别中发挥着至关重要的作用。通过构建分子和靶蛋白之间的交互图,图神经网络可以预测潜在的靶点。常用的靶点预测算法包括:
- **图卷积网络(GCN)**:GCN通过在图上执行卷积操作来学习分子和靶蛋白之间的关系。它可以捕获分子结构和靶蛋白序列的局部特征,并预测分子与靶蛋白之间的相互作用。
```python
import dgl
import torch
# 构建分子和靶蛋白之间的交互图
graph = dgl.graph((src_nodes, dst_nodes), num_nodes=num_nodes)
# 定义图卷积网络模型
model = GCN(input_dim=node_features.shape[1], hidden_dim=hidden_dim, output_dim=num_classes)
# 训练模型
optimizer = torch.optim.Adam(model.parameters())
for epoch in range(num_epochs):
logits = model(graph, node_features)
loss = F.cross_entropy(logits, labels)
optimizer.zero_grad()
loss.backward()
optimizer.step()
# 预测分子与靶蛋白之间的相互作用
predictions = model(graph, node_features).argmax(dim=1)
```
- **图注意力网络(GAT)**:GAT通过在图上执行注意力机制来学习分子和靶蛋白之间的重要交互。它可以识别分子结构和靶蛋白序列中与靶点识别最相关的特征。
```python
import dgl
import torch
# 构建分子和靶蛋白之间的交互图
graph = dgl.graph((src_nodes, dst_node
```
0
0





