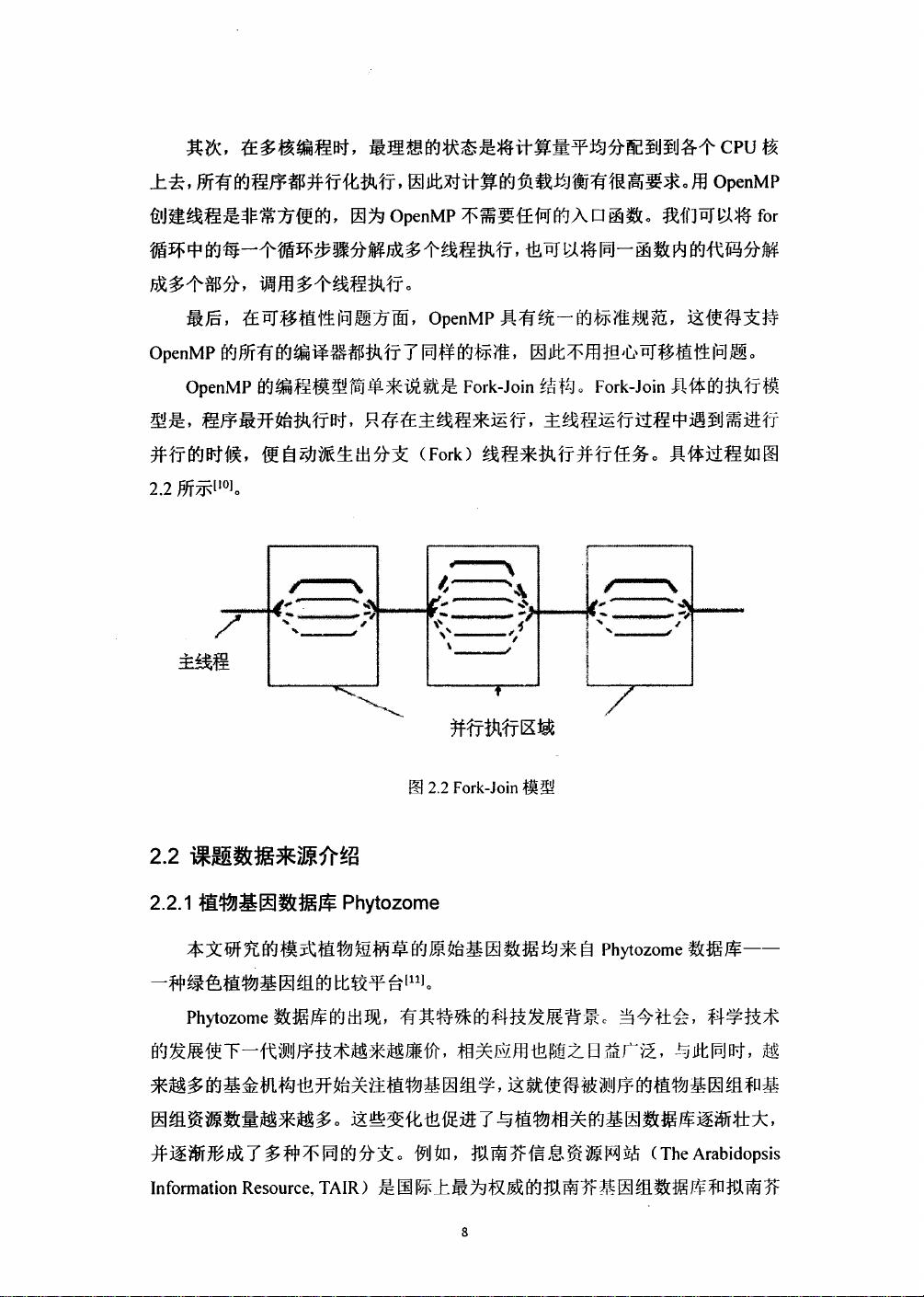
并行聚类算法:模体识别与短柄草启动子预测的进展
版权申诉
93 浏览量
更新于2024-07-02
收藏 10.35MB PDF 举报
本文档深入探讨了"计算机研究 -模体的并行聚类算法研究及在短柄草核心启动子预测的应用.pdf"的主题,着重于在现代生物信息学背景下,随着基因测序技术的飞速发展,大规模基因组数据的产生和分析方法的进步。作者指出,为了解析不同基因组中保守序列的功能,特别是转录调控过程中的关键环节,即转录因子(Transcription Factors, TFs)对特定DNA序列(称为TF结合位点, Transcription Factor Binding Sites, TFBSs)的识别和作用,研究模体(Motif)变得尤为重要。
模体是一组被同一转录因子识别的TFBS的共同特征的抽象表示。随着大量新发现的潜在模体的积累,后续任务通常涉及将这些模体聚类到相应的转录因子家族,以便于分类和功能理解。这要求开发出新颖且高效的聚类算法,以合并具有相似性质的同一TF家族的模体。
论文的核心内容可能包括以下几点:
1. 背景与挑战:阐述了当前生物信息学领域面临的挑战,如海量基因组数据的处理需求,以及如何通过并行计算技术加速模体识别和聚类过程。
2. 并行聚类算法设计:介绍了一种新的并行聚类算法,该算法可能是基于分布式计算框架(如MapReduce、Spark或GPU)来提高模体聚类的效率,减少时间和资源消耗。
3. 算法原理:详细解释了算法的工作原理,可能包括模体特征向量的构建,相似性度量方法,以及如何在多核处理器或分布式系统中划分和同步任务。
4. 性能评估:可能讨论了新算法在实际数据集上的表现,比如在短柄草(一种模式生物,常用于基因调控研究)核心启动子预测中的准确性和效率对比。
5. 应用实例:论文可能提供了具体的实例,展示如何使用该算法成功地将不同的模体分组,并揭示了对转录因子功能的理解是如何增强的。
6. 未来方向:最后,论文可能对未来的研究方向提出展望,如如何进一步优化算法,或者将并行聚类与其他生物信息学工具集成,以提升整体的基因调控分析能力。
这篇论文在计算机科学与生物学交叉领域,为解决模体聚类问题提供了一种创新的并行计算解决方案,这对于理解和解读大规模基因组数据中的转录调控机制具有重要意义。
2022-06-24 上传
2022-06-24 上传
2022-06-26 上传
2022-06-25 上传
2019-07-22 上传
2019-07-22 上传