德塔自然语言图灵系统与DNA计算 - 第5修订版
需积分: 0 174 浏览量
更新于2024-06-30
收藏 14.01MB PDF 举报
"DNA元基催化与肽计算_第5修订版本V00051021"
本书详细探讨了多种计算机科学与信息技术在自然语言处理、数据分析、人工智能及数据库管理等多个领域的应用。以下是各章节的主要知识点:
1. **第一章_德塔自然语言图灵系统**
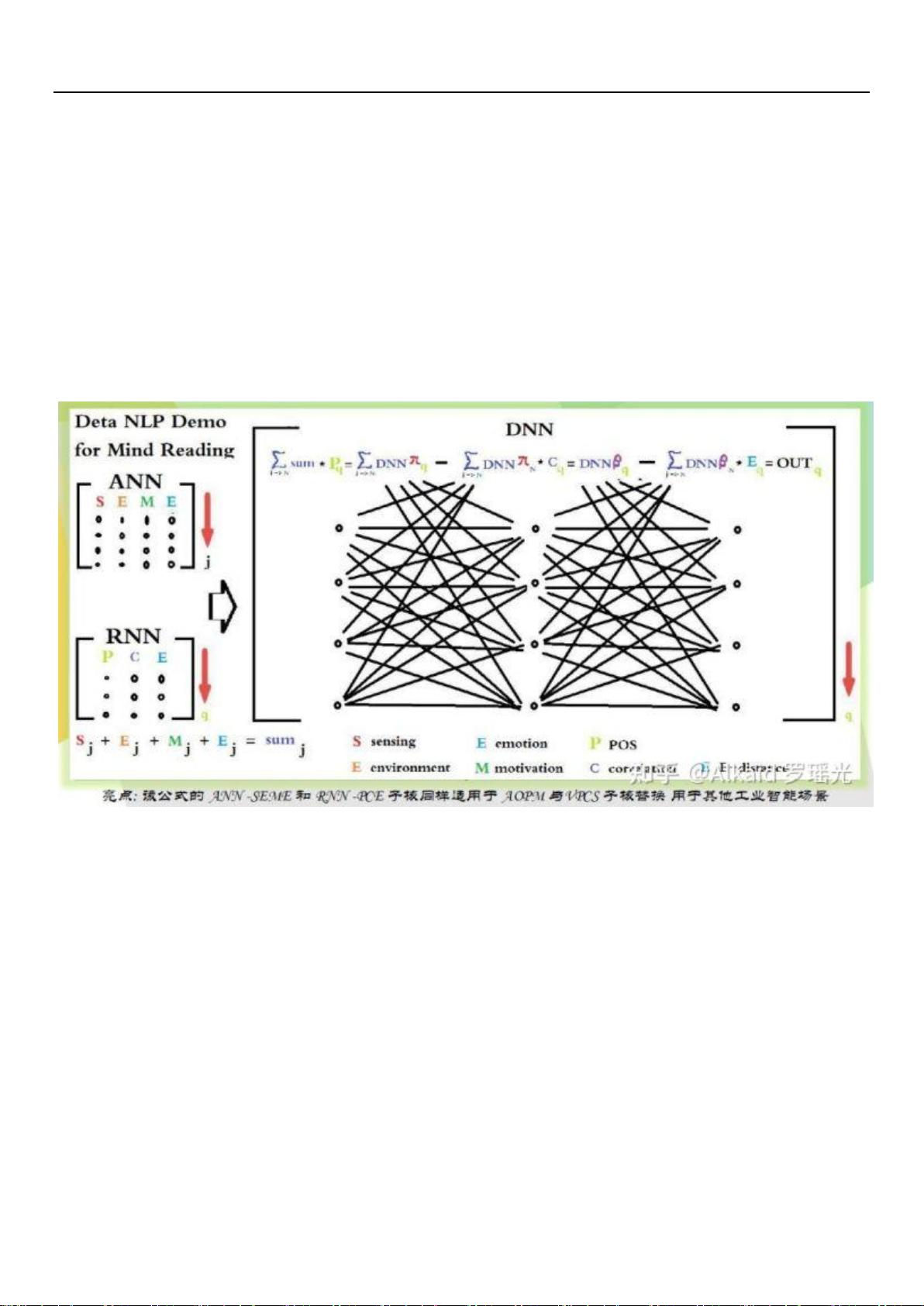
- **德塔分词的催化切词优化方式**:本章介绍了德塔算法在分词上的优化技术,通过催化切词提高分词效率和准确性。
- **分词**:讨论了如何将文本分割成有意义的词语,这是自然语言处理的基础。
- **排序**:在分词后的词汇进行排序,有助于后续处理和索引。
- **神经网络索引**:利用神经网络建立高效的文本索引,提升查询速度。
- **动态POS函数流水阀门细化遍历内核匹配**:介绍了一种动态标注词汇部分-of-speech(POS)的方法,用于更精细化的语言理解。
2. **第二章Java数据分析算法引擎系统**
- **微分催化排序**:提出了一种新的排序算法,结合微分计算和催化原理。
- **计算力与算能优化的思想手稿**:探讨如何提高计算效率和计算能力。
- **线性与非线性**:涵盖了线性和非线性数据分析方法。
- **EtlUnicorn对音频卷积处理**:利用EtlUnicorn工具处理音频数据,可能涉及音频信号的卷积操作。
- **排序与搜索**:在数据处理中,排序和搜索是基本操作。
3. **第三章德塔ETL人工智能可视化数据流分析引擎系统**
- **可视化界面**:讨论了用户友好的数据流分析界面设计。
- **流存储与节点**:介绍流式数据处理和节点在网络中的作用。
- **插件与档案**:讨论了扩展系统功能的插件和数据管理。
- **拓扑与神经网络**:探讨了数据流的拓扑结构和神经网络的应用。
4. **第四章德塔Socket流可编程数据库语言引擎系统**
- **SocketrestTCP握手协议**:涉及网络通信中的Socket、REST和TCP协议交互。
- **文件数据库**:讨论了文件为基础的数据库系统。
- **VPCS服务器与调度架构**:介绍虚拟化控制平台服务器及其调度策略。
- **PLSQL语言与编译机**:讲解PL/SQL这种数据库编程语言及其编译机制。
- **PLORM语言**:可能是一种针对对象关系映射的数据库语言。
- **灾后重建**:讨论了数据库系统的恢复和灾难应对策略。
5. **第五章_德塔数据结构变量快速转换**
- **内存结构、数据结构与类结构**:深入到计算机内存管理和数据组织。
- **转换加速**:探讨如何优化数据转换过程以提高速度。
- **不规则对象的变换**:处理非标准或复杂数据结构的转换问题。
- **场景变换**:可能是指在特定上下文下的数据变换。
6. **第六章_数据预测引擎系统**
- **坐标系统预测**:预测模型在地理空间数据中的应用。
- **环境预测**:利用数据预测环境变化或影响。
- **雷达机与状态机**:介绍了预测模型的构建和监控工具。
- **离散模型预测、概率机、向量机**:涵盖多种预测模型和算法,如离散事件模拟、概率模型和支持向量机。
- **商旅TSP**:旅行商问题(TSP)在商业路线规划中的应用。
- **眼睛团坐标识别**:可能涉及图像处理中的眼部特征识别。
7. **第七章类人DNA与神经元基于催化算子映射编码方式**
- **类人认知模拟**:讨论了模拟人类认知过程的算法和系统。
- **DETAhumanoidcognitionhistory, development, application**:涵盖了该领域的发展历程、技术实现和实际应用。
这些章节的内容展示了信息技术在处理自然语言、数据处理、人工智能和数据库管理等领域的深度应用和创新。每章都包含了丰富的理论和技术细节,适合对相关领域有深入研究的读者。
2022-08-04 上传
2022-08-04 上传
2022-08-04 上传
2023-06-12 上传
2023-06-07 上传
2023-06-06 上传
2023-06-12 上传
2023-04-05 上传
2023-07-17 上传
ask_ai_app
- 粉丝: 24
- 资源: 326
最新资源
- SSM Java项目:StudentInfo 数据管理与可视化分析
- pyedgar:Python库简化EDGAR数据交互与文档下载
- Node.js环境下wfdb文件解码与实时数据处理
- phpcms v2.2企业级网站管理系统发布
- 美团饿了么优惠券推广工具-uniapp源码
- 基于红外传感器的会议室实时占用率测量系统
- DenseNet-201预训练模型:图像分类的深度学习工具箱
- Java实现和弦移调工具:Transposer-java
- phpMyFAQ 2.5.1 Beta多国语言版:技术项目源码共享平台
- Python自动化源码实现便捷自动下单功能
- Android天气预报应用:查看多城市详细天气信息
- PHPTML类:简化HTML页面创建的PHP开源工具
- Biovec在蛋白质分析中的应用:预测、结构和可视化
- EfficientNet-b0深度学习工具箱模型在MATLAB中的应用
- 2024年河北省技能大赛数字化设计开发样题解析
- 笔记本USB加湿器:便携式设计解决方案