LwM2M技术规范v1.0.2官方文档
需积分: 9 121 浏览量
更新于2024-07-19
收藏 3.15MB PDF 举报
"OMA-TS-LightweightM2M-V1_0_2-20180209-A.pdf" 是一份由OpenMobile Alliance (OMA)发布的技术规范文档,详细介绍了LightweightM2M (LwM2M) 协议的版本1.0.2。LwM2M是一种物联网(IoT)通信标准,旨在为资源受限的设备提供高效、轻量级的机器到机器(M2M)通信。
LwM2M协议设计的核心目标是为了简化物联网设备的管理和服务,特别是针对那些计算能力有限、电池寿命要求高或者网络带宽有限的设备。它采用了简洁的协议栈和数据表示方式,以降低设备的复杂性和功耗,同时保证了可靠的数据传输和远程管理功能。
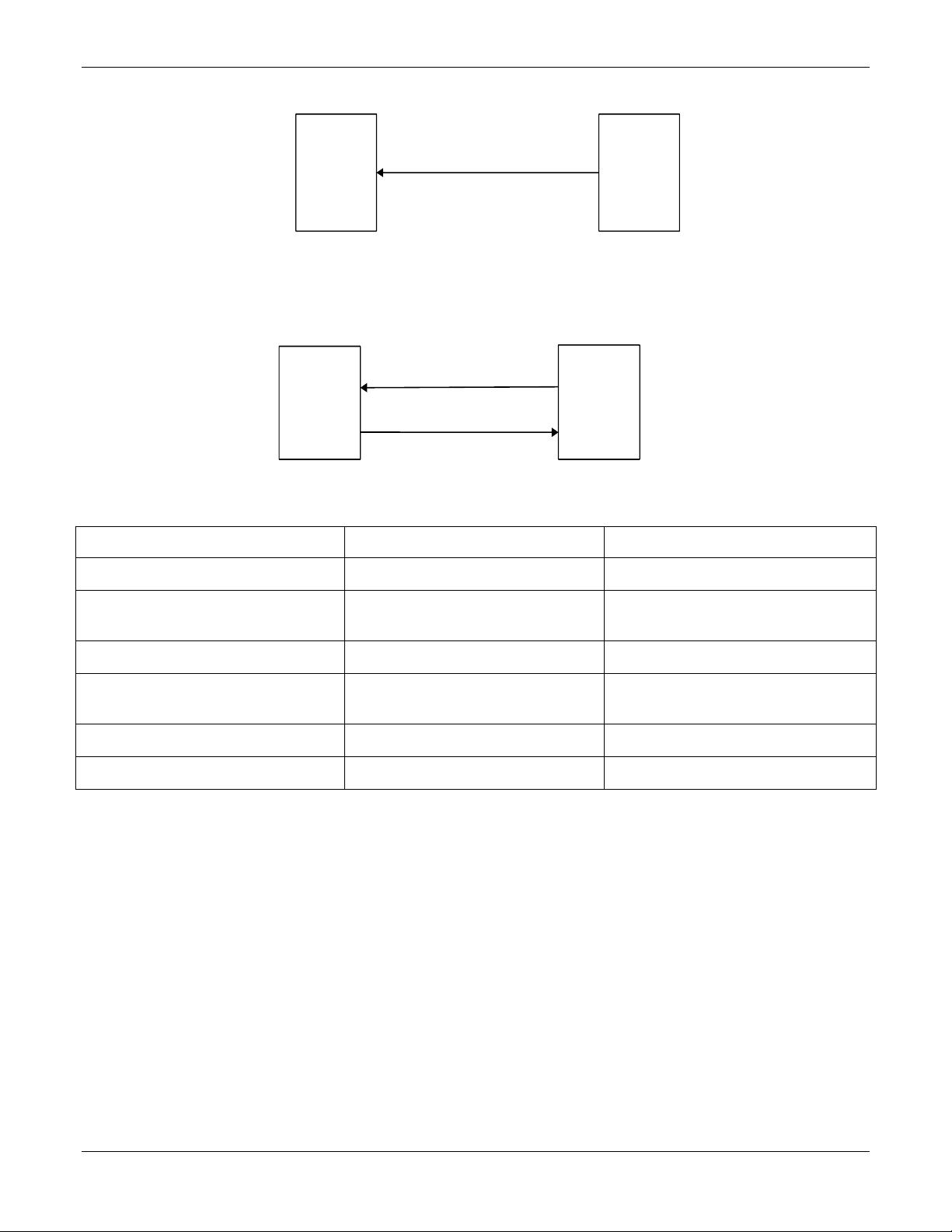
在LwM2M协议中,主要有三个关键组件:LwM2M客户端、LwM2M服务器和LwM2M中间件。LwM2M客户端安装在物联网设备上,负责与服务器进行通信,执行数据收集、设备管理以及应用执行等功能。LwM2M服务器则负责接收客户端发送的数据,处理请求,执行远程配置或固件更新等操作。LwM2M中间件是连接客户端和服务器的桥梁,它处理协议转换,使得不同平台和协议的设备可以互相通信。
协议采用了层次化的数据模型,称为对象、资源和实例。对象代表特定的功能集,如设备管理、传感器数据等;资源是对象内的具体数据点或功能,如温度读数、开关状态;实例则是特定对象的多个实体,每个实例可能有不同的数据值。
LwM2M支持多种传输层协议,包括CoAP(Constrained Application Protocol)和TCP,以适应不同的网络环境。CoAP是专门为物联网设计的一种低开销、基于UDP的协议,而TCP则提供了更可靠的连接性,适用于需要高稳定性的场景。
此外,LwM2M还定义了安全机制,确保数据传输的安全性,包括使用DTLS(Datagram Transport Layer Security)协议进行加密和身份验证,以防止未授权访问和数据篡改。
LwM2M协议通过提供一套标准化的通信框架,降低了物联网设备开发的复杂性,提升了部署效率,同时也保证了系统的可扩展性和互操作性。这份文档详细描述了LwM2M的各个方面,包括协议结构、操作模式、数据模型、安全特性以及实现指南,对于开发者和物联网解决方案设计者来说具有极高的参考价值。
2009-12-23 上传
2023-06-01 上传
2023-06-01 上传
2023-06-01 上传
2023-06-01 上传
2023-06-01 上传
2023-06-06 上传
2023-06-01 上传
tlfnewcsdn
- 粉丝: 0
- 资源: 5
最新资源
- Java集合ArrayList实现字符串管理及效果展示
- 实现2D3D相机拾取射线的关键技术
- LiveLy-公寓管理门户:创新体验与技术实现
- 易语言打造的快捷禁止程序运行小工具
- Microgateway核心:实现配置和插件的主端口转发
- 掌握Java基本操作:增删查改入门代码详解
- Apache Tomcat 7.0.109 Windows版下载指南
- Qt实现文件系统浏览器界面设计与功能开发
- ReactJS新手实验:搭建与运行教程
- 探索生成艺术:几个月创意Processing实验
- Django框架下Cisco IOx平台实战开发案例源码解析
- 在Linux环境下配置Java版VTK开发环境
- 29街网上城市公司网站系统v1.0:企业建站全面解决方案
- WordPress CMB2插件的Suggest字段类型使用教程
- TCP协议实现的Java桌面聊天客户端应用
- ANR-WatchDog: 检测Android应用无响应并报告异常