NAMD与VMD安装及在Windows7中的使用教程
需积分: 39 127 浏览量
更新于2024-07-22
1
收藏 1.13MB PPT 举报
"NAMD是一个高性能的分子动力学模拟软件,由美国伊利诺伊大学开发,主要用于研究生物大分子如蛋白质、核酸和脂类的动态行为。NAMD利用经验力场和数值方法求解原子级别的运动方程。CHARMM力场是NAMD常用的力场,包含top和par两种文件类型。另一方面,VMD是一款强大的可视化工具,拥有图形用户界面,能够进行模型构建、分子可视化和数据分析。在Windows 7系统中,NAMD和VMD的安装需要下载预编译的版本或安装程序,并调整系统环境变量以确保软件可被系统识别。在使用VMD时,可以通过命令行调用,改变路径并指定工作目录,例如通过cmd命令行来运行。VMD和NAMD都需要配置文件,如.pdb文件存储原子坐标,.psf文件存储结构信息,力场参数文件定义了分子间相互作用,而.conf文件则包含了模拟的具体参数设定,如温度、压力、系综选择等。"
NAMD全称为Northwestern Molecular Dynamics,是一个专门为生物大分子设计的并行分子动力学模拟软件。它采用牛顿运动定律,结合经验力场(如CHARMM力场),通过数值方法解决多体问题,模拟分子系统的动力学行为。CHARMM力场是一种广泛使用的力场,包括topology(top)文件和parameter(par)文件,分别描述分子的拓扑结构和物理参数,如原子间距离、角度和扭转角的限制,以及电荷和Lennard-Jones参数。
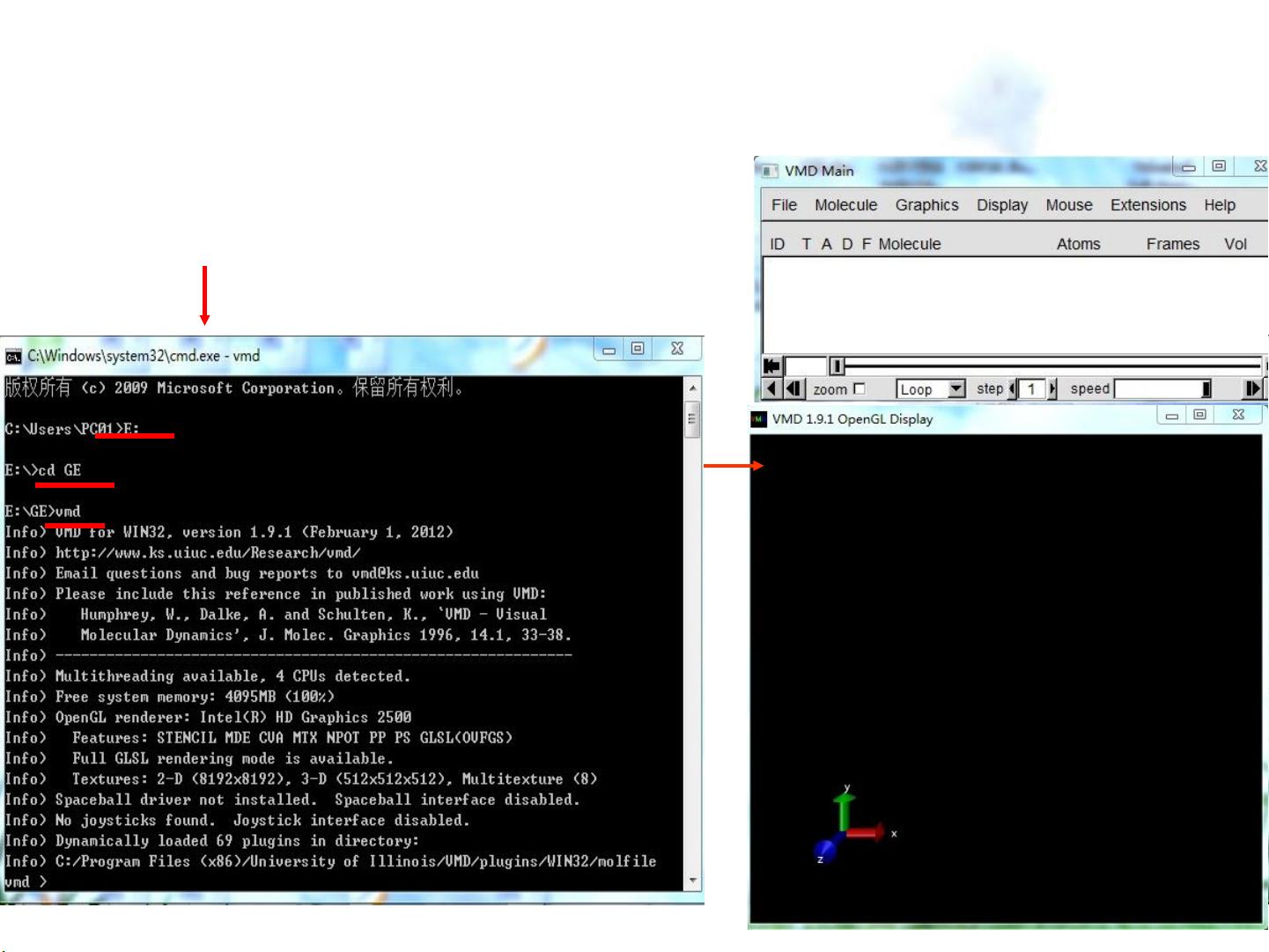
VMD,全称Visual Molecular Dynamics,是一款强大的分子可视化软件,具有丰富的图形界面,支持实时三维分子结构显示、动画制作、分子建模以及数据分析。在Windows系统中,VMD的安装过程类似于常规软件,下载安装文件后进行标准安装流程。安装完成后,需要对环境变量进行设置,确保系统能够在任何路径下找到VMD执行文件。
在实际操作中,用户通常需要在命令行环境中调用VMD,例如在Windows的命令提示符下,通过改变当前目录(cd命令)定位到含有VMD所需文件的目录,然后直接输入软件名称启动VMD。在进行分子动力学模拟前,还需要准备一系列输入文件,如:
1. .pdb文件:这是Protein Data Bank格式的文件,记录了分子中原子的坐标信息。
2. .psf文件:Particle Mesh Ewald Structure Factor文件,包含了分子的拓扑信息,如化学键长、键角、二面角以及电荷和非键相互作用参数。
3. 力场参数文件:提供了模拟中使用的力场参数,这些参数决定了分子间的相互作用力计算方式。
4. 配置文件 (.conf):定义了模拟的详细参数,如初始结构文件(.pdb和.psf)、温度和压力控制器、统计力学系综、边界条件以及输出结果的存储方式等。
通过合理的参数配置和准确的力场,NAMD和VMD相结合,可以为科研人员提供深入理解生物大分子的结构、动力学性质以及它们在生理过程中的功能提供强有力的研究工具。
2013-10-05 上传
点击了解资源详情
点击了解资源详情
点击了解资源详情
suny77
- 粉丝: 0
- 资源: 1
最新资源
- 完整版读文本文件(一行行的加入到列表框).rar
- 简单优雅个人主页模板
- SpiderWebServer:EPITA 2023 Spider Web服务器
- 可爱动物图标下载
- Illumyno:向Autodesk Dynamo添加一些基本的RADIANCE功能
- shakhes-crx插件
- kmeans-ISODATA algotithm:k-means 和 ISODATA 的聚类算法。-matlab开发
- A09
- 跨父母育儿:跨境育儿示范网站
- 完整版读文本文件(一行行的加入到列表框).e.rar
- 3d水晶动物图标下载
- GDevelopCoursework2:CSC3224计算机游戏开发课程2
- INI 文件读取实用程序:通过提供应用程序和密钥字符串读取 INI 文件条目。-matlab开发
- 2019-2020中国品牌海外传播报告-蓝标-202012精品报告2020.rar
- 可爱qq熊图标下载
- Tutorials:我的一些教程的代码