muma R包:代谢组学分析教程与实例
需积分: 23 153 浏览量
更新于2024-07-19
收藏 1.82MB PDF 举报
muma是针对代谢组学数据分析的R语言包,特别设计用于处理和解读大规模代谢数据集。这个补充材料详细介绍了如何利用muma进行一系列的分析步骤,包括但不限于主成分分析(PCA)、单变量分析、多变量分析(如部分 least squares discriminant analysis, PLS-DA 和 orthogonal projection to latent structures-discriminant analysis, OPLS-DA)以及NMR谱解析工具,如统计总相关谱(STOCSY)、1D STOCSY、正交信号校正(OSC)和比率分析核磁共振谱学(RANSY)。
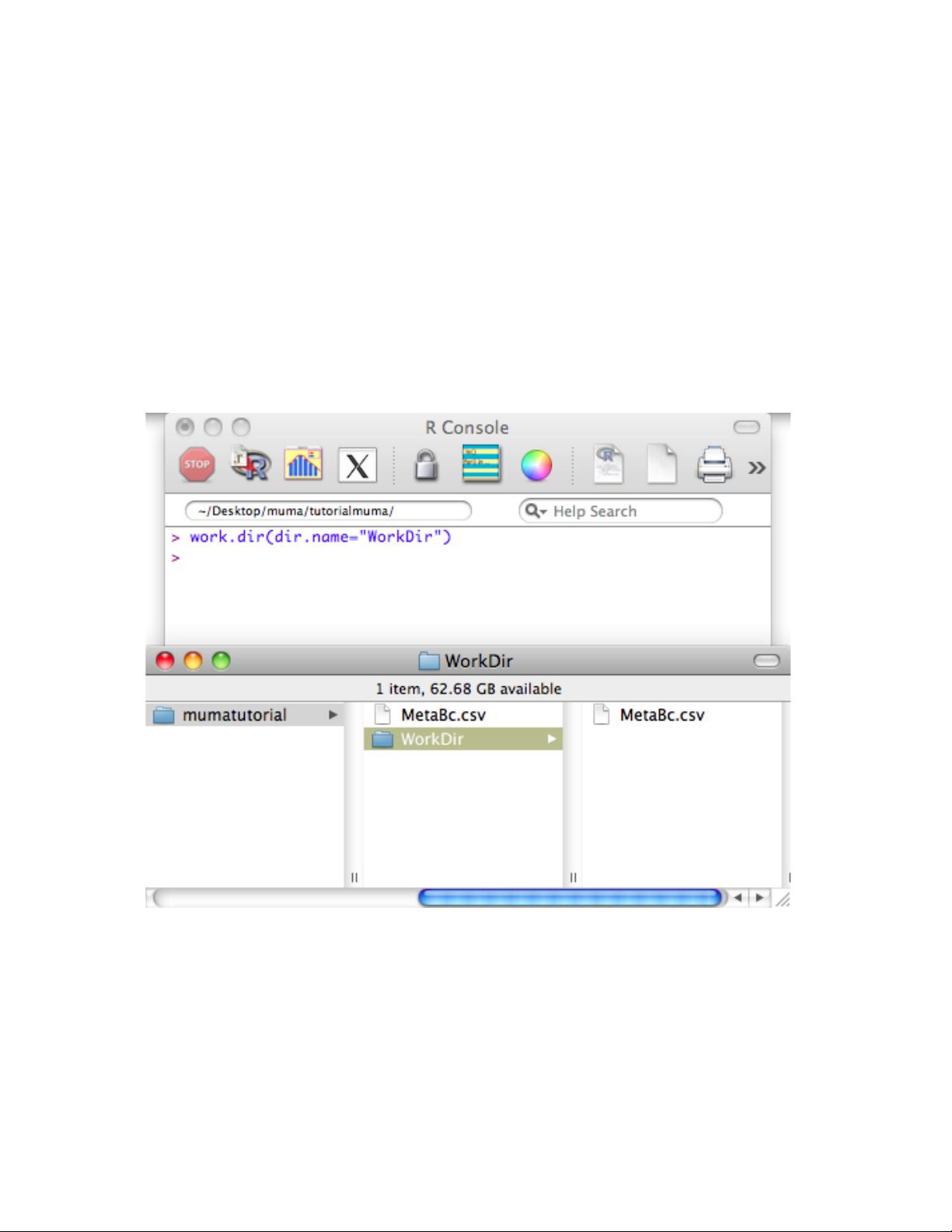
在使用muma之前,首先需要下载并安装该包。包内提供的功能列表详细列出了所有可用于数据预处理、探索性分析和解释性分析的函数,确保了用户能够对复杂的数据集进行全面的处理。例如,分析流程开始于创建工作目录,然后进行数据加载和格式验证,接着执行PCA来揭示数据的主要变异模式和样本间的关系。
在单变量分析部分,用户可以使用muma进行显著性检验,识别出与特定变量或生理状态关联显著的代谢物。合并单变量和多变量信息有助于建立更全面的分析视角,通过比较不同分析方法的结果,挖掘隐藏的模式和潜在的生物标志物。
PLS-DA和OPLS-DA是muma中的核心功能,它们是常用于分类和区分不同组别或疾病状态的有力工具。这两种方法结合了变量选择和数据降维的优势,有助于提高模型的预测能力和解释性。
NMR谱解析工具部分提供了对代谢物结构鉴定的支持,如STOCSY用于消除化学位移伪随机性,提高谱峰解析的精度;1D STOCSY则针对简单的一维NMR谱;而OSC-STOCSY结合了OSC和STOCSY,进一步增强了一维谱的分辨能力;RANSY通过比率分析可以帮助识别和定量某些类型的化合物。
整个muma包的设计目标是简化代谢组学数据的处理流程,使得研究人员能够更加高效地进行数据探索、模式识别和结果解读。这份补充材料不仅包含了使用示例,还提供了参考文献,以供深入研究和进一步了解相关的统计原理和技术细节。通过学习和实践这些内容,用户将能够熟练掌握muma,并将其应用到实际的代谢组学研究中。
2015-04-26 上传
2021-02-24 上传
2023-07-17 上传
2023-07-16 上传
2023-12-05 上传
2023-07-16 上传
2024-09-15 上传
2024-09-15 上传
2024-09-15 上传
weixin_41817681
- 粉丝: 0
- 资源: 2
最新资源
- C++多态实现机制详解:虚函数与早期绑定
- Java多线程与异常处理详解
- 校园导游系统:无向图实现最短路径探索
- SQL2005彻底删除指南:避免重装失败
- GTD时间管理法:提升效率与组织生活的关键
- Python进制转换全攻略:从10进制到16进制
- 商丘物流业区位优势探究:发展战略与机遇
- C语言实训:简单计算器程序设计
- Oracle SQL命令大全:用户管理、权限操作与查询
- Struts2配置详解与示例
- C#编程规范与最佳实践
- C语言面试常见问题解析
- 超声波测距技术详解:电路与程序设计
- 反激开关电源设计:UC3844与TL431优化稳压
- Cisco路由器配置全攻略
- SQLServer 2005 CTE递归教程:创建员工层级结构