德塔自然语言图灵系统与DNA计算
需积分: 0 95 浏览量
更新于2024-06-30
收藏 15.05MB PDF 举报
"DNA元基催化与肽计算_第5修订版本V00055121"
本文档详细介绍了多个与计算机科学、人工智能和生物计算相关的知识点,特别是将生物学概念应用于信息技术的创新方法。以下是各章节的主要内容:
1. **第一章_德塔自然语言图灵系统**:
- **知识来源**:这部分可能讨论了自然语言处理(NLP)领域中的理论基础。
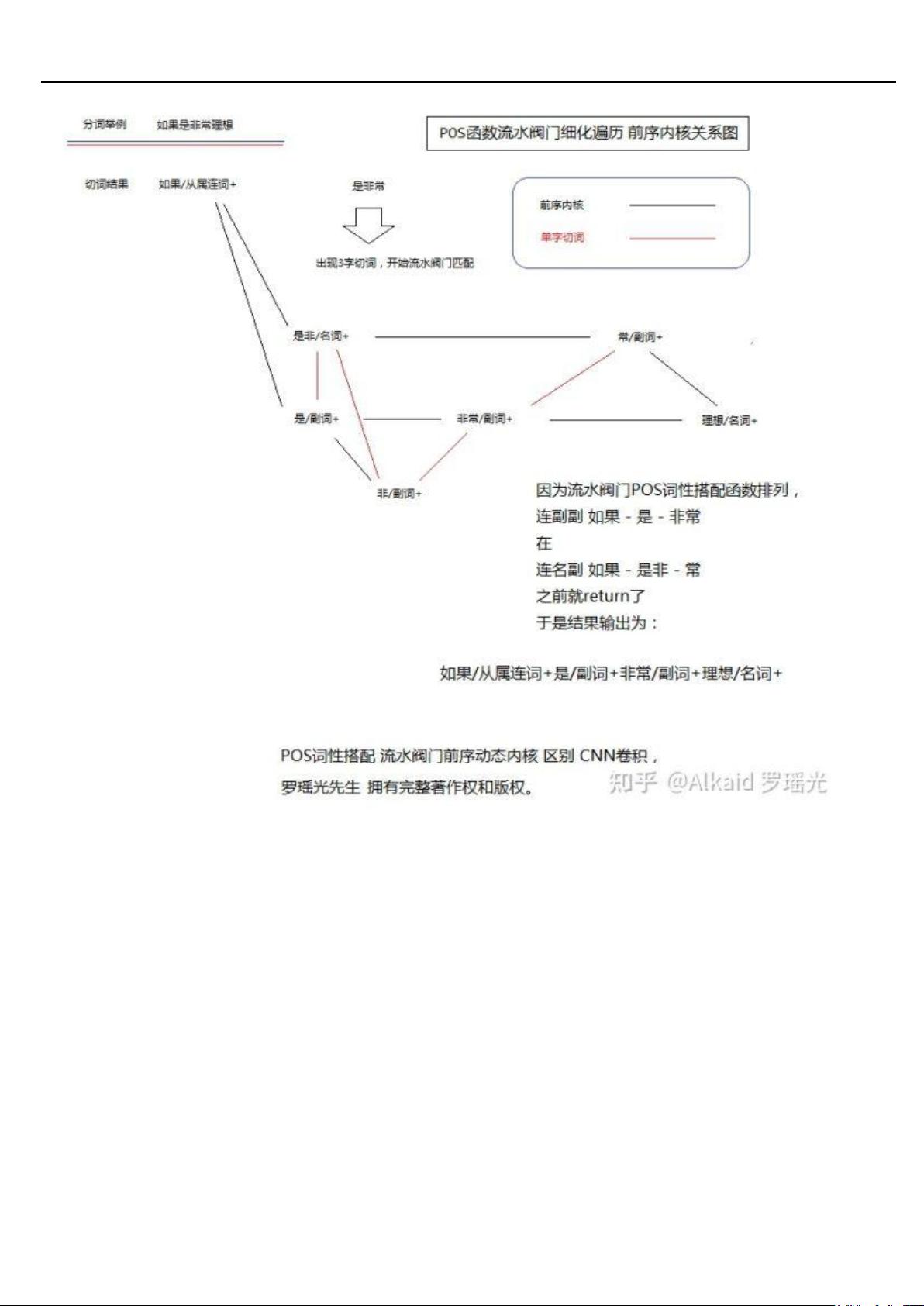
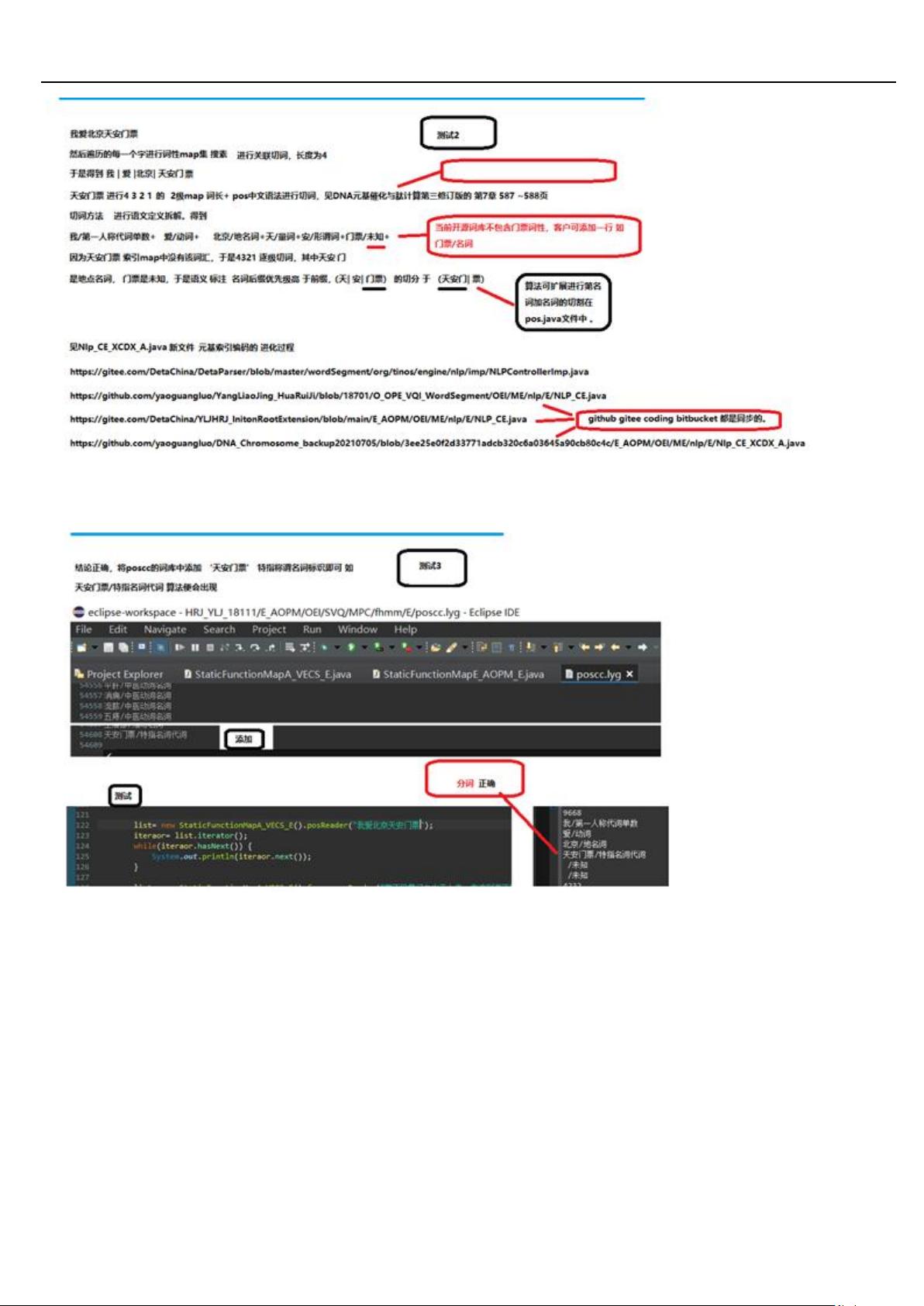
- **德塔分词的催化切词优化方式**:介绍了一种提高分词效率的方法,分词是NLP中的基础步骤,用于将连续的文本切分成有意义的词汇单元。
- **分词**:阐述了分词技术及其在文本处理中的重要性。
- **排序**:可能讨论了如何对分词结果进行排序,以提高搜索或分析性能。
- **神经网络索引**:介绍了利用神经网络构建高效索引的策略。
- **分词在线性文本搜索中的应用**:探讨了分词技术如何提升线性文本搜索的速度和准确性。
- **动态POS**:可能涉及动态Part-of-Speech(词性标注),用于识别词汇在句子中的功能。
2. **第二章Java数据分析算法引擎系统**:
- **微分催化排序**:可能是一种基于微积分原理的排序算法,用于优化数据处理。
- **计算力与算能优化的思想手稿**:讨论了提高计算效率的策略。
- **线性与非线性**:可能涵盖了线性和非线性数据分析方法。
- **音频卷积处理**:介绍了处理音频数据的卷积技术。
- **仿生听觉与视觉**:讨论了模拟生物听觉和视觉系统来改善数据分析的方法。
- **排序与搜索**:深入研究了不同类型的排序和搜索算法。
3. **第三章德塔ETL人工智能可视化数据流分析引擎系统**:
- **界面与皮肤**:设计和自定义用户界面的元素。
- **流存储**:涉及实时数据流处理和存储。
- **节点与插件**:解释了如何通过可扩展的节点和插件系统增强分析引擎的功能。
- **档案与拓扑**:管理数据文件和分析流程的拓扑结构。
- **神经网络**:可能提到了用于数据分析的神经网络模型。
4. **第四章德塔Socket流可编程数据库语言引擎系统**:
- **SocketrestTCP握手协议**:讨论了网络通信协议,如TCP/IP和RESTful API。
- **文件数据库**:涉及基于文件的数据库管理系统。
- **VPCS服务器与调度架构**:介绍了服务器平台和调度策略。
- **PLSQL语言**:讲解了结构化查询语言的编程方面。
- **PLORM语言**:可能是数据库操作和对象关系映射的语言。
5. **第五章_德塔数据结构变量快速转换**:
- **内存的结构**:解析了计算机内存的组织和管理。
- **数据的结构**:涵盖了不同的数据结构(如数组、链表、树等)。
- **类的结构**:讨论面向对象编程中的类和对象。
- **转换加速**:讲述了优化数据转换的方法。
- **不规则对象的变换**:处理非标准或复杂数据对象的方法。
- **场景变换**:可能涉及到虚拟现实或图形渲染中的场景转换。
- **计算的模式变换**:可能包括了算法改进和计算模式的优化。
6. **第六章_数据预测引擎系统**:
- **坐标系统预测**:使用坐标系统进行预测分析。
- **环境预测**:可能涵盖了环境因素对预测的影响。
- **雷达机与状态机**:两种预测模型或算法。
- **离散模型预测**:处理离散数据的预测方法。
- **概率机**:基于概率理论的预测模型。
- **向量机**:支持向量机(SVM)在预测分析中的应用。
- **商旅TSP**:旅行商问题(Traveling Salesman Problem)的解决方案,一种经典的组合优化问题。
- **眼睛团坐标识别**:可能涉及图像处理和人脸识别技术。
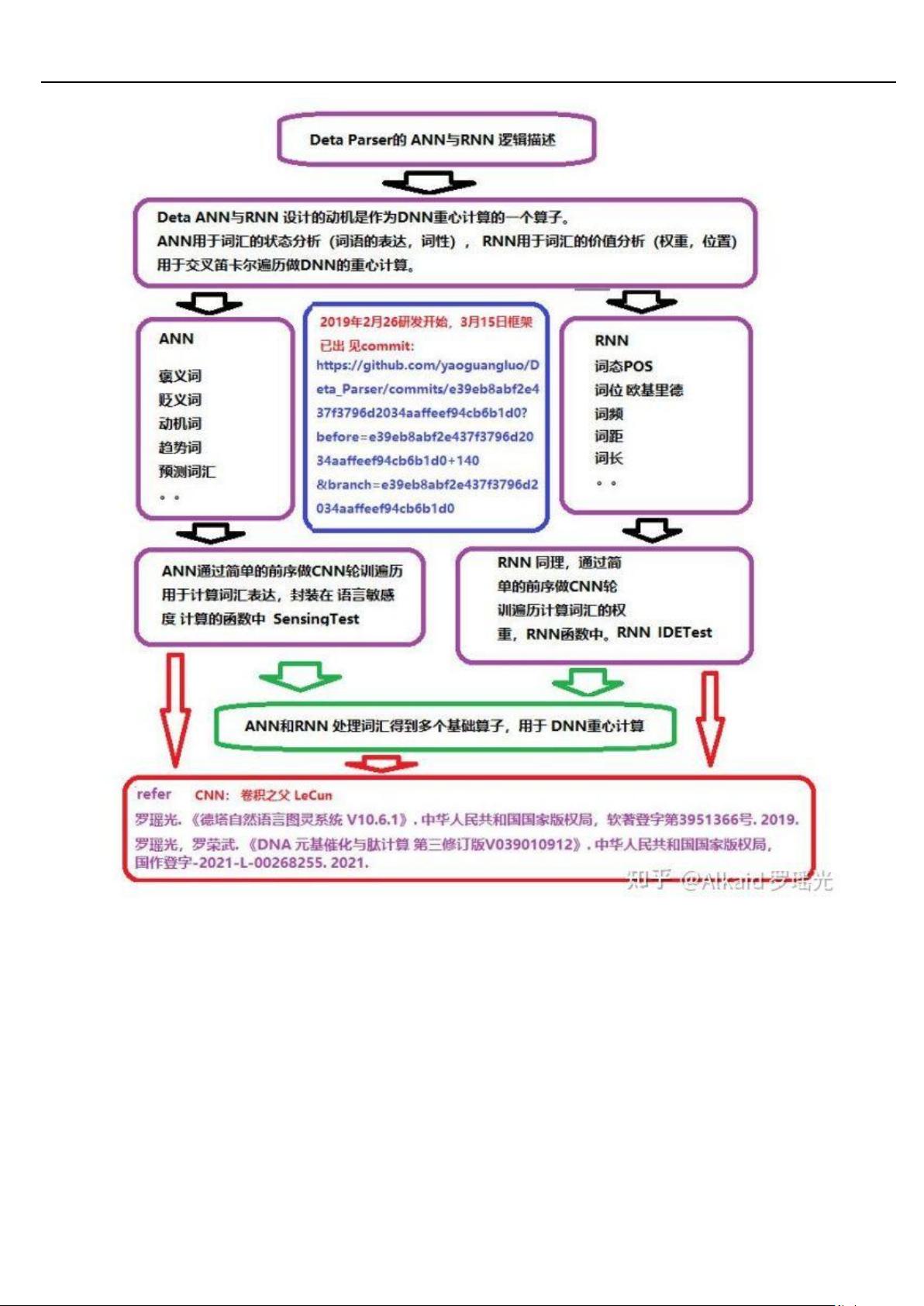
7. **第七章类人DNA与神经元基于催化算子映射编码方式**:
- **DETAhumanoidcognition**:探讨了类人认知的模拟,可能涉及生物启发式的人工智能。
- **DETAhumanoidcognitionhistory**:回顾了该领域的历史发展。
这份文档涵盖了广泛的主题,从自然语言处理、数据排序和索引到生物启发的计算模型,展示了多学科交叉的深度研究。
2022-08-04 上传
2022-08-04 上传
2022-08-04 上传
2023-06-12 上传
2023-06-07 上传
2023-06-06 上传
2023-06-12 上传
2023-04-05 上传
2023-07-17 上传
Crazyanti
- 粉丝: 26
- 资源: 302
最新资源
- 磁性吸附笔筒设计创新,行业文档精选
- Java Swing实现的俄罗斯方块游戏代码分享
- 骨折生长的二维与三维模型比较分析
- 水彩花卉与羽毛无缝背景矢量素材
- 设计一种高效的袋料分离装置
- 探索4.20图包.zip的奥秘
- RabbitMQ 3.7.x延时消息交换插件安装与操作指南
- 解决NLTK下载停用词失败的问题
- 多系统平台的并行处理技术研究
- Jekyll项目实战:网页设计作业的入门练习
- discord.js v13按钮分页包实现教程与应用
- SpringBoot与Uniapp结合开发短视频APP实战教程
- Tensorflow学习笔记深度解析:人工智能实践指南
- 无服务器部署管理器:防止错误部署AWS帐户
- 医疗图标矢量素材合集:扁平风格16图标(PNG/EPS/PSD)
- 人工智能基础课程汇报PPT模板下载