RNA-seq入门详解:从样本收集到数据分析流程
需积分: 10 24 浏览量
更新于2024-07-22
1
收藏 3.5MB PDF 举报
RNA-seq (全称为RNA测序)是一种高通量的技术,用于研究基因表达、转录组学和非编码RNA等分子水平的信息。本文提供了一个全面的入门介绍,涵盖了RNA-seq的基本步骤和实验设计的关键要素。
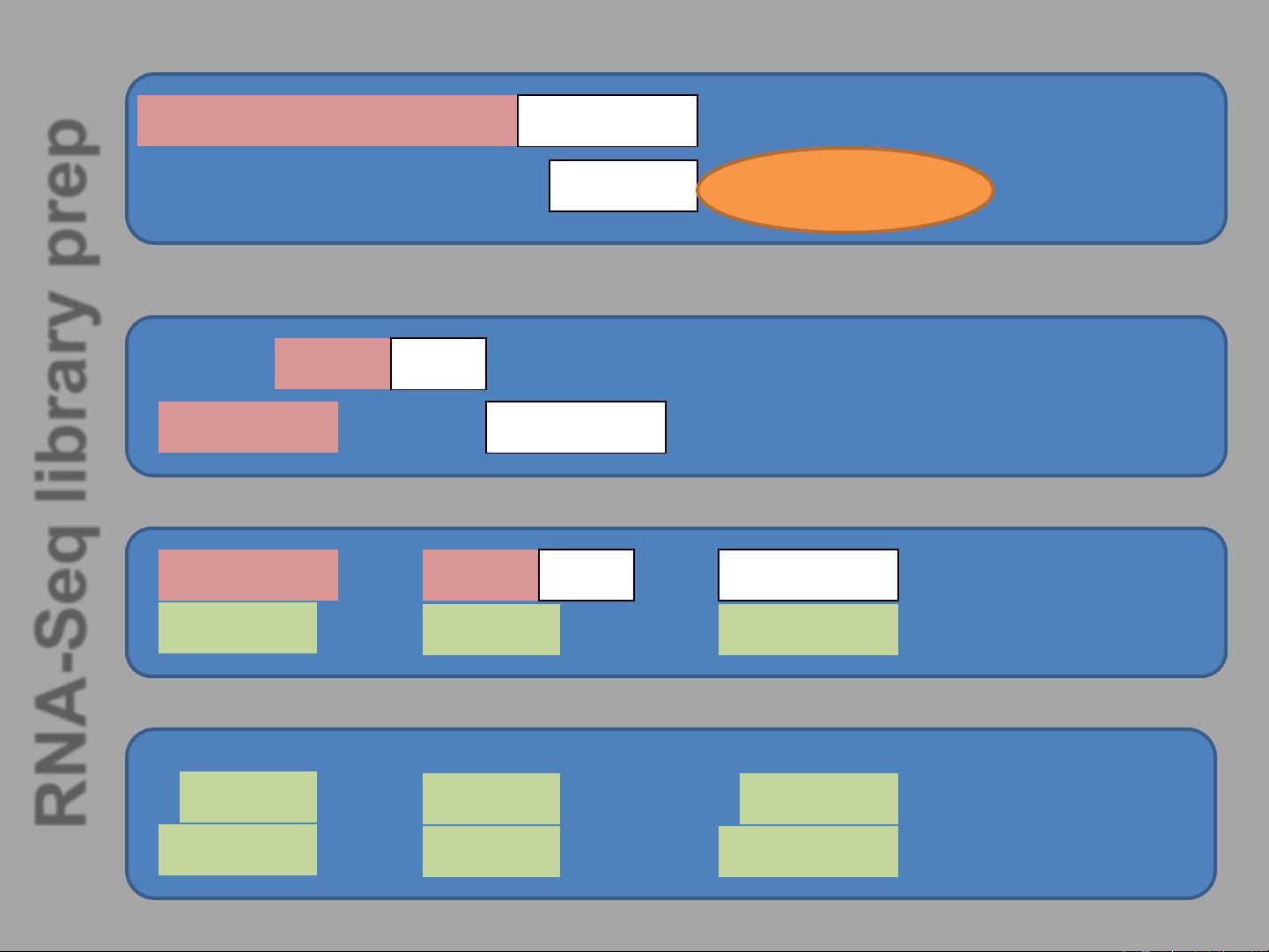
1. **组织采集与RNA提取**:
RNA-seq的第一步是选择合适的组织样本,如植物(例如P. m. var. menziesii 和 P. m. var. glauca)在特定环境下的生长样本。样本的选择要考虑生物钟的影响,确保时间点的选择能够反映生物体的正常生理状态。采集后,RNA的提取至关重要,包括使用磁性珠子分离出总RNA,以及进一步纯化mRNA。
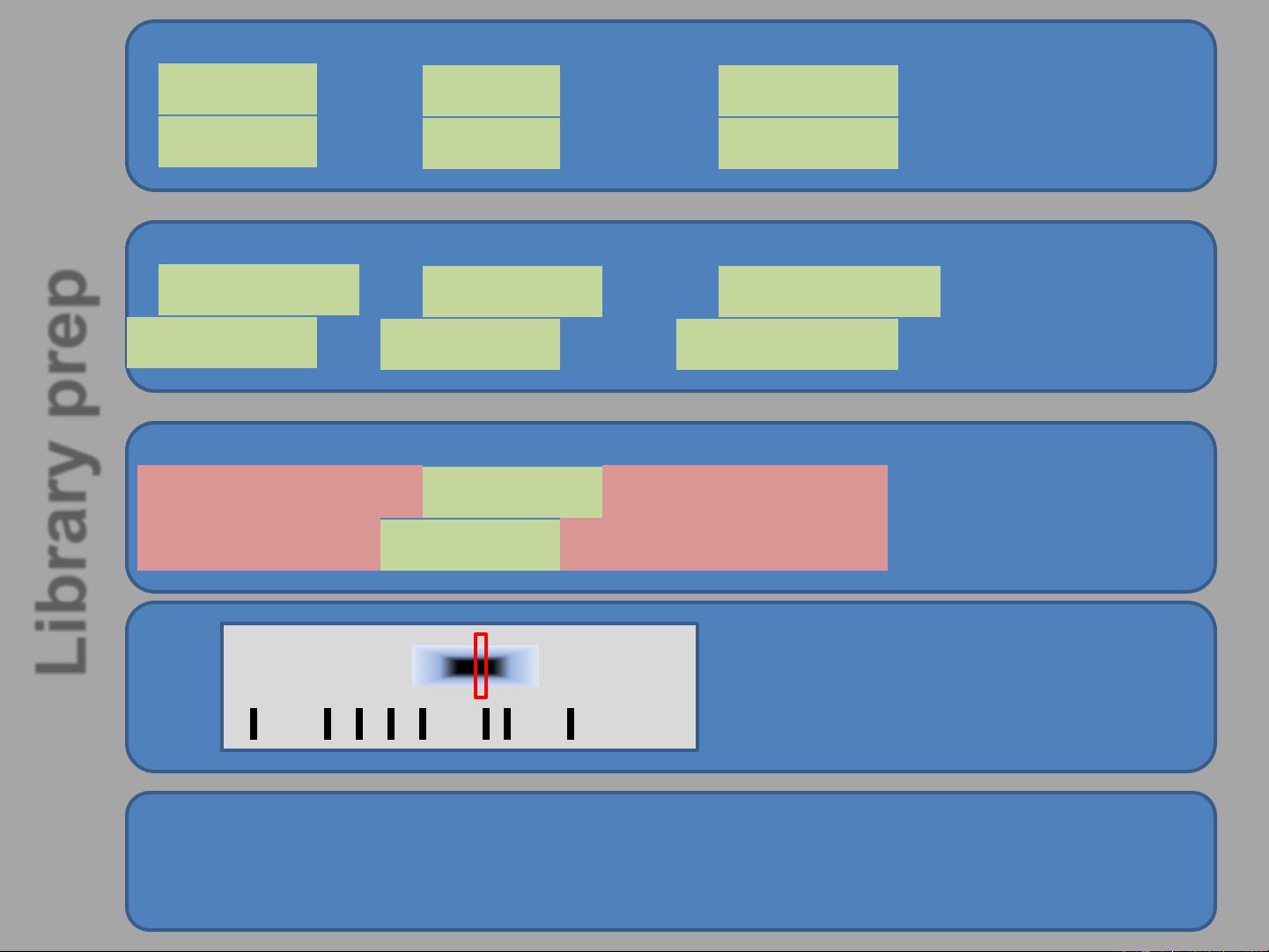
2. **图书馆准备**:
mRNA的处理通常包括去除可能干扰分析的非编码RNA部分,然后通过逆转录过程将mRNA转化为第一链cDNA。这个过程中,随机引物被用于引导合成,形成带有随机序列的DNA片段。接着进行第二链合成,生成双链DNA。整个过程涉及到碱基修复,消除末端的不规则序列,并添加适配器,以便后续的测序。
3. **Illumina测序**:
序列准备完成后,通常使用Illumina平台进行高通量测序,产生大量读取(reads)。在这个例子中,给出了几个样本的测序数据量,展示了每个样本的测序深度,这对于统计分析和基因表达研究至关重要。
4. **读取映射**:
测序数据经过碱基序列比对,将原始读取(reads)映射到参考基因组上,以便识别哪些基因区域被转录。这一步骤有助于了解转录本的多样性,包括剪接事件和异质性。
5. **统计假设检验**:
读取数量可以转换为表达值,并通过统计方法进行分析,比如t-test或差异表达分析,来检测在不同条件或组织间是否存在显著的基因表达变化。实验设计时需要考虑样本间的重复性,这里提到的每个样本由六个生长在同一环境下的种子苗组成,确保了结果的可靠性和可重复性。
6. **实验设计与注意事项**:
一个有效的实验设计应控制潜在的混杂因素,如样本来源、生长条件等。这里强调了样本间的均一性,每个样本都是来自共同环境的六株幼苗,以减少误差并提高结果的生物学意义。
RNA-seq是一种强大的工具,它不仅揭示了基因表达的定量信息,还提供了关于转录后修饰和调控的洞察。从样本收集到数据分析,每一个步骤都需要严谨的操作和细致的规划,以确保得到准确和有代表性的结果。对于想要进入这一领域的研究人员来说,理解这些关键步骤及其背后的原理至关重要。
258 浏览量
194 浏览量
164 浏览量
497 浏览量
488 浏览量
114 浏览量
185 浏览量
308 浏览量
Xiaofei_iseq
- 粉丝: 1
- 资源: 2
最新资源
- laravel-simple-order-system
- VulkanSharp:Vulkan API的开源.NET绑定
- 网络游戏-网络中的帧传送方法以及节点、帧传送程序.zip
- bc19-webapp
- bagging算法
- c语言课程设计-职工资源管理系统
- 类似WINDOWS进度复制文件夹例子-易语言
- CPSC471-Project
- uzkoogle
- CBEmotionView(iPhone源代码)
- crunchyroll-ext
- 2016年数学建模国赛优秀论文.zip
- 运输成本估算器:允许用户估算物品的运输成本
- Unrar调用模块 - RAR解压、测试、查看全功能版-易语言
- 鸿蒙轮播图banner.7z
- Mailican-crx插件