宏基因组分析流程与关键步骤详解
需积分: 0 185 浏览量
更新于2024-07-01
收藏 2.8MB PDF 举报
"这篇文章是关于微生物组学中的宏基因组分析流程的总结,涵盖了数据预处理、基于读长分析和组装/拼接分析三个关键步骤。由易生信提供的生信宝典宏基因组研讨会内容,包括了从DNA提取到最终结果解析的整个流程,并提到了多个常用的软件和数据库。"
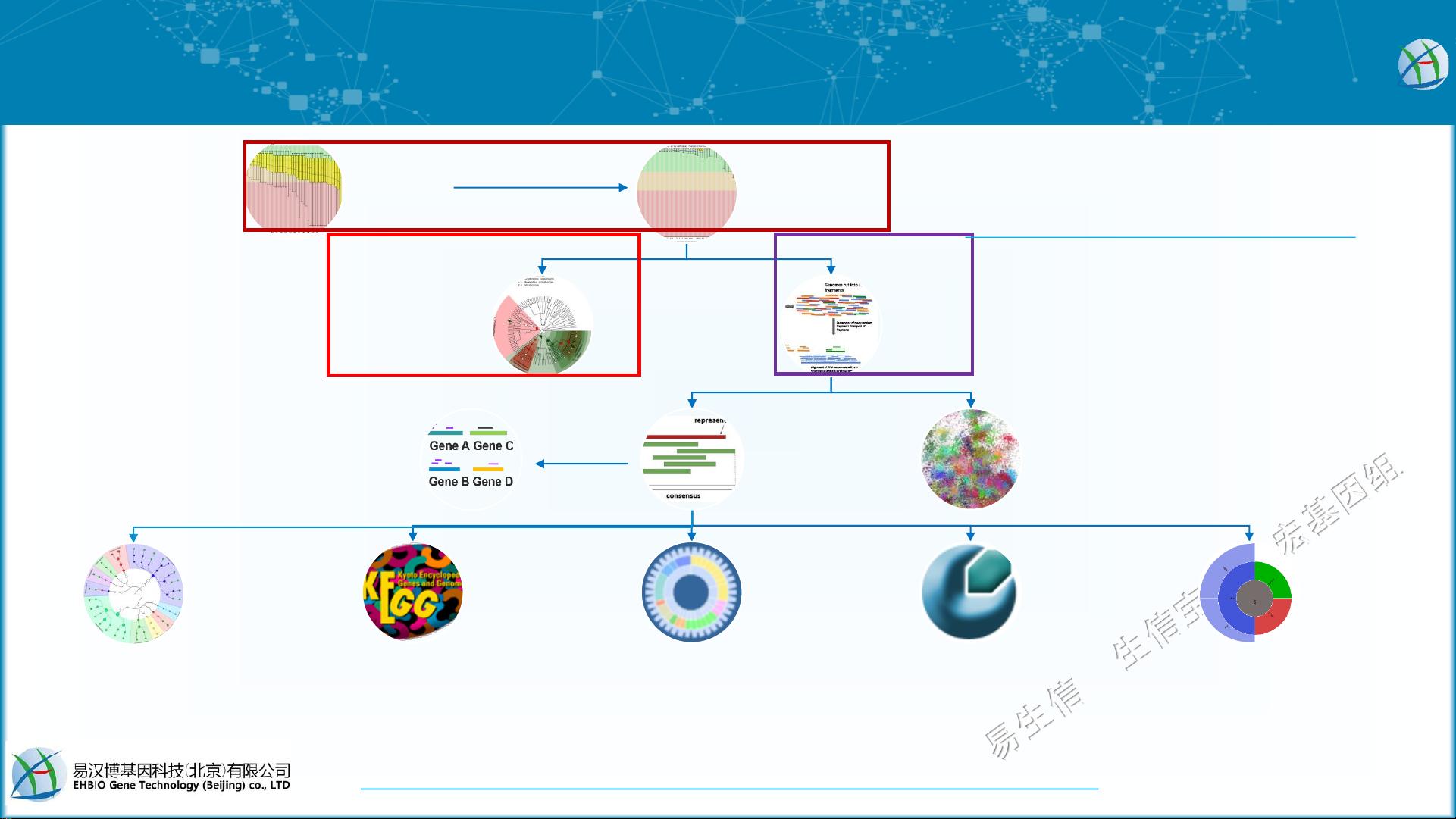
在微生物组研究中,宏基因组分析是探索复杂环境或生物体内微生物群落结构和功能的重要手段。以下是对核心知识点的详细说明:
1. **数据预处理**:
- 在宏基因组分析开始之前,首先进行DNA提取,然后通过随机打断生成适合测序的小片段。
- 测序后的数据需要进行质量控制(QC),去除低质量序列和可能的宿主DNA污染,得到纯净序列。
- 数据预处理还包括比对到参考数据库,如NCBI物种分类数据库,进行物种和功能组成的初步分析。
- 常用的软件有Kraken2,用于快速识别序列的物种来源。
2. **基于读长分析**(Reads-based):
- 这一步通常涉及短读序列比对,通过比对到已知数据库,确定序列的物种归属和功能注释。
- 工具如GhostKOALA用于KEGG基因通路注释,eggNOG-mapper用于eggNOG同源基因簇注释,dbCAN用于鉴定碳水化合物活性基因,RGI用于抗生素抗性基因的识别。
3. **组装/拼接分析**(Assemble-based):
- 长读序列或短读的组合可以用来组装成更大的片段,即重叠群(Contigs)或基因集(Genecatalog)。
- 组装有助于提高基因预测的准确性,尤其是对于那些在数据库中未完全代表的微生物群落。
- 分箱(Binning)技术则用于将组装的基因分配到潜在的物种或菌株中,帮助构建基因组草图。
- 这一步骤通常涉及多种组装工具,如SPAdes、MetaSPAdes等。
4. **基因注释和功能预测**:
- 完成组装后,需要预测基因编码区(CDS),并对其进行功能注释。
- 使用如CAZy数据库来识别碳水化合物代谢相关的酶,CARD用于抗生素抗性基因的研究。
- 基因丰度的计算是理解不同样本间微生物群落差异的关键。
5. **数据分析和可视化**:
- 得到的特征表(物种和功能组成)和统计表需进行统计分析,以揭示微生物群落的多样性和动态变化。
- 通过生成各种图表(如箱形图、热图等)来直观展示结果,帮助研究人员理解和解释数据。
6. **数据库和工具**:
- 文中提到了多个重要的数据库,如NCBI物种分类数据库、KEGG基因通路数据库、eggNOG、CAZy和CARD,它们在宏基因组注释中起着关键作用。
- 同时,也提到了一系列软件工具,如Kraken2、GhostKOALA、eggNOG-mapper、dbCAN和RGI,这些工具在不同阶段的应用是宏基因组分析流程中的核心。
宏基因组分析是一个多步骤的过程,涉及到从实验操作到生物信息学分析的各个环节。理解这个流程以及其中的关键工具和数据库,对于有效解析微生物群落的结构和功能至关重要。
点击了解资源详情
点击了解资源详情
147 浏览量
2021-06-30 上传
2009-02-25 上传
2008-08-21 上传
2021-06-29 上传
2021-08-27 上传
2018-05-31 上传
洋葱庄
- 粉丝: 21
- 资源: 311
最新资源
- B2C_UQ云商系统 v1.3.1
- FrontEnd:回购协议
- StocksEvolutionApp:python实现的应用程序,使用bokeh来显示和操纵股票图
- Javaweb+mybatis+Springboot+SpringMVC活动拼团项目
- 飞机大战初级版
- 新建文件夹,新建文件夹2,matlab
- personal_portfolio:使用HTML,CSS,JS和AOS创建的个人投资组合网站,用于存储个人项目和文件以显示给朋友,家人和未来的雇主
- RoveClone:罗夫克隆
- Registry Finder(注册表管理)2.53 中文绿色版
- AnchorBooks
- AvalonDock的基本用法
- ATM-MACHINE-CODE:带有纯PYTHON的简单后端ATM代码
- 行业文档-设计装置-高压线路检修作业平台.zip
- html5 canvas模拟的见缝插针小游戏源码
- opentelemetry-指标收集和分布式跟踪框架-Rust开发
- WTAB-Wp-Pnl:我在WordPress中创建设置面板的基本插件