三代测序技术:预处理与数据分析流程
版权申诉
"三代测序预处理.pdf"
三代测序,又称单分子实时(Single Molecule, Real-Time,SMRT)测序,是生物信息学领域中的一个重要技术,由Pacific Biosciences公司(PacBio)开发。这项技术以其长读长和对DNA分子的直接观察而著称,为基因组学研究提供了全新的视角。PacBio的三代测序系统主要包括PacBio RSII和Sequel System两个平台,这两个系统都利用了Zero-Mode Waveguides(ZMWs)来捕捉和检测单个DNA分子的荧光信号。
ZMWs是PacBio测序技术的核心组件,它们是一种微小的光学装置,能够限制荧光信号的检测范围,使得只有在ZMW底部的DNA分子片段能被观测到。当DNA聚合酶在模板链上移动并添加荧光标记的核苷酸时,会释放出特定的荧光信号,这些信号被转化为数字数据,进而构建出序列信息。
测序过程中,数据会分为不同的质量等级。P0代表Empty ZMW,意味着没有检测到高质量的读取;P1表示检测到了高质量的读取;P2则表示检测到了信号,但没有得到高质量的读取。这些数据通常以三种不同的文件格式之一呈现:`.h5`、`.bam`或`.fastq`,其中`.h5`是PacBio特有的原始数据格式。
PacBio测序产生的原始数据需要经过预处理,包括去除低质量读段(如P0和P1)、基质校正、纠错等步骤,才能用于后续的分析。其中,Subreads是原始的短读段,而Circular Consensus Sequences (CCS) 是通过多次循环读取同一分子,提高准确性的产物。Chaisson MJ和Tesler G在2012年的文章中介绍了使用Basic Local Alignment with Successive Refinement (BLASR) 算法进行单分子测序读段映射的方法。
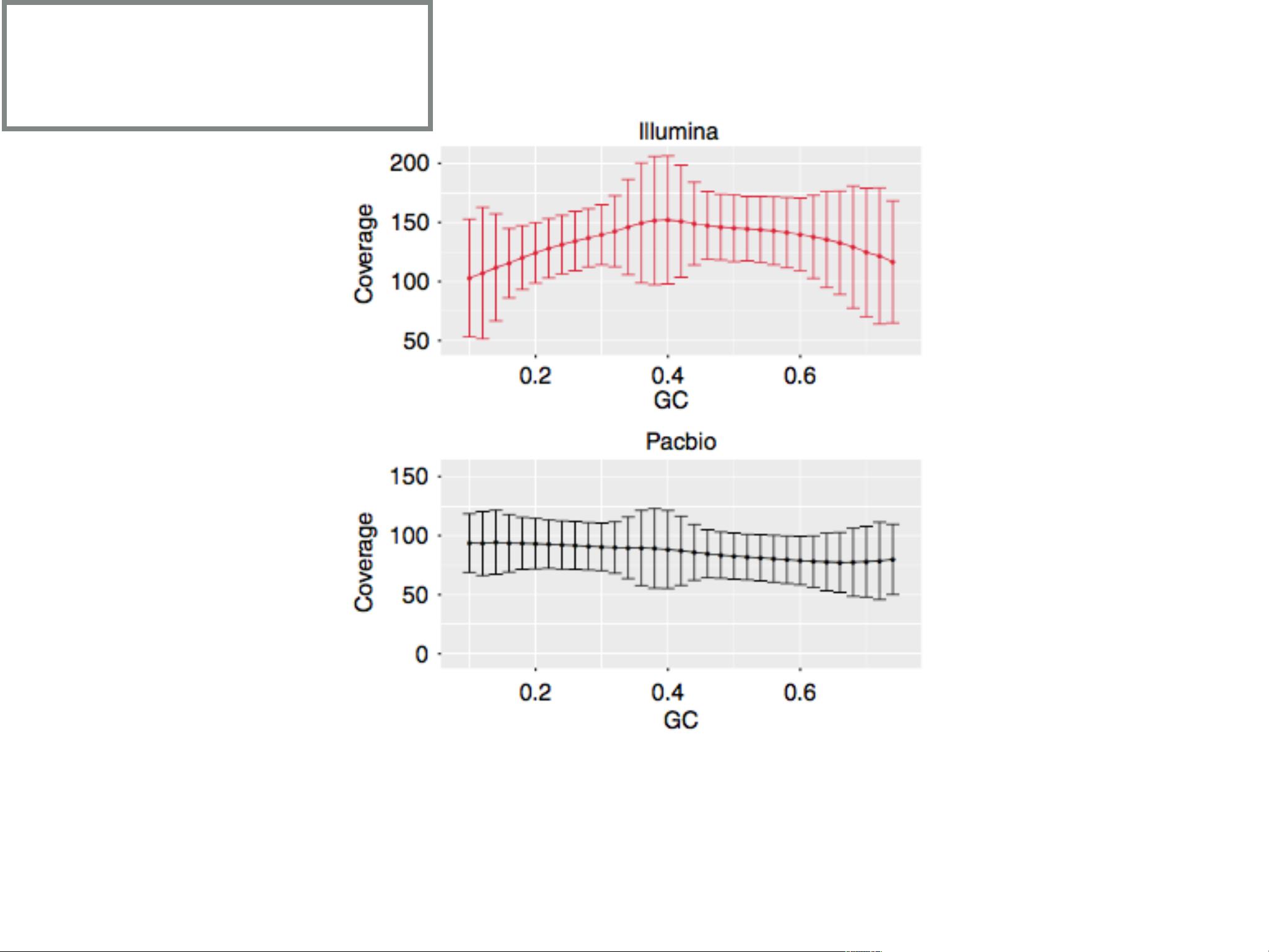
三代测序的一个显著优势是没有明显的GC偏倚,与Illumina的短读测序数据相比,如Shi等人在2016年发表的研究所示。这使得三代测序在处理GC含量变化大的区域时表现优越。
下机数据通常以`.h5`格式存储,需要通过特定的软件工具转换成`.bam`格式,以便于常规的生物信息学分析。PacBio提供了SMRT Analysis软件套件(如SMRTAnalysis 2.3 for PacBio RSII和SMRTLink v5.1.0 for PacBio Sequel),以及相关的转换工具和BAM处理的配方。同时,开源社区如GitHub上的项目FALCON-formatter也提供了帮助转换和解析三代测序数据的解决方案。
三代测序预处理的完整管道通常涉及多个步骤,包括数据质量检查、基线校正、信号到碱基的转换、错误率降低等,每个步骤都需要使用专门的软件和算法。完成这些预处理后,数据可以用于组装基因组、检测结构变异、表观遗传学研究等多种生物学应用。三代测序预处理是基因组学研究中不可或缺的一环,它对于充分利用PacBio测序技术的优势至关重要。
199 浏览量
2021-10-31 上传
104 浏览量
256 浏览量
178 浏览量
fanyucai1
- 粉丝: 36
- 资源: 49
最新资源
- webservice
- EXTJS 中文手册
- ubuntu8.04速成手册1.0
- Installing & Configuring Developing With XAMPP
- c#中treeview的使用方法
- 《华为认证网络工程师》自测题
- c#中进度条的使用技巧
- cn_foundation_Actionscript3.0_Animation
- R1762_R2632_R2700 RGNOS10.2配置指南_第四部分 应用协议配置指南
- 一个中专生的程序员之路
- R1762_R2632_R2700 RGNOS10.2配置指南_第三部分 IP地址与服务配置指南
- 详解西门子间接寻址详解西门子间接寻址
- 微 软 C 编 程 精 粹
- MyEclipse 6 Java 开发中文教程
- C#完全手册.pdf
- VARIANT的用法