MATLAB sort函数在生物信息学中的应用:序列排序与基因组分析
发布时间: 2024-06-11 03:54:09 阅读量: 78 订阅数: 35 


matlab 使用 sort 函数来对数组进行排序 代码
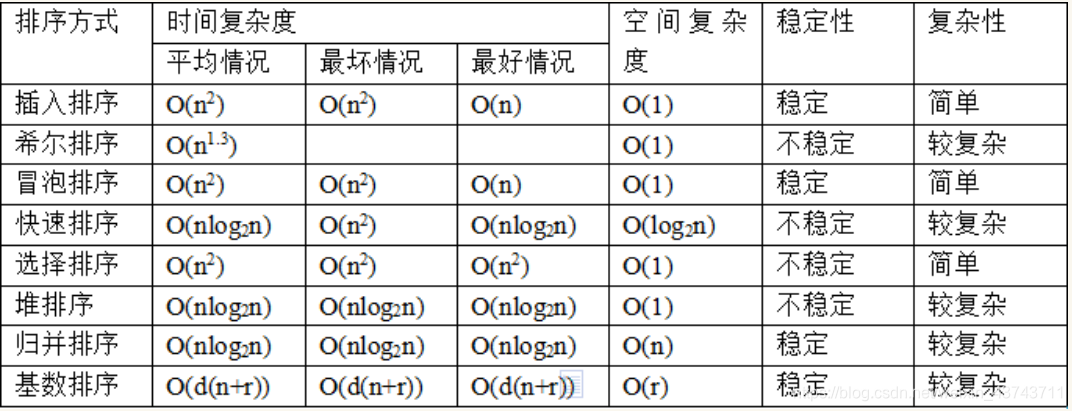
# 1. MATLAB sort函数简介**
MATLAB 中的 `sort` 函数用于对数组或矩阵中的元素进行排序。它是一个内置函数,具有以下语法:
```
[sortedArray, sortedIndices] = sort(array, dimension, direction)
```
其中:
* `array`:要排序的数组或矩阵。
* `dimension`(可选):指定沿哪个维度进行排序。默认为 1(按行)。
* `direction`(可选):指定排序方向。默认为 'ascend'(升序)。
# 2. 序列排序应用
序列排序是生物信息学中的一项基本技术,用于确定DNA或RNA分子的碱基序列。MATLAB提供了各种函数和工具箱,用于序列排序和分析。本章将介绍序列排序在DNA和RNA序列中的应用。
### 2.1 DNA序列排序
**2.1.1 序列读取和预处理**
DNA序列排序的第一步是读取原始序列数据。MATLAB可以使用`fastaread`函数从FASTA文件中读取序列。读取的序列可能包含错误和杂质,因此需要进行预处理以提高排序精度。预处理步骤包括:
```matlab
% 读取FASTA文件
sequence = fastaread('sequence.fasta');
% 转换序列为字符数组
sequence = char(sequence.Sequence);
% 修剪末端空白字符
sequence = strtrim(sequence);
% 移除非碱基字符
sequence = regexprep(sequence, '[^ACGT]', '');
```
**2.1.2 序列比对和排序**
序列预处理后,可以进行序列比对和排序。MATLAB提供了`bioinfo`工具箱,其中包含用于序列比对和排序的函数。序列比对可以识别序列之间的相似性和差异性,而排序可以将序列按相似性或其他标准排列。
```matlab
% 序列比对
alignment = nwalign(sequence1, sequence2);
% 序列排序
[sortedSequences, order] = sort(sequences);
```
### 2.2 RNA序列排序
**2.2.1 序列拼接和注释**
RNA序列排序涉及将来自不同片段的RNA序列拼接成一个连续序列。MATLAB可以使用`bioinfo`工具箱中的`assemble`函数进行序列拼接。拼接后的序列需要进行注释,以识别基因、外显子和内含子等特征。
```matlab
% 序列拼接
assembledSequence = assemble(sequences);
% 序列注释
annotation = annotate(assembledSequence, 'gene', 'exon', 'intron');
```
**2.2.2 序列比对和表达分析**
注释后的RNA序列可以进行比对和表达分析。比对可以识别不同样本或条件下的RNA序列差异。表达分析可以量化不同基因或转录本的表达水平。
```matlab
% 序列比对
alignment = nwalign(sequence1, sequence2);
% 表达分析
expressionLevels = rnaseq(sequences, 'gene', 'counts');
```
0
0





