【R语言高级用户指南】:掌握dbscan包的进阶技能
发布时间: 2024-11-03 17:14:41 阅读量: 54 订阅数: 50 

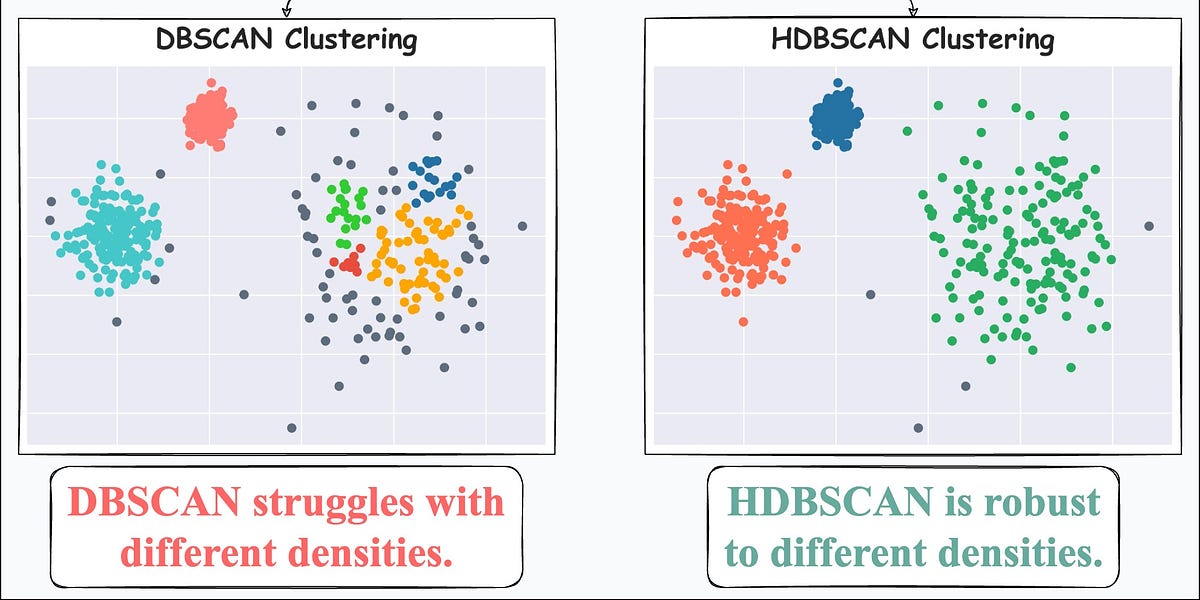
# 1. dbscan包概述与核心概念
数据聚类是一种无监督学习方法,用于将相似的观测数据点组合在一起。`dbscan`是R语言中用于执行基于密度的空间聚类算法的一个包,其核心思想是将紧密连接的点归为一类。它主要关注数据点密度的变化,特别适用于发现任意形状的簇,而不需预先设定簇的数量。`dbscan`通过两个主要参数——邻域半径(epsilon)和邻域内的最小点数(minPts)来确定数据点之间的关系。在处理含有噪声的数据集时,dbscan显示出独特的优势,因为它可以识别并标记出那些不属于任何簇的点。
接下来的章节将详细介绍如何安装和载入`dbscan`包,使用它进行数据聚类,以及如何解读聚类结果和进行可视化。我们还将探讨dbscan在不同领域的应用,并展望它的未来发展趋势。
# 2. dbscan包的基础使用
在深入了解和应用dbscan包之前,有必要掌握其基础使用方法。这一章将从安装、载入到数据聚类,直至结果解释与可视化,逐层深入介绍dbscan包的基本用法。
## 2.1 安装与载入dbscan包
### 2.1.1 安装方法与注意事项
在使用dbscan包之前,首先需要在R环境中安装它。安装可以借助CRAN镜像进行,使用以下命令:
```R
install.packages("dbscan")
```
安装完成之后,应检查该包是否可以被正常载入。在R中使用`library()`函数来载入dbscan包:
```R
library(dbscan)
```
一旦包被成功载入,我们就可以开始使用其中包含的函数和算法了。安装和载入时需要特别注意几个方面:
- 确保R语言的版本与dbscan包兼容。
- 在安装过程中,若使用了特定的CRAN镜像,要确保该镜像的同步状态良好。
- 载入包时,若出现警告或错误信息,应该根据提示进行相应的解决操作,例如安装缺失的依赖包。
### 2.1.2 载入dbscan包与基础函数介绍
dbscan包中包含多个重要的函数,其中核心的几个函数如下:
- `dbscan()`: 执行DBSCAN聚类算法。
- `kNNdistplot()`: 绘制k-最近邻距离图,有助于确定DBSCAN算法的参数。
- `frNN()`: 计算样本的最近邻距离。
- `kNN()`: 计算样本的k-最近邻。
下面举例说明如何使用`kNNdistplot()`函数:
```R
# 生成一些随机数据
set.seed(123)
data <- matrix(rnorm(300), ncol=2)
# 计算最近邻距离
kdist <- kNNdistplot(data, k = 5)
# 绘制k最近邻距离图
plot(kdist)
```
通过上述代码,我们能够得到一个k=5时的k-最近邻距离图,这有助于我们在使用`dbscan()`函数时确定合适的`eps`和`minPts`参数。
## 2.2 使用dbscan进行数据聚类
### 2.2.1 dbSCAN算法的基本原理
DBSCAN(Density-Based Spatial Clustering of Applications with Noise)是一种基于密度的聚类算法,其核心思想是将具有足够高密度的区域划分为簇,并能在带有噪声的空间数据库中发现任意形状的聚类。
### 2.2.2 核心参数的理解与设置
DBSCAN算法有两个核心参数:`eps`(邻域半径)和`minPts`(核心点的最小邻居数)。
- `eps`确定了用于衡量邻域密度的半径大小。
- `minPts`定义了一个点至少需要多少个邻居才能被认为是核心点。
在实际应用中,这两个参数的选择往往需要根据数据的特性进行调整。例如,数据分布密集时可以减小`minPts`,而数据分布较为稀疏时,则需适当增大`minPts`。
```R
# 聚类分析示例
set.seed(123)
dbscan_results <- dbscan(data, eps = 0.5, minPts = 10)
```
在上述示例中,我们将eps设为0.5,minPts设为10,意味着我们将搜索以每个点为中心,0.5为半径的邻域,并且至少需要10个点在该邻域内才能成为核心点。
## 2.3 结果解释与可视化
### 2.3.1 聚类结果的解读
DBSCAN算法返回的聚类结果包括每个点的标签,我们可以使用这些标签来分析数据的聚类情况。
```R
table(dbscan_results$cluster)
```
该命令将返回每个聚类中的点的数量统计,有助于我们理解数据的分布情况。
### 2.3.2 可视化工具与方法
可视化是解释聚类结果的直观手段。常用的可视化工具包括R的`ggplot2`包,以及dbscan包自带的`plot()`函数。
```R
# 使用ggplot2进行高级绘图
library(ggplot2)
ggplot(data, aes(x, y, color = as.factor(dbscan_results$cluster))) +
geom_point() +
theme_minimal()
```
上面的代码块使用`ggplot2`将DBSCAN聚类结果绘制在二维数据点散点图上,不同颜色代表不同的聚类。
为了更进一步理解如何解读聚类结果,以下是通过使用dbscan包进行聚类分析并将其结果可视化的具体步骤:
1. 使用`dbscan()`函数执行DBSCAN聚类,得到聚类结果。
2. 使用基础R的`plot()`函数绘制聚类的散点图,根据聚类结果中每个点的标签指定颜色。
3. 如果有需要,可以使用`ggplot2`包对散点图进行进一步美化和定制,例如添加坐标轴标签、图例标题、调整主题等。
通过上述步骤,我们可以将聚类结果更直观地展示出来,并且根据可视化的结果进一步分析和解释数据的聚类情况。
# 3. dbscan包的高级特性
随着数据规模的扩大和应用的深入,DBSCAN算法的高级特性变得尤为重要。这一章节将深入探讨如何处理大规模数据集、自定义邻域参数以及应对高维数据聚类的挑战。本章节将会揭示dbscan包在数据科学家手中变得更加灵活与强大。
## 3.1 处理大规模数据集
### 3.1.1 数据预处理技巧
处理大规模数据集时,数据预处理是关键的一步,它能够显著影响聚类算法的效率和准确性。在使用DBSCAN之前,应该对数据进行归一化处理,以消除不同量纲的影响。使用诸如PCA(主成分分析)或者t-SNE(t-分布随机邻域嵌入)的技术可以有效降低数据的维度,同时保留数据的重要结构特征。此外,识别并处理离群点也是预处理的一个重要方面,因为离群点可能会显著影响聚类结果。
```python
# 示例代码:数据预处理
from sklearn.preprocessing import StandardScaler
from sklearn.decomposition import PCA
# 假设df是需要处理的DataFrame数据
scaler = StandardScaler()
scaled_data = scaler.fit_transform(df)
# 应用PCA进行降维
pca = PCA(n_components=0.95) # 保留95%的方差
reduced_data = pca.fit_transform(scaled_data)
```
在上述代码中,首先使用`StandardScaler`对数据进行了归一化处理,接着通过`PCA`将数据降维到只保留95%的方差,这是一种常见的降维策略。通过降维,可以减少DBSCAN处理数据的时间,尤其是在处理大规模数据集时。
### 3.1.2 高效算法选择与参数优化
在大规模数据集上使用DBSCAN时,选择一个高效的算法实现是至关重要的。dbscan包中的`HDBSCAN`(Hierarchical DBSCAN)是DBSCAN的一种优化
0
0





