OpenCV模板匹配在生物信息学中的应用:分析基因序列,解锁生命奥秘
发布时间: 2024-08-05 23:10:45 阅读量: 34 订阅数: 29 


OpenCV 模板匹配全解析:从单模板到多模板的实战指南
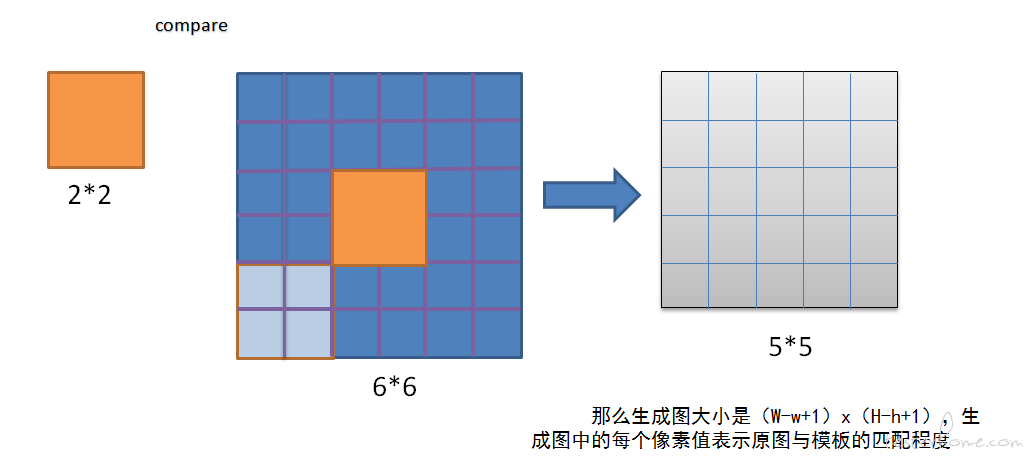
# 1. OpenCV模板匹配简介**
模板匹配是一种图像处理技术,用于在图像中查找与给定模板图像相匹配的区域。在OpenCV中,模板匹配通过`cv2.matchTemplate`函数实现,该函数计算模板图像与目标图像之间的相似性度量。
OpenCV提供了多种相似性度量方法,包括相关性系数和归一化互相关。相关性系数衡量两个图像之间的线性相关性,而归一化互相关通过归一化相关性系数来消除图像亮度差异的影响。
# 2. OpenCV模板匹配在基因序列分析中的理论基础
### 2.1 模板匹配算法原理
模板匹配算法是一种图像处理技术,用于在目标图像中查找与模板图像相似的区域。在基因序列分析中,模板图像可以是已知的基因序列,而目标图像可以是待分析的基因序列。
#### 2.1.1 相关性系数
相关性系数是一种衡量两个序列相似性的统计量。它计算为两个序列中对应元素的协方差除以两个序列的标准差的乘积。相关性系数的值在-1到1之间,其中:
* -1表示完全负相关
* 0表示不相关
* 1表示完全正相关
在模板匹配中,相关性系数用于衡量模板图像和目标图像中对应区域的相似性。相关性系数越高,表明两个区域越相似。
#### 2.1.2 归一化互相关
归一化互相关是相关性系数的一种变体,它通过将相关性系数除以模板图像和目标图像的标准差的乘积来归一化。这使得归一化互相关的值在0到1之间,其中:
* 0表示完全不相似
* 1表示完全相似
归一化互相关在模板匹配中更常用于衡量相似性,因为它不受模板图像和目标图像大小的影响。
### 2.2 生物信息学中基因序列匹配的挑战
在生物信息学中,基因序列匹配面临着以下挑战:
* **序列长度差异:**基因序列的长度可以有很大差异,这使得模板匹配算法难以在不同长度的序列上有效工作。
* **序列变异:**基因序列中存在变异,例如单核苷酸多态性(SNP)和插入和缺失(InDel),这可能会影响模板匹配算法的准确性。
* **背景噪声:**基因序列中可能存在背景噪声,例如测序错误和重复序列,这可能会干扰模板匹配算法。
为了克服这些挑战,需要对模板匹配算法进行调整和优化,以使其适用于基因序列分析。
# 3. OpenCV模板匹配在基因序列分析中的实践
### 3.1 基因序列预处理
基因序列预处理是模板匹配算法应用前的必要步骤,旨在提高匹配精度和效率。主要包括以下两个方面:
#### 3.1.1 序列转换
基因序列通常以FASTA或FASTQ格式存储,需要将其转换为OpenCV可以处理的格式。常用的转换方法是将序列转换为NumPy数组,其中每个元素代表一个碱基。
```python
import numpy as np
# FASTA格式序列
fasta_sequence = ">seq1\nACGTACGT"
# 转换为NumPy数组
sequence_array = np.array([ord(c) for c in fasta_sequence])
```
#### 3.1.2 序列对齐
序列对齐是指将两个或多个序列进行比较,以找出它们的相似性和差异性。在模板匹配中,序列对齐用于将查询序列与模板序列进行比对,以确定匹配区域。常用的序列对齐算法包括Needleman-Wunsch算法和Smith-Waterman算法。
```python
from Bio import pairwise2
# Needleman-Wunsch算法对齐
alignment = pairwise2.align.globalms(seq1, seq2, 1, -1, -1, -1)
# 获取对齐序列
aligned_seq1, aligned_seq2 = alignment[0][0], alignment[0][1]
```
### 3.2 模板匹配算法的应用
#### 3.2.1 匹配模式的选取
匹配模式是模板匹配算法中用于在查询序列中搜索的子序列。在基因序列分析中,匹配模式通常是长度为15-25个碱基的短序列,代表已知的基因序列或突变位点。
#### 3.2.2 匹配结果的评估
模板匹配算法返回一个匹配结果数组,其中每个元素表示查询序列中与模板序列匹配的起始位置。匹配结果的评估需要考虑以下指标:
* **匹配得分:**匹配区域的相似性度量,通常使用相关性系数或归一化互相关。
* **匹配长度:**匹配区域的长度,反映匹配的准确性。
* **匹配位置:**匹配区域在查
0
0





