神经诱导:双全基因组编码与lncRNA筛选的深度解析
157 浏览量
更新于2024-06-18
收藏 2.51MB PDF 举报
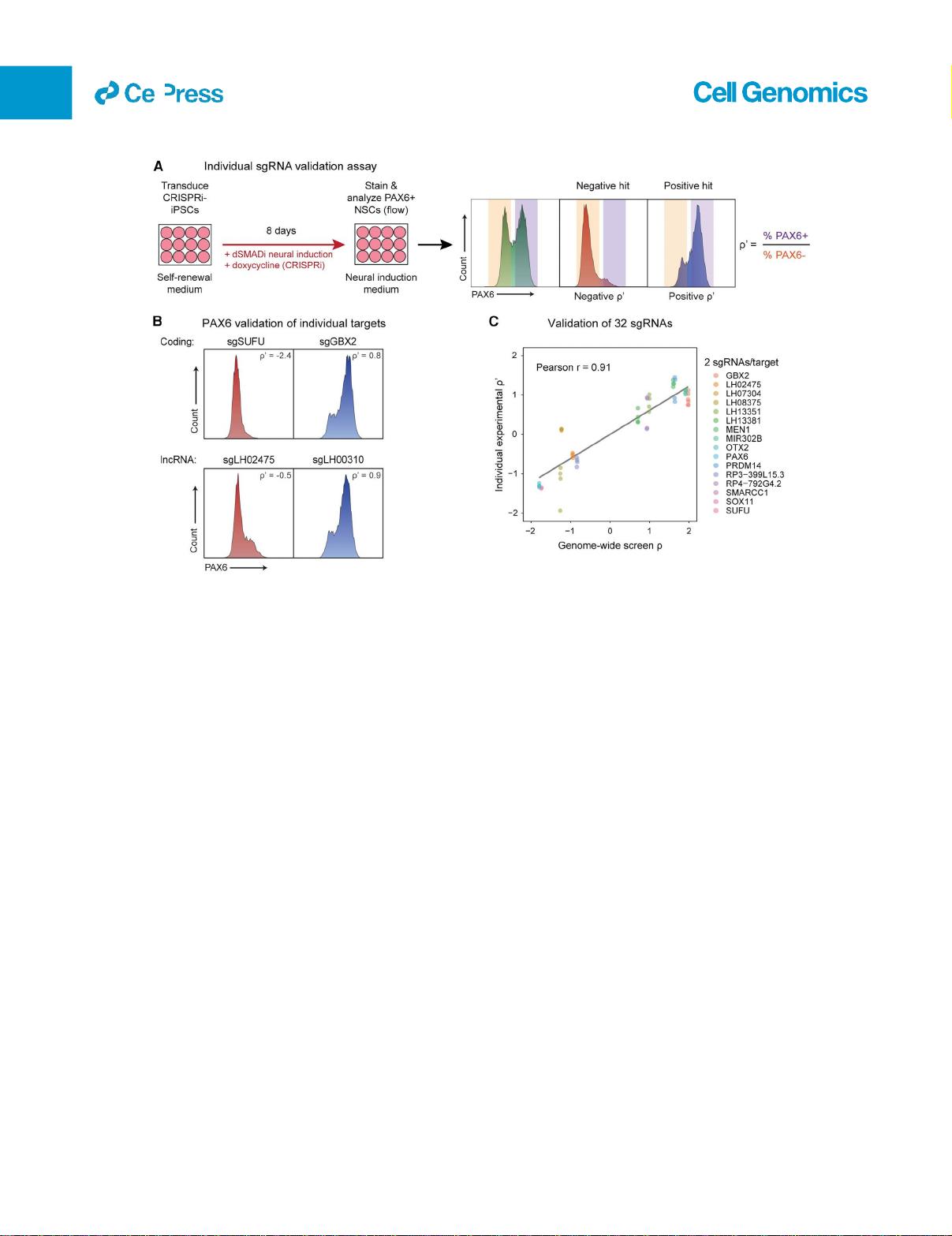
在"神经诱导中的双全基因组编码和lncRNA筛选"的研究中,David Wu等作者探讨了一种创新的方法来深入理解诱导性多能干细胞(iPSC)向神经元方向分化的过程。他们利用CRISPRi (Clustered Regularly Interspaced Short Palindromic Repeats) 技术,同时进行全基因组编码和长非编码RNA (lncRNA) 的筛选,以揭示这两种类型基因在这一复杂生物过程中独特的功能。
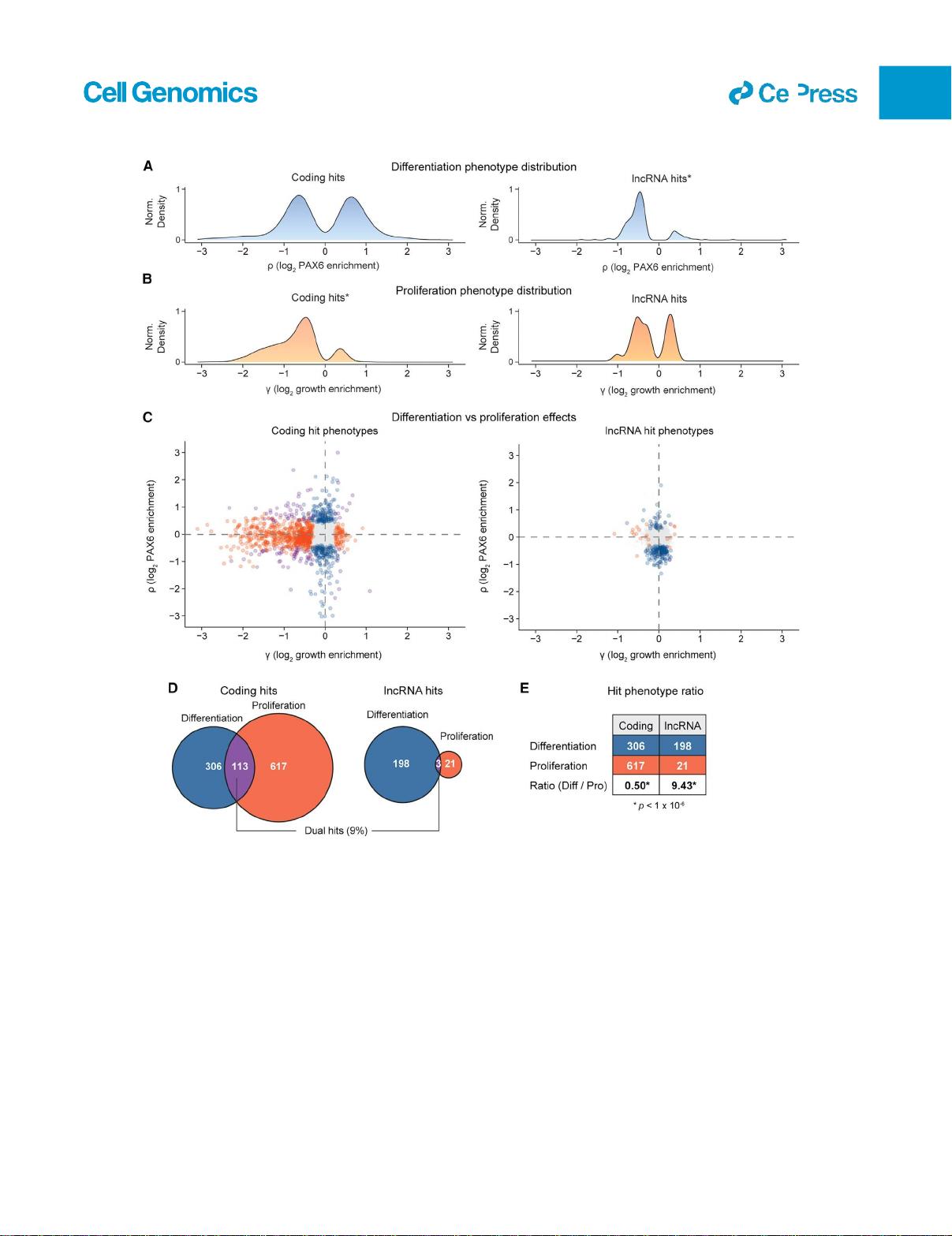
全基因组编码和lncRNA的双重筛选策略提供了前所未有的视角,因为通常编码基因在研究中受到更多关注。然而,Wu等人发现,在神经诱导过程中,lncRNA的作用可能更为丰富和复杂,它们可能参与调控细胞命运决定和分化路径的不同阶段。通过Perturb-seq技术,研究人员能够精细地分析这些非编码RNA对基因表达的影响,揭示了它们与编码基因之间动态的相互作用。
亮点部分着重于以下几点:
1. **方法创新**:使用双全基因组筛选策略,这在以前对于lncRNA的功能研究中较少见,为非编码基因的生物学角色提供了新的见解。
2. **功能差异**:编码基因和lncRNA在神经诱导中的作用呈现出显著的差异,表明它们各自在调控途径中扮演着独特且互补的角色。
3. **数据可视化工具**:研究者提供了danlimlab.shinyapps.io/dualgenomewide这样的交互式资源,便于其他研究者探索和分析数据。
4. **研究成果**:论文发表在《细胞基因组学》上,支持开放获取,强调了科学共享的重要性。
研究团队由多个领域的专家组成,包括David Wu、Aunoy Poddar、Elpiniki Ninou、Elizabeth Huang等人,他们在再生医学、神经科学和基因组学等领域具有深厚背景,共同推进了对iPSC分化机制的理解。
这项工作的主要贡献在于填补了当前对编码基因组和lncRNA在细胞分化过程中的系统理解空白,为进一步优化神经细胞生成策略提供了新的理论基础。未来的研究可能会基于这些发现,开发出更精确的调控手段,促进组织修复和再生医学的发展。
点击了解资源详情
点击了解资源详情
点击了解资源详情
2021-05-22 上传
2021-06-28 上传
2021-05-30 上传
2021-07-26 上传
2023-06-12 上传
cpongm
- 粉丝: 5
- 资源: 2万+
最新资源
- 基于Python和Opencv的车牌识别系统实现
- 我的代码小部件库:统计、MySQL操作与树结构功能
- React初学者入门指南:快速构建并部署你的第一个应用
- Oddish:夜潜CSGO皮肤,智能爬虫技术解析
- 利用REST HaProxy实现haproxy.cfg配置的HTTP接口化
- LeetCode用例构造实践:CMake和GoogleTest的应用
- 快速搭建vulhub靶场:简化docker-compose与vulhub-master下载
- 天秤座术语表:glossariolibras项目安装与使用指南
- 从Vercel到Firebase的全栈Amazon克隆项目指南
- ANU PK大楼Studio 1的3D声效和Ambisonic技术体验
- C#实现的鼠标事件功能演示
- 掌握DP-10:LeetCode超级掉蛋与爆破气球
- C与SDL开发的游戏如何编译至WebAssembly平台
- CastorDOC开源应用程序:文档管理功能与Alfresco集成
- LeetCode用例构造与计算机科学基础:数据结构与设计模式
- 通过travis-nightly-builder实现自动化API与Rake任务构建