JTI和MOSTWAS模型的参考面板。对每个祖先组和每个生物库
进行关联测试。最后,我们使用逆方差加权对每个生物库和每个
祖先组的效应量进行荟萃分析。最后,我们将GTA与多个后续分
析结合起来。
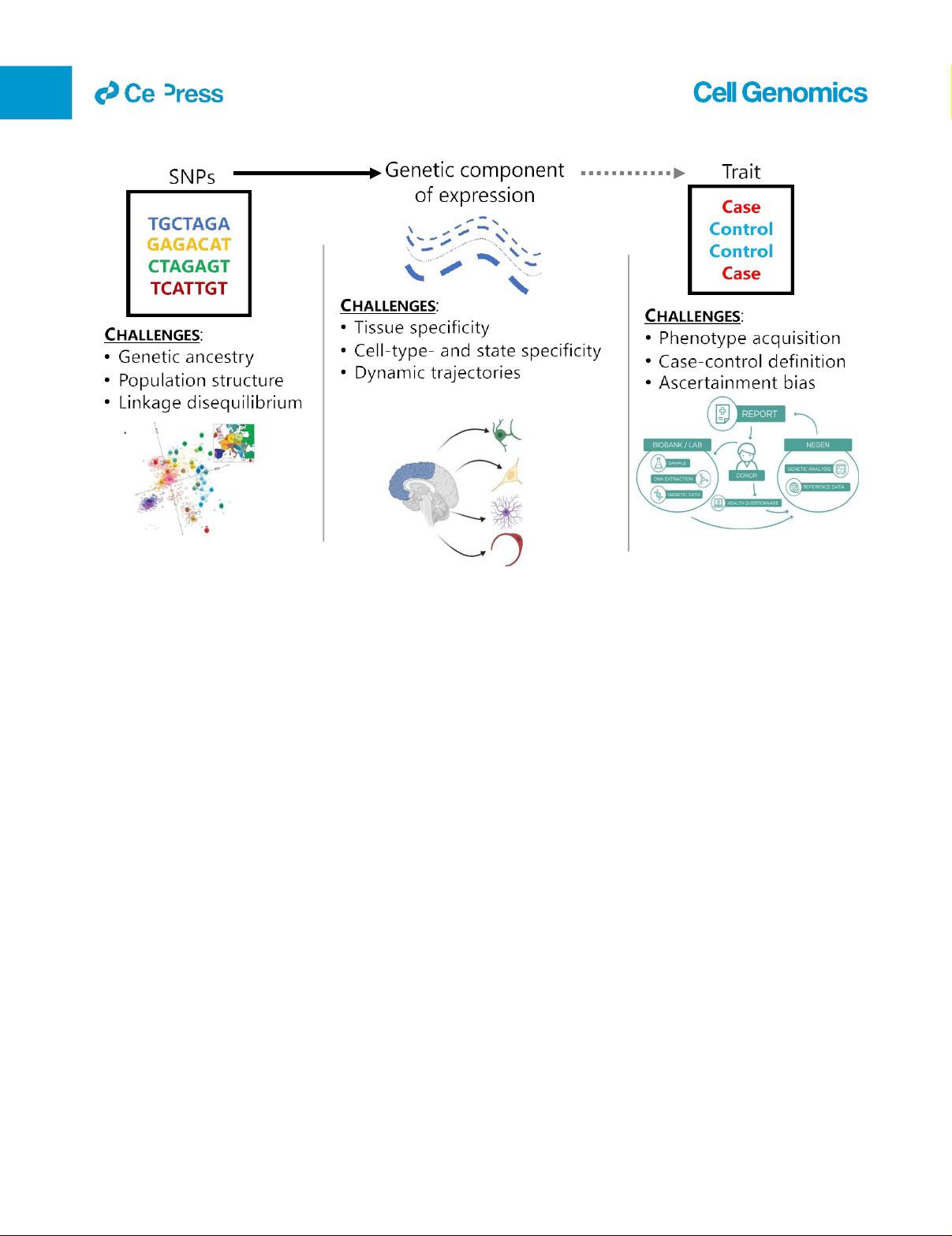
表达式模型不能跨祖先组移植
GBMI然而,最佳TWAS需要遗传学和组织特异性基因表达数据
的有效训练数据集,这对于非欧洲人群仍然缺乏。在TWAS中检
测GTA的能力取决于表达遗传力和表达模型的预测能力。
27
因
此,准确预测不同人群的表达是必要的,以确保TWAS关联不限
于欧洲人群。对于第一个GBMI TWAS,我们将分析限制在欧洲
血统群体,因为eQTL参考组中的非欧洲血统个体数量较少。
28
随着非欧洲人群中eQTL数据集的样本量增加,TWAS管道将包
括这些人群的表达模型(STAR方法)。我们举例说明了在构建
这些跨祖先群体的表达模型时的一些挑战。
在 GTEx 中 的 五 个 组 织 中 ,具有来 自 欧 洲 ( EUR ) 和 非 洲
(AFR)血统的>70个样本,我们使用弹性网络正则化回归训练
EUR和AFR特异性模型,并将表达插补到对齐的(即,训练和插
补样本具有相似的祖先)和未对齐(即,训练和插补样本具有不
同的祖先)组。
29
,
30
对于背景,我们还建立了祖先未知的模型,
将EUR和AFR样本合并在一起。预测性能采用调整后的5倍交叉
验证R
2
计算,以考虑样本量(STAR方法)。对于该分析,我们
不使用JTI或MOSTWAS模型,因为它们分别需要多组织样本或
比AFR祖先个体更大的样本量。这两种方法都借用了弹性网络回
归,从这个分析的结果是适用于这两种方法。
在这些组织中,与在AFR样本中训练的模型相比,在EUR样本
中训练的模型在AFR样本中的表现平均差3超过80%的基因模型
在AFR样本中训练时具有更强的性能。祖先对齐和未对齐的调整
R2和百分比差异的分布(图S2)强调,0.02
0.04
预测R2的增加是明显的。类似的趋势适用于插补到下采样
EUR插补样本中的祖先特定模型(图S3和S4;表S1-事实上,我
们观察到,与祖先不知情(训练样本中有EUR和AFR祖先的个
体)模型相比,祖先特异性模型在具有一致祖先的样本中表现出
更大的预测
R2
(
图1)。
图2B;表S4),尽管样本量增加如果我们通过包括其他祖先(亚
洲人、美洲印第安人和所谓的未知祖先)的个体来进一步增加训
练样本量,这一观察结果也成立(图S5)。这一观察结果不仅
与Patel等人最近的结果一致,这些结果显示了不同祖先群体之
间基因表达的因果效应大小的差异,
31
还强调了在表达预测中进
行祖先匹配的必要性,以及在eQTL研究中招募更多的非欧洲祖
先患者。
祖先未知模型可能在AFR样本中表现不佳的一个原因是由于
EUR 和 AFR 祖 先 群 体 之 间 预 测 SNP 的 次 要 等 位 基 因 频 率
(MAF)的差异重要的是,这种差异通常不是特定于任何一个祖
先。相反,由于MAF差异,训练或参考数据集中的祖先不平衡为
了在AFR和EUR祖先群体中结合常见的SNP,我们使用在AFR
和EUR样本中MAF超过各种阈值的SNP训练祖先未知和祖先特
异性模型。排除MAF 0.01的SNP改善了所有组织中祖先未知模
型的预测性能(图S6;表S5)。然而,当MAF截止值增加时,祖
先特异性模型和祖先未知模型之间的预测性能差距并未减小(图
2B;表S4)。这一观察结果可能反映了丢弃祖先特异性的稀有
SNP忽略了对基因表达具有大的祖先特异性影响的变体。此外,
排除罕见的祖先特异性SNP并不能解决EUR和AFR样本中LD的
差异,这些差异导致不同的正则化路径,从而导致SNP基因权
重。解决表达模型的可移植性仍然是一个开放的研究方向;从功
能注释或跨不同细胞类型或细胞状态特异性背景借用信息的方法
可以弥补预测性能的这一差距,类似于多基因评分(PGS)
32
或
多基因转录组风险评分的最新发展,这是一种使用基因表达的多
SNP预测因子构建的PGS类似物。
三十三,三十四
荟萃分析 战略 必须 被 GBMI的另一个关键考虑因素涉及
使用GWAS汇总统计量的荟萃分析。TWAS通过用来自表达模型
的SNP-基因权重对来自GWAS汇总统计的标准化SNP-性状效应
大小进行加权来估计GTA。为了解释SNP之间的相关性,外部
LD参考面板,如1000个基因组计划,
35
用于估计TWAS关联的标
准误差。因此,GWAS队列中参考和样本内LD的差异影响了基
于统计学的汇总TWAS关联和直接插补到个体水平基因型的
TWAS关联理想情况下,样本内LD将给出TWAS标准误差的最佳
估计,但根据特定的遗传数据共享和隐私政策,一些生物库无法
提供此信息。甚至欧元血统人群亚组之间LD的偏离此外,由于
SNP-基因权重的估计值受到eQTL组中LD的影响,因此eQTL和
GWAS组之间LD的差异也将影响TWAS效应大小的估计。