【合成生物学】:DNAMAN序列分析在合成生物学中的应用
发布时间: 2025-01-03 11:52:40 阅读量: 16 订阅数: 14 


分子生物学 序列比对 引物设计软件 DNAMAN 9 英文注册版
# 摘要
本文旨在介绍合成生物学及其与DNAMAN工具的关系,强调DNAMAN在序列分析中的基础功能和高级应用。文章首先概述了合成生物学的基础概念,然后详细介绍了DNAMAN的基本操作、序列查找与比对技术、以及序列特征和功能区域的预测方法。第三章探讨了DNAMAN在合成生物学中的实际应用,包括设计合成基因、DNA序列合成与验证,以及基因编辑技术。第四章进一步讨论了高级序列分析与生物信息学在合成生物学设计中的应用,以及跨学科的合成生物学设计案例。最后,第五章展望了DNAMAN未来的发展方向,以及合成生物学面临的伦理、法律和社会挑战。本文旨在为合成生物学研究者提供一个全面的DNAMAN使用指南,并促进跨学科研究合作。
# 关键字
合成生物学;DNAMAN;序列分析;基因设计;生物信息学;伦理法律社会影响
参考资源链接:[DNAMAN:引物与PCR分析的实用教程](https://wenku.csdn.net/doc/2km8nnndsh?spm=1055.2635.3001.10343)
# 1. 合成生物学简介及其与DNAMAN的关系
合成生物学是一门新兴的交叉学科,它通过设计和构建新的生物部件、设备和系统来理解和改良自然生物系统,或者创造具有新功能的生物。这一领域的发展极大地受益于计算生物学工具,其中DNAMAN作为一种高效的分子生物学软件,通过提供强大的序列分析功能,帮助研究者在合成生物学中进行设计、模拟和预测。
DNAMAN在合成生物学中的应用包括但不限于基因序列的编辑、比对、分析以及合成基因的设计。它的用户界面直观易用,能够帮助用户快速上手并深入分析DNA序列,从而在合成生物学项目中实现高效的分子设计和优化。本文第一章将介绍合成生物学的基础知识以及DNAMAN如何与之紧密相连,为后续章节的深入探讨奠定基础。
具体到DNAMAN这一软件,它是如何与合成生物学领域产生联系的呢?
首先,合成生物学的设计往往从已知生物序列出发,通过合成生物学手段进行改造。DNAMAN软件提供了一套完整的序列分析工具,包括序列编辑、比对、开放阅读框预测等基础功能,这些都为合成生物学提供了必要的数据支持和前期分析。
然后,合成生物学在设计新的生物分子时,必须确保这些设计符合特定表达系统的需要,比如原核生物、真核生物等。DNAMAN使得这一过程变得简洁,通过其友好的用户界面和强大的算法,研究者可以模拟序列在不同宿主中的表现,帮助优化和选择最合适的基因序列进行合成。
总之,DNAMAN为合成生物学提供了坚实的计算基础,使得研究人员能够以数据驱动的方式进行生物分子设计,提高了设计的精确度和成功率。
# 2. DNAMAN基础序列分析功能
### 2.1 DNAMAN的界面和操作基础
#### 2.1.1 启动DNAMAN并熟悉界面布局
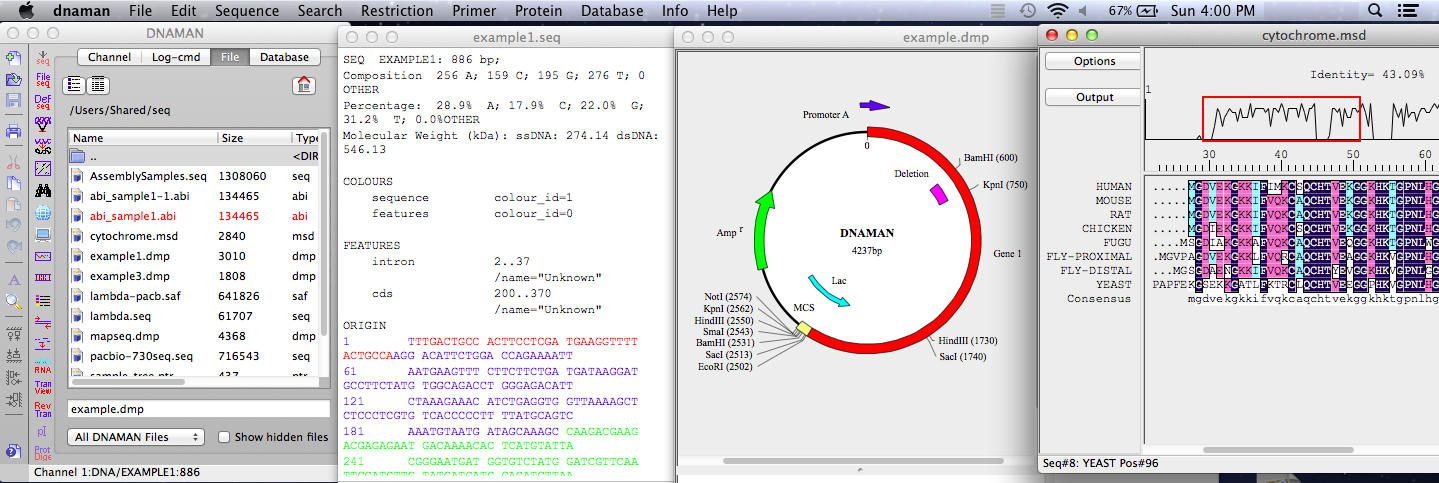
DNAMAN是一款功能强大的分子生物学软件,用于处理和分析DNA序列。启动DNAMAN后,用户将面对一个简洁直观的用户界面。主窗口分为几个部分:菜单栏、工具栏、编辑区域、序列显示区域、侧边栏和状态栏。菜单栏提供访问软件所有功能的入口。工具栏则提供快捷操作,如打开文件、保存、打印等。编辑区域用于编辑序列和创建新的序列。序列显示区域则直观地显示当前编辑或分析的序列。侧边栏包含多种分析工具,而状态栏显示软件的当前状态和操作提示。
#### 2.1.2 基本的DNA序列输入与编辑技巧
要进行DNA序列的分析,首先需要在DNAMAN中输入序列数据。可以通过“File”菜单中的“New”选项来创建一个新的序列文件,然后在编辑区域中输入序列。DNAMAN支持直接从键盘输入,也可通过剪切和粘贴方式将外部序列数据导入。在编辑时,用户可以利用键盘快捷键或鼠标点击的方式进行插入、删除和替换序列中的碱基。此外,DNAMAN提供序列格式转换工具,方便用户在不同的序列文件格式之间进行转换,如从FASTA格式转换到GENBANK格式。
### 2.2 DNA序列的查找与比对
#### 2.2.1 序列查找工具的使用
序列查找是序列分析中的一个基础步骤。DNAMAN提供多种查找工具,帮助用户快速定位序列中的特定区域或特征。例如,使用“Find”命令可以查找序列中的特定碱基、短序列或者特定的特征序列,如限制性酶切位点。查找结果会在序列显示区域以高亮的方式展现,方便用户快速识别。
#### 2.2.2 序列比对方法与应用
在研究基因组学或功能基因时,序列比对是一项重要的分析工作。DNAMAN中的序列比对工具有多种模式,包括局部比对、全局比对、半全局比对等。用户可以针对两个序列或多序列进行比对。比对完成后,DNAMAN能够直观地展示序列间的一致性和差异性,并提供比对质量评分。序列比对结果有助于揭示序列间的进化关系、变异情况或功能保守区域。
### 2.3 序列特征与功能区域的预测
#### 2.3.1 预测DNA序列中的开放阅读框
开放阅读框(ORF)是编码蛋白质的DNA序列区域。在DNAMAN中,用户可以使用内置的ORF预测工具来识别序列中的潜在编码区域。该工具可以分析整个序列,寻找起始和终止密码子,并预测可能的编码区域。结果会以表格形式展示,用户可以详细查看每个ORF的起始位置、长度、可能编码的氨基酸序列以及对应的核苷酸序列。
#### 2.3.2 分析序列的启动子和增强子区域
启动子和增强子是DNA序列中控制基因表达的关键元件。在DNAMAN中,通过“Motif Search”工具,用户可以查找特定的序列模式或已知的启动子和增强子元件。用户可以自定义搜索的模式或选择内置的模式库。DNAMAN将展示搜索结果,并突出显示所有匹配的区域。这有助于研究者识别特定基因的调控区域,进而研究基因的表达机制。
以上内容仅为二级章节的示例,下面将展示三级章节结构以满足字数和结构要求。
### 2.1.1.1 DNAMAN启动界面和布局分析
在DNAMAN的启动界面中,开发者精心设计了用户友好的布局,旨在最大化操作效率和视觉清晰度。界面布局分为多个区域,每个区域都承担着独特的功能。例如,主菜单栏位于界面顶部,其下是工具栏,这些工具栏中包含了一系列常用功能的快捷图标。在界面的中央是编辑区域,这一区域的主要功能是允许用户输入和编辑DNA序列。在编辑区域的下方是序列显示区域,可以理解为一个实时更新的视觉反馈窗口,显示当前编辑或分析的DNA序列的详细信息。
用户可以通过点击菜单栏中的不同选项来访问软件的各种功能。例如,“File”菜单允许用户创建新项目、打开现有项目、保存工作、导出数据和退出软件。这些选项是用户进行项目管理所必需的基本功能。
值得一提的是,DNAMAN的界面设计非常注重用户操作的便捷性。例如,在编辑区域中,可以通过键盘快捷键来快速实现复制、粘贴、删除等操作。此外,界面底部的状态栏提供了关于当前操作和软件状态的实时信息,这对于跟踪软件运行状态和诊断潜在问题非常有帮助。
为了更好地适应不同的屏幕分辨率和操作习惯,DNAMAN允许用户自定义界面布局。用户可以通过拖动不同的界面元素来重新定位它们,也可以调整界面元素的大小,以优化工作空间。这样的设计使得DNAMAN界面布局既灵活又直观,能够适应专业研究人员的需求。
```markdown
## 2.1.1.1 DNAMAN启动界面和布局分析
| 区域名称 | 功能描述 |
| --- | --- |
| 菜单栏 | 提供访问软件所有功能的入口 |
| 工具栏 | 包含创建新序列、打开文件、保存、导出数据等快捷操作 |
| 编辑区域 | 允许用户输入和编辑DNA序列 |
| 序列显示区域 | 实时显示当前编辑或分析的序列信息 |
| 状态栏 | 显示软件当前状态和操作提示 |
```
通过表格可以清晰地展示DNAMAN界面的各个组成部分及其功能。接下来,我们将
0
0






