基因组数据处理:Dask在生物信息学中的革命性应用
发布时间: 2024-09-29 22:58:55 阅读量: 101 订阅数: 38 

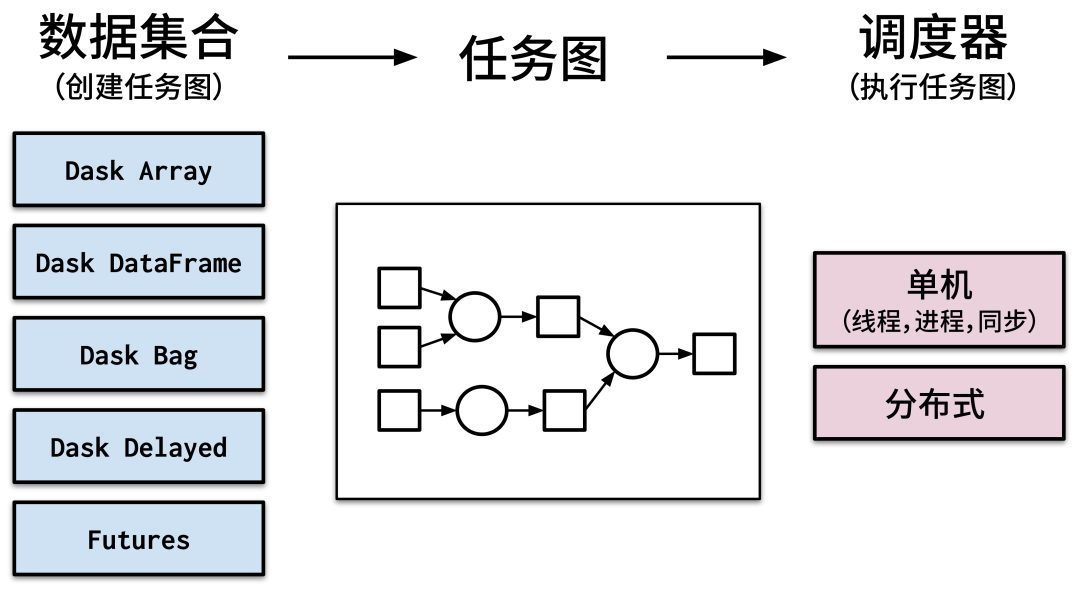
# 1. Dask概述及其在生物信息学中的潜力
## 1.1 Dask简介
Dask是一个开源的灵活并行计算库,专为处理大型数据集和复杂的计算任务而设计。它与Python生态系统中的其他工具,如NumPy、Pandas、Scikit-Learn等,保持良好的兼容性,并在这些库的基础上增加了并行处理的能力。Dask的核心在于它的动态任务调度系统,能够自动处理复杂的计算流程,并有效利用多核处理器和集群资源。
## 1.2 在生物信息学中的应用潜力
生物信息学领域不断产出海量数据,要求分析工具具备高度的可扩展性和处理大数据的能力。Dask的并行计算特性使其成为处理基因组学、表观基因组学、转录组学等大规模生物信息学数据的理想选择。通过Dask,研究人员可以轻松扩展计算任务到多核处理器或整个计算集群,显著提高数据处理速度,加速科学发现过程。
## 1.3 案例分析
例如,在基因组数据的处理中,Dask可以用来并行化处理大量的序列读取、比对、变异检测等任务。这不仅加快了分析流程,还减少了对昂贵硬件资源的依赖。对于非并行化的数据分析工具,Dask提供了一种便捷的方式,使得这些工具能够在现代计算集群上运行,为生物信息学研究提供了新的可能性。
# 2. Dask基础和并行计算理论
### 2.1 Dask的核心概念
Dask是一种灵活的并行计算库,专为Python生态系统设计,它利用了现有的Python工具,如NumPy、Pandas和Scikit-learn,使得并行计算既轻松又自然。Dask通过延迟计算(lazy evaluation)允许用户定义复杂的任务图(task graphs),并通过动态调度(dynamic scheduling)来优化执行。
#### 2.1.1 Dask的基本组件和架构
Dask的主要组件包括:
- **Dask DataFrame**:模仿Pandas DataFrame,用于处理结构化数据。
- **Dask Array**:模仿NumPy数组,用于处理数值计算。
- **Dask Bag**:用于处理非结构化数据,如JSON文件、日志文件。
- **Dask Delayed**:用于并行化任意Python函数。
Dask的架构允许在单机上并行计算,也可以扩展到分布式计算集群。其核心是一个动态任务调度器,它会自动将计算任务分配到可用的计算资源上。
#### 2.1.2 Dask与传统计算模型的对比
与传统的计算模型相比,Dask的主要优势在于:
- **延迟计算**:仅在需要结果时才执行计算,这有助于避免不必要的计算开销。
- **任务依赖性管理**:Dask自动追踪任务之间的依赖关系,并据此优化执行顺序。
- **内存高效**:Dask可以处理超出内存大小的数据集,因为它利用了硬盘空间来存储临时结果。
### 2.2 并行计算理论基础
#### 2.2.1 任务并行与数据并行的区别
并行计算主要有两种模型:任务并行和数据并行。
- **任务并行**侧重于同时执行不同的任务,这在处理具有多个独立子任务的问题时非常有效。
- **数据并行**侧重于将数据分成多个块,然后并行处理每个数据块。
Dask支持这两种并行模式,并且可以通过构建任务图来灵活组合它们。
#### 2.2.2 并行计算中的内存管理和调度策略
Dask在内存管理上采取了优化策略:
- **即时计算**:在处理数据时,尽可能在内存中保留计算结果,减少读写硬盘的次数。
- **内存优先级**:Dask会根据数据的重要性赋予不同的内存优先级,确保关键数据常驻内存。
在调度策略方面,Dask使用的是动态调度系统,能够根据工作负载的实际情况动态调整任务的执行,以获得最优性能。
### 2.3 Dask的并行计算机制
#### 2.3.1 Dask的动态任务调度
Dask的核心是其动态任务调度器,它:
- **监控任务状态**:跟踪哪些任务已经完成,哪些正在运行,哪些还在等待。
- **优先级决策**:根据任务依赖关系和资源可用性,动态调整任务执行顺序。
动态调度器使得Dask能够适应并优化计算环境的变化,例如当新的计算资源加入时,调度器会自动利用这些资源执行任务。
#### 2.3.2 处理大数据集时的性能优化
在处理大数据集时,性能优化至关重要:
- **数据局部性**:尽可能在数据所在的计算节点上执行计算任务,减少数据传输时间。
- **高效I/O**:优化数据读写操作,使用更高效的数据格式,比如Parquet,减少磁盘I/O时间。
通过这些策略,Dask可以显著提升大数据处理的效率和速度。
```python
import dask
from dask import delayed
# 示例代码:Dask Delayed的使用
@delayed
def compute(x, y):
return x + y
# 定义一些计算任务
a = compute(1, 2)
b = compute(3, 4)
c = compute(a, b)
# 执行计算任务
result = ***pute(c)
print(result)
```
在上述代码块中,我们定义了一个使用`@delayed`装饰器的函数`compute`,它延迟了实际的加法操作。然后,我们定义了三个计算任务`a`、`b`和`c`,它们依赖于前面计算的结果。通过调用`***pute(c)`,Dask将自动调度和执行这些任务。
在本章节中,我们详细探讨了Dask的基础知识和并行计算理论。在下一章中,我们将深入探讨Dask在生物信息学中的应用实践,展示如何利用Dask来处理基因组数据,以及Dask如何成为生物信息学研究的强大工具。
# 3. Dask在基因组数据处理中的应用实践
基因组学是一门研究生物体遗传信息的科学,它通常需要处理大量的数据。Dask,作为一个强大的并行计算库,已经在基因组学的数据处理和分析中展示了其巨大的潜力。这一章将详细探讨Dask在基因组数据处理中的应用实践。
## 3.1 基因组数据处理流程概述
### 3.1.1 从原始数据到分析结果的步骤
基因组数据处理是一个复杂的过程,它涵盖了从原始测序数据获取,到最终获得科学上有价值的信息的所有步骤。以下是基因组数据处理的基本步骤:
1. **数据获取与初步检查**:这一步涉及从测序仪器获取原始数据,并进行基本的质量检查。
2. **数据清洗与质量控制**:质量不佳的测序数据将被过滤和修正,这通常涉及到对测序读数进行剪切和校正。
3. **对齐与变异检测**:清洗后的读数需要被映射(对齐)到参考基因组,然后检测其中的遗传变异。
4. **基因表达分析**:对于转录组数据,接下来是量化基因表达水平。
5. **变异注释与分析**:最后,变异需要被注释和解释,以确定它们可能对生物学过程的影响。
### 3.1.2 数据格式和预处理的重要性
在处理基因组数据时,数据的格式和预处理步骤至关重要。常见的基因组数据格式包括FASTQ(原始测序数据)、BAM(对齐后的序列数据)、VCF(变异数据)等。预处理能够确保数据的准确性和一致性,这对后续的分析至关重要。Dask能够在这些预处理步骤中发挥其优势,通过并行处理快速处理大量数据。
## 3.2 Dask实现基因组数据的预处理
### 3.2.1 数据清洗和质量控制
在基因组数据的预处理阶段,Dask可以通过并行处理提高效率。下面的示例展示了如何使用Dask对FASTQ文件中的质量值进行检查和清洗:
```python
import dask.dataframe as dd
# 读取FASTQ文件
fastq_df = dd.read_csv('fastq_file.fastq', sep='\n', header=None, names=['read_id', 'sequence', 'comment', 'quality'])
# 数据清洗和质量控制
fastq_df = fastq_df[fastq_df['quality'] >= 'I'] # 过滤低质量序列
fastq_df = fastq_df[fastq_df['comment'] != '@']
```
在这个例子中,我们使用了Dask的DataFrame来并行读取和处理FASTQ文件。这一过程允许我们在多个核心上处理大量数据,而不会耗尽内存资源。Dask会自动管理任务的调度和内存管理。
### 3.2.2 对齐和变异检测
对齐和变异检测是基因组学数据处理中的关键步骤。使用Dask可以显著提高这些计算密集型任务的效率。下面是一个使用Dask进行简单对齐任务的示例:
```python
import dask.array as da
# 假设我们有一个表示序列的Dask数组
sequences = da.from_array(sequencing_data, chunks=
```
0
0





