生物信息学中的GA算法:基因序列分析与疾病诊断,探索生命奥秘
发布时间: 2024-07-03 22:50:48 阅读量: 71 订阅数: 27 

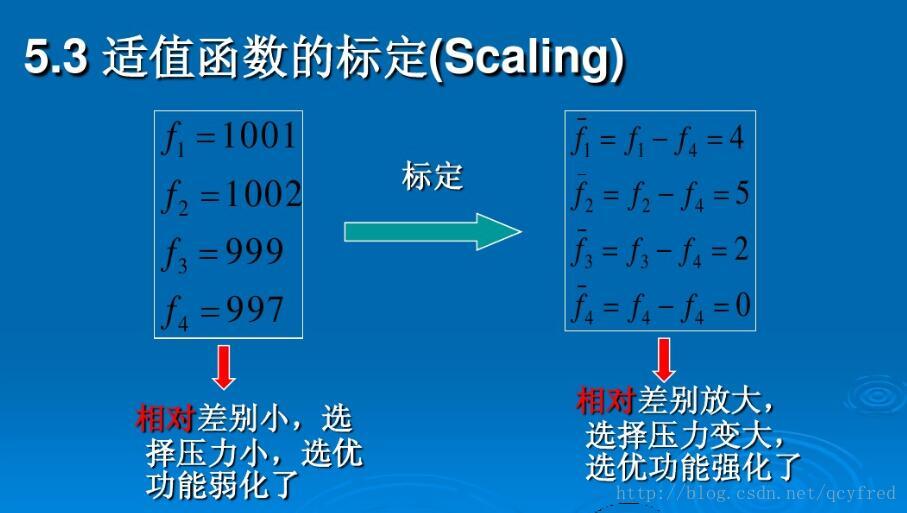
# 1. 生物信息学与GA算法概述**
生物信息学是一门交叉学科,它将计算机科学、数学和统计学等学科应用于生物学数据的分析和处理。随着生物技术的发展,生物信息学在基因组学、蛋白质组学、代谢组学等领域发挥着越来越重要的作用。
遗传算法(GA)是一种受进化论启发的优化算法。它模拟自然选择的过程,通过不断地选择、交叉和变异,从一组候选解中找到最优解。GA算法具有鲁棒性强、全局搜索能力好等优点,在生物信息学领域得到了广泛的应用。
# 2. GA算法在基因序列分析中的应用
GA算法在基因序列分析中发挥着至关重要的作用,尤其是在基因序列比对和组装以及基因变异检测方面。
### 2.1 基因序列比对与组装
**2.1.1 序列比对算法**
序列比对算法旨在比较两个或多个基因序列,找出它们的相似性和差异性。GA算法可以优化序列比对过程,提高比对的准确性和效率。
```python
import numpy as np
from scipy.spatial import distance
def ga_sequence_alignment(seq1, seq2):
# 初始化种群
population = np.random.randint(0, 2, size=(pop_size, seq_len))
# 迭代进化
for generation in range(num_generations):
# 计算适应度
fitness = np.apply_along_axis(lambda x: distance.hamming(x, seq2), 1, population)
# 选择
parents = np.random.choice(population, size=(pop_size, 2), replace=True, p=fitness / np.sum(fitness))
# 交叉和变异
offspring = np.empty_like(population)
for i in range(pop_size):
parent1, parent2 = parents[i]
crossover_point = np.random.randint(0, seq_len)
offspring[i] = np.concatenate((parent1[:crossover_point], parent2[crossover_point:]))
offspring[i] = mutate(offspring[i], mutation_rate)
# 更新种群
population = offspring
# 返回最优解
return population[np.argmax(fitness)]
```
**参数说明:**
* `seq1`, `seq2`: 待比对的基因序列
* `pop_size`: 种群规模
* `num_generations`: 迭代次数
* `mutation_rate`: 变异率
**代码逻辑分析:**
1. 初始化种群,每个个体代表一个可能的比对结果。
2. 迭代进化,计算每个个体的适应度(与目标序列的相似度),选择适应度高的个体作为父母。
3. 通过交叉和变异产生新的后代,形成新的种群。
4. 重复迭代,直到达到预定的进化代数或满足终止条件。
5. 返回适应度最高的个体,即最优的比对结果。
**2.1.2 序列组装策略**
序列组装是将短读序列拼接成更长的连续序列的过程。GA算法可以优化序列组装策略,提高组装的准确性和完整性。
```python
import networkx as nx
def ga_sequence_assembly(reads):
# 构建重叠图
graph = nx.Graph()
for read in reads:
for i in range(len(read) - overlap_len):
graph.add_edge(read[i:i+overlap_len], read[i+overlap_len:])
# 找到欧拉路径
eulerian_path = nx.eulerian_circuit(graph)
# 拼接序列
assembled_sequence = ''.join(eulerian_path)
# 返回组装后的序列
return assembled_sequence
```
**参数说明:**
* `reads`: 短读序列列表
* `overlap_len`: 重叠长度
**代码逻辑分析:**
1. 将短读序列构建成重叠图,其中节点表示重叠区域,边表示重叠关系。
2. 找到重叠图的欧拉路径,该路径代表一条遍历所有边的路径。
3. 将欧拉路径中的重叠区域拼接起来,得到组装后的序列。
### 2.2 基因变异检测
**2.2.1 SNP检测**
单核苷酸多态性(SNP)是基因序列中单个碱基的变异。GA算法可以优化SNP检测过程,提高检测的准确性和灵敏度。
```python
import numpy as np
from sklearn.metrics import roc_auc_score
def ga_snp_detection(ref_seq, reads):
# 初始化种群
population = np.random.randint(0, 4, size=(pop_size, seq_len))
# 迭代进化
for generation in range(num_generations):
# 计算适应度
fitness = np.apply_along_axis(lambda x: roc_auc_score(ref_seq, x), 1, population)
# 选择
parents = np.random.choice(population, size=(pop_size, 2), replace=True, p=fitness / np.sum(fitness))
# 交叉和变异
offspring = np.empty_like(population)
for i in range(pop_size):
parent1, parent2 = parents[i]
crossover_point = np.random.randint(0, seq_len)
offspring[i] = np.concatenate((parent1[:crossover_point], parent2[crossover_point:]))
offspring[i] = mutate(offspring[i], mutation_rate)
# 更新种群
population = offspring
# 返回最优解
return population[np.argmax(fitness)]
```
**参数说明:**
* `ref_seq`: 参考基因序列
* `reads`: 待检测的读序列列表
* `pop_size`: 种群规模
* `num_generations`: 迭代次数
* `mutation_rate`: 变异率
**代码逻辑分析:**
1. 初始化种群,每个个体代表一个可能的SNP
0
0





