金融风险管理中的GA算法:预测和减轻金融风险,守护财富安全
发布时间: 2024-07-03 23:16:01 阅读量: 95 订阅数: 27 

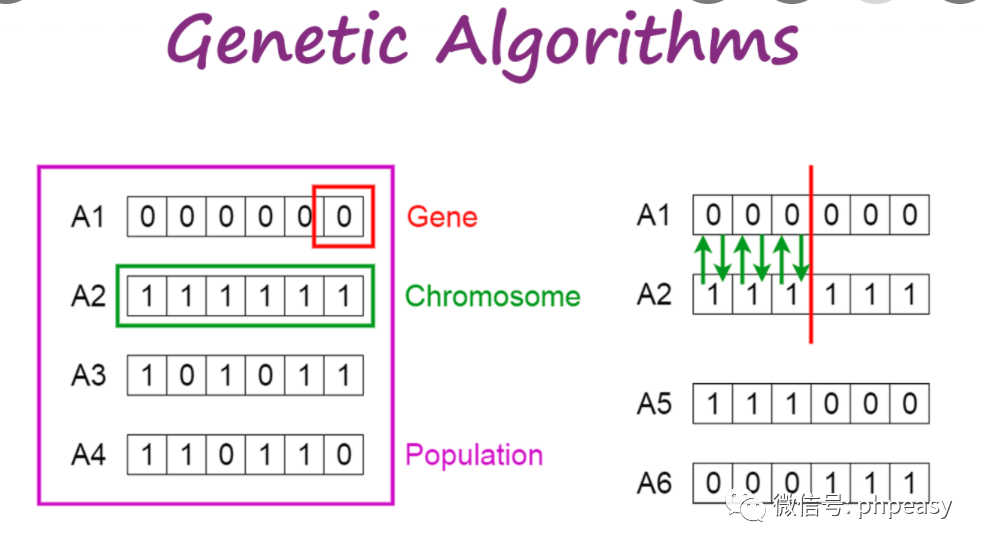
# 1. 金融风险管理概述
金融风险管理是金融行业中至关重要的一个领域,旨在识别、评估和管理金融机构面临的各种风险。这些风险可能包括市场风险、信用风险、操作风险和流动性风险等。金融风险管理的目标是最大程度地减少这些风险对机构财务状况和运营的负面影响。
金融风险管理涉及一系列技术和方法,包括量化分析、建模和优化。遗传算法(GA)是一种强大的优化算法,近年来在金融风险管理领域得到了广泛的应用。GA基于自然选择原理,通过迭代过程来搜索最优解,在解决复杂优化问题方面具有独特的优势。
# 2. 遗传算法(GA)在金融风险管理中的应用
遗传算法(GA)是一种受自然选择和遗传学启发的优化算法,它在金融风险管理中得到了广泛的应用。GA能够有效地解决金融风险管理中复杂的优化问题,并为风险预测和风险减轻提供有价值的见解。
### 2.1 GA的基本原理和优势
GA模拟了自然界中生物进化的过程。它从一个随机生成的种群开始,其中每个个体代表一个潜在的解决方案。个体通过选择、交叉和变异等算子进行进化,以产生更优的解。
GA具有以下优势:
- **全局搜索能力:**GA通过随机搜索和交叉算子探索整个解空间,从而提高找到全局最优解的可能性。
- **鲁棒性:**GA对噪声和局部最优解不敏感,因为它使用种群而不是单个解。
- **并行性:**GA可以并行执行,这使得它非常适合处理大规模优化问题。
### 2.2 GA在金融风险管理中的具体应用
GA在金融风险管理中得到了广泛的应用,包括:
#### 2.2.1 风险预测
GA可以用来预测金融风险,例如信用风险、市场风险和操作风险。GA模型通过学习历史数据中的模式和关系来构建预测模型。
**代码块:**
```python
import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.metrics import mean_squared_error
# 导入金融数据
data = pd.read_csv('financial_data.csv')
# 数据预处理
data = data.dropna()
data['Date'] = pd.to_datetime(data['Date'])
data = data.set_index('Date')
# 特征提取
features = ['Open', 'High', 'Low', 'Close', 'Volume']
# 划分训练集和测试集
X_train, X_test, y_train, y_test = train_test_split(data[features], data['Return'], test_size=0.2)
# 构建GA模型
model = GeneticAlgorithm(population_size=100, generations=100, crossover_rate=0.8, mutation_rate=0.2)
# 训练模型
model.fit(X_train, y_train)
# 评估模型
y_pred = model.predict(X_test)
mse = mean_squared_error(y_test, y_pred)
print('MSE:', mse)
```
**逻辑分析:**
这段代码演示了如何使用GA模型预测金融风险。它首先导入金融数据并进行预处理,然后提取相关特征。接下来,它将数据划分为训练集和测试集。GA模型被构建并训练,最后使用测试集评估模型的性能。
#### 2.2.2 风险减轻
GA还可以用于减轻金融风险,例如优化投资组合和制定风险管理策略。GA通过搜索满足特定约束条件的最优解来实现风险减轻。
**代码块:**
```python
import numpy as np
import pandas as pd
from cvxopt import matrix, solvers
# 导入资产收益率数据
returns = pd.read_csv('asset_returns.csv')
# 协方差矩阵
cov_matrix = np.cov(returns)
# 风险厌恶系数
risk_aversion = 0.5
# 构建GA模型
model = GeneticAlgorithm(population_size=100, generations=100, crossover_rate=0.8, mutation_rate=0.2)
```
0
0





