R语言生物信息学:数据包在基因组数据分析中的角色
发布时间: 2024-11-04 18:47:18 阅读量: 26 订阅数: 19 

# 1. R语言在生物信息学中的应用概述
生物信息学作为一门综合了生物学、计算机科学、数学和统计学的交叉学科,在过去几十年里已经取得巨大进步。R语言,作为一种开放源代码的统计编程语言,已经成为生物信息学数据分析中不可或缺的工具。它强大的数据处理能力、丰富的包资源,以及灵活的扩展性,使得它在处理生物信息学的海量数据和复杂问题时尤为突出。
## 1.1 R语言与生物信息学的结合
R语言的流行很大程度上归因于其在统计分析和图形表示方面的专长。生物信息学家经常需要执行大量的实验数据分析,这包括基因表达数据的处理、序列分析、群体遗传学研究等。R语言通过各种专门的数据包,如Bioconductor项目中的,提供了处理这些任务的工具。
## 1.2 生物信息学研究中的挑战与机遇
在生物信息学领域,数据的多样性和复杂性带来了挑战,同时也提供了巨大的机遇。R语言的应用可以帮助研究者解决这些挑战,例如通过高级统计方法识别基因之间的关联,或者利用机器学习技术预测蛋白质的功能。本章将概述R语言在生物信息学中的应用,并探讨如何有效地利用R语言解决生物学问题。
# 2. 生物信息学数据包的基础理论
在生物信息学领域,数据包作为R语言的重要组成部分,扮演着不可或缺的角色。从基因组数据的导入到复杂的系统生物学分析,数据包都为研究人员提供了强大而灵活的工具。本章将深入探讨生物信息学数据包的基础理论,包括数据类型概览、数据包的安装与管理,以及数据包在生物数据分析中的核心作用。
## 2.1 生物信息学数据类型概览
### 2.1.1 基因组数据的特点
基因组数据是生物信息学研究的基础,其特点是数据量巨大、数据类型多样以及数据结构复杂。典型的基因组数据包括基因测序数据、基因表达数据、表型数据和临床数据等。随着高通量测序技术的进步,基因组数据不仅在数量上呈指数级增长,在质量上也越来越精确。
基因组数据的主要特点可以概括为:
1. **高维性**:基因组数据通常包含成千上万个变量(例如基因表达水平),这些变量之间可能存在复杂的相互作用关系。
2. **异质性**:不同类型的数据(如DNA、RNA、蛋白质组数据)具有不同的性质和尺度。
3. **稀疏性**:在大规模基因组数据集中,大量的变量可能会显示为零或在统计上不显著,这意味着并非所有信息都是有用的。
4. **时间序列性**:基因表达和其他类型的生物数据往往随时间变化,这种动态性质是基因组数据分析的一个重要方面。
### 2.1.2 基于R的生物信息学数据结构
在R语言中,有多种数据结构可以用来处理生物信息学数据。数据框(DataFrame)、矩阵(Matrix)、列表(List)和环境(Environment)是R中最常用的数据结构,它们各有优势,并被广泛应用于不同类型的数据处理任务中。
数据框(DataFrame)是R中最常用的数据结构之一,特别适合处理表格形式的生物信息学数据。它是一种以行和列形式组织数据的结构,每一列可以是不同的数据类型,但每一列的长度必须相同。
```r
# 创建一个简单的数据框
data <- data.frame(
gene_id = c("gene1", "gene2", "gene3"),
expression_level = c(1.5, 2.8, 3.2),
p_value = c(0.01, 0.05, 0.001)
)
print(data)
```
在上面的例子中,`data.frame` 函数创建了一个包含三个基因的表达水平和P值的数据框。每一列可以存储不同的数据类型,例如表达水平是数值型,基因ID是字符型。
矩阵(Matrix)是另一种用于生物信息学的R数据结构,适合于进行数学运算。矩阵是二维的数据结构,其中所有的数据元素都必须是相同的类型。
列表(List)是R中最灵活的数据结构,可以包含不同类型的R对象(如向量、矩阵、函数等)。由于其灵活性,列表被广泛用于复杂数据结构的创建和处理。
## 2.2 数据包的安装与管理
### 2.2.1 CRAN和Bioconductor的介绍
CRAN(Comprehensive R Archive Network)是R语言的主要包仓库,提供了大量用于各种统计分析的R包。而Bioconductor是一个专注于生物信息学的R包项目,它提供了许多专门用于处理基因组数据的包。Bioconductor中的包通常与CRAN上提供的包在功能上有较大差异,强调了对生物数据的特殊处理需求。
### 2.2.2 数据包的搜索与安装方法
在R中安装数据包通常有几种方式。最简单的方法是从R控制台中直接安装:
```r
# 从CRAN安装数据包
install.packages("ggplot2")
# 从Bioconductor安装数据包
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("Biostrings")
```
在上面的代码中,`install.packages` 函数用于安装CRAN上的包,而`BiocManager::install` 用于安装Bioconductor中的包。
### 2.2.3 数据包的更新与依赖管理
随着R版本的更新,数据包可能需要相应的更新来兼容新版本的R。R提供了一种方便的方法来更新包:
```r
# 更新所有已安装的包
update.packages(ask = FALSE)
```
依赖管理是安装和更新包时需要考虑的重要因素。一些包可能会依赖于其他包。R语言的安装函数会自动尝试解决这些依赖关系,但在某些情况下,用户可能需要手动处理复杂的依赖问题。
## 2.3 数据包在生物数据分析中的核心作用
### 2.3.1 数据包对生物信息学的贡献
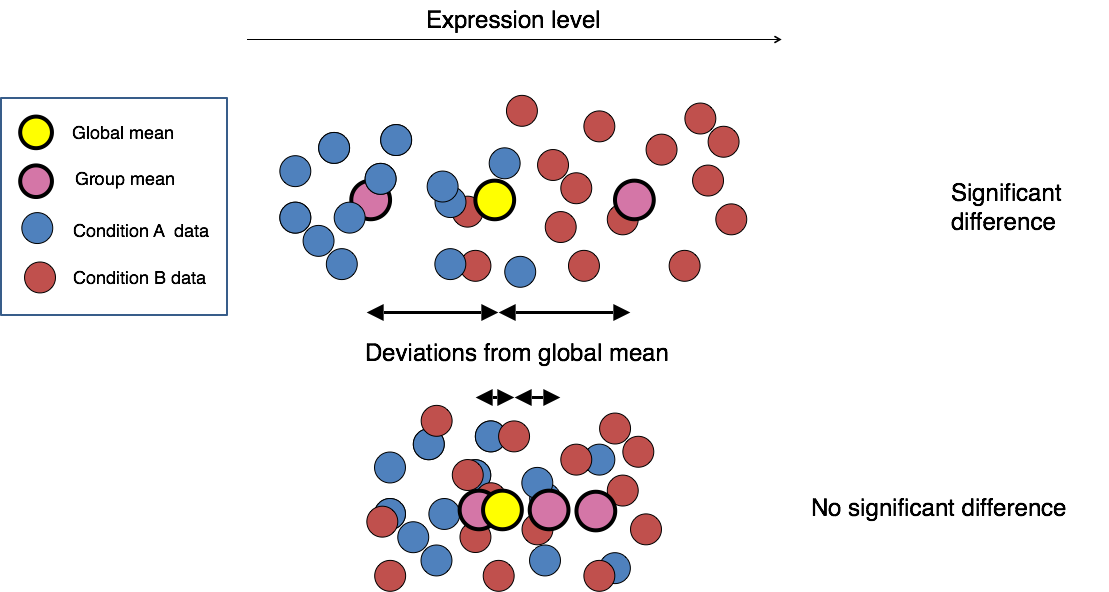
R语言的数据包为生物信息学研究者提供了大量工具,极大地推动了该领域的发展。这些包涵盖从基础统计分析到高级生物数据挖掘、可视化和报告生成的各个方面。例如,`Bioconductor`社区提供的`DESeq2`包,它是一种流行的用于差异基因表达分析的工具。另一个例子是`Gviz`包,它提供了强大的功能来可视化基因组数据。
### 2.3.2 案例研究:基因表达分析
基因表达分析是生物信息学研究中的一个典型应用。通过分析不同条件下的基因表达模式,研究人员可以发现潜在的疾病标记物、了解基因调控机制以及探索药物作用的新靶点。
例如,使用`limma`包进行基因表达分析通常包括以下步骤:
1. 导入基因表达数据矩阵。
2. 应用线性模型分析来识别差异表达的基因。
3. 对分析结果进行校验和可视化。
在实际操作中,这些步骤涉及到复杂的统计方法和生物信息学知识。数据包通过提供易于使用的函数和强大的统计模型,极大地简化了这一过程。
```r
# 基于limma包的简单差异表达分析示例
library(limma)
# 假设我们有表达矩阵 expr_matrix 和样本分组信息 group_info
design <- model.matrix(~ group_info)
fit <- lmFit(expr_matrix, design)
fit <- eBayes(fit)
# 提取差异表达基因
topTable(fit)
```
以上代码展示了如何使用`limma`包进行基本的差异表达分析。这段代码首先使用`model.matrix`创建一个设计矩阵,然后使用`lmFit`函数拟合线性模型,并通过`eBayes`函数进行贝叶斯校准。最后,`topTable`函数用于从拟合结果中提取差异表达的基因列表。
# 3. R语言数据包在基因组数据分析中的实践
## 3.1 基因组数据的导入与预处理
### 3.1.1 数据包在数据导入中的应用
在进行基因组数据分析前,首先需要导入各种类型的数据,如基因序列、表达数据集、变异信息等。R语言提供了丰富且强大的数据包,使得数据导入变得非常便捷和高效。例如,`readr`包可以快速读取和解析大型文本文件;`Biostrings`包则专门用于导入和处理序列数据。
使用`readr`包导入文本数据文件的示例代码如下:
```r
library(readr)
data <- read_csv("path/to/datafile.csv")
```
上述代码块中,`read_csv()`函数是用来导入逗号分隔值文件(CSV),并将其转换为R中的tibble数据框。tibble是一个更加现代化的数据框版本,它在显示和性能上有优势。在导入数据时,还可以通过参数对数据进行预处理,比如指定列的数据类型等。
接下来,使用`Biostrings`包导入序列数据的示例代码如下:
```r
```
0
0





