VectorNTI11:高效序列比对工具及使用步骤
"本教程详细介绍了如何使用DNA序列比对软件Vector NTI11进行多重序列比对。Vector NTI11提供了两种比对程序,AlignX适用于核酸和蛋白质序列比对,而AlignXBlocks则专门用于蛋白质序列比对。教程涵盖了从启动比对程序、加载序列到执行比对和查看比对结果的全过程。"
在生物信息学领域,Vector NTI11是一款强大的DNA序列分析工具,尤其在序列比对方面表现出色,被认为比DNAstar等其他软件更为易用。本教程主要针对的是第十章——多重序列比对功能的使用。
首先,启动序列比对有两种方式:通过程序集直接打开AlignX或从主程序中启动。用户可以在主程序界面有操作序列的情况下,选择AlignX-AlignSelected Molecules直接加载序列进行比对。
加载序列时,用户需要将序列文件添加至Vector NTI,这可以通过点击"Add Files"或从"Project"菜单中选择。在选择文件时,需要注意文件名不能过长,以免在比对过程中显示不全。一旦选择了文件并确认其类型(核酸或蛋白质),就可以导入程序。
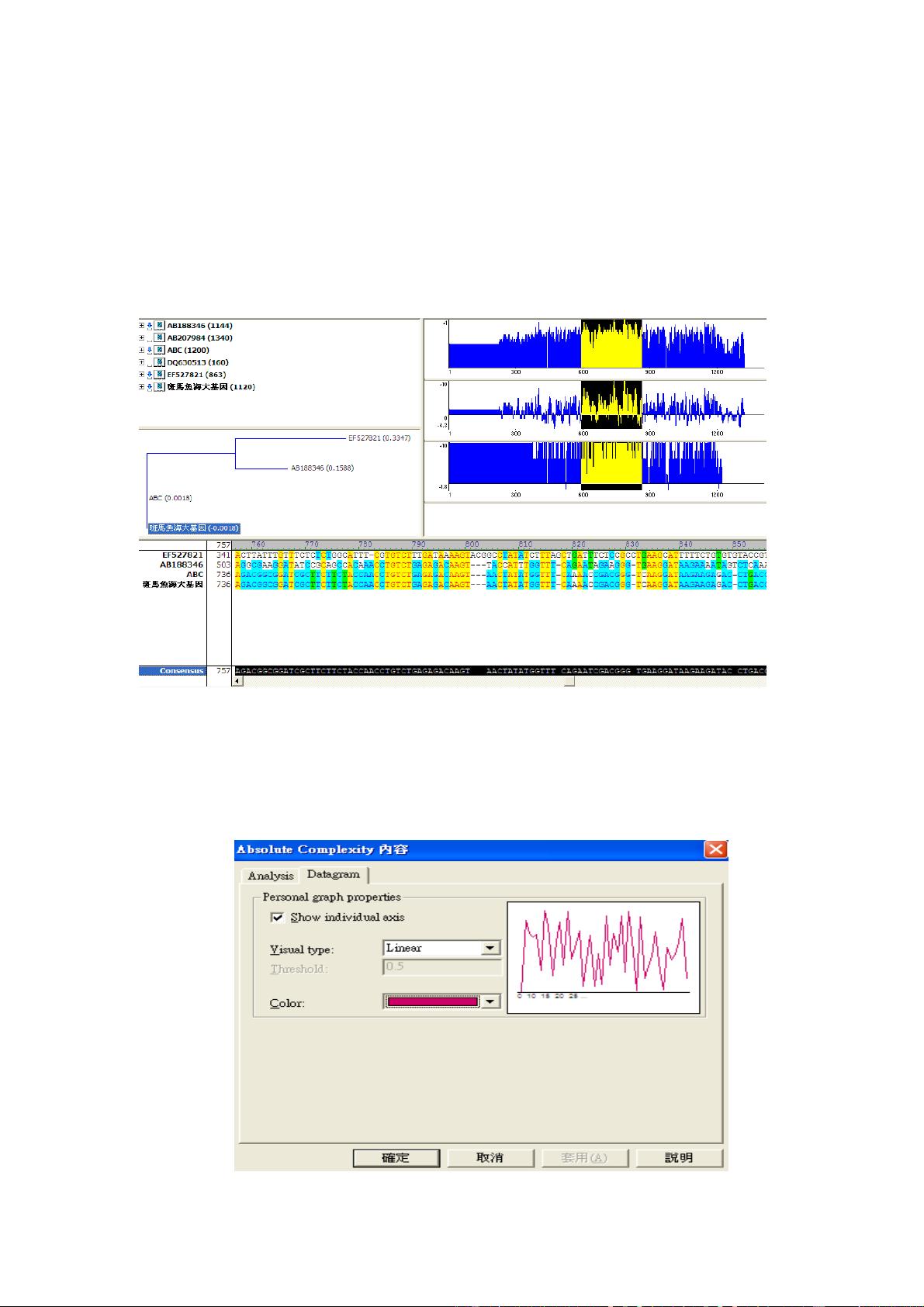
比对前,用户需要先选定要进行比对的序列,通过鼠标圈选。然后,只需点击"Align"按钮或从"Align"菜单选择"AlignSelectedSequence",比对过程就开始了。比对完成后,程序会展示比对结果,包括比对的图形表示和导引树(Guidetree),导引树可以帮助理解序列之间的进化关系。
在图10.10所示的比对结果显示界面,用户可以看到每个序列的排列以及它们之间的对应关系。图10.11中的比对相关结果提供了更深入的分析,包括可能的同源性区域和差异点。这些信息对于生物学家研究基因组、进化关系和功能注释等任务至关重要。
Vector NTI11的多重序列比对功能简化了生物信息学中的一项复杂任务,使其对初学者和专业人士都相对友好。通过本教程,用户能够掌握基本的序列比对操作,进一步利用这些工具进行科研工作。
点击了解资源详情
点击了解资源详情
点击了解资源详情
117 浏览量
点击了解资源详情
2689 浏览量
点击了解资源详情
点击了解资源详情
qq_17087985
- 粉丝: 0
最新资源
- 深入解析ASP.NET底层架构:Web请求的流转与处理
- UML中文版:Java程序员指南
- Jboss EJB3.0 实战教程:从入门到精通
- 提升IE技巧:智能ABC与加密文件实用操作
- Windows CE.NET入门教程:配置与调试
- C++编程提升技巧:专家Scott Meyers作品精华解读
- 林锐博士的《高质量C++/C编程指南》要点解析
- Eclipse实战指南:Java开发者入门宝典
- VxWorks文件压缩与硬盘加载优化
- JSP数据库开发全攻略:Oracle集成与实战指南
- JBuilder9中构建Struts应用实战教程
- VxWorks下BSD4.4规范网络程序设计详解
- Struts框架详解:构建高效Web应用
- Velocity模板引擎:Java中的强大工具
- 智能奥秘:无机生命体的创建与智能原理探索
- C++在嵌入式系统中的关键技术与应用深度探讨