【生物信息学中的应用】:支持向量机在基因表达数据分析中的威力!
发布时间: 2024-09-03 18:37:59 阅读量: 237 订阅数: 73 


数分1.11Tableau安装及使用教程
# 1. 生物信息学与基因表达数据概述
## 生物信息学简介
生物信息学是一个跨学科的领域,它结合了生物学、计算机科学、统计学、数学和工程学等多个学科的知识,用以处理和分析生物学数据。该领域的一个核心任务是解读和管理基因组、蛋白质组以及其他类型的生物分子数据。
## 基因表达数据的意义
基因表达数据是生物信息学中的关键数据类型之一,它反映了在不同条件下,基因表达其功能产物——通常是mRNA或蛋白质的水平。这些数据对于理解生命活动过程、疾病机理以及药物作用等方面具有重要意义。
## 数据分析工具的重要性
为了从这些复杂的数据中提取有用的信息,需要借助先进的数据分析工具和算法。支持向量机(SVM)作为一种强大的机器学习算法,在基因表达数据分析中扮演着重要角色。通过本章的介绍,我们将为后续章节的深入探讨打下基础。
# 2. 支持向量机理论基础
## 2.1 支持向量机的基本概念
### 2.1.1 统计学习与机器学习的关系
统计学习和机器学习是两个密切相关但又有所区别的概念。统计学习强调通过统计方法从数据中学习模式和结构,而机器学习则更多地强调在计算机程序中实现自动学习和泛化能力。支持向量机(SVM)作为一种分类器,其核心是寻找数据的最优分割边界,这背后的理论基础来源于统计学习领域,但其应用和实现更多体现在机器学习的范畴。
在构建SVM模型时,我们通常需要确定一个最优的分类边界,这个边界能够将不同类别的数据以最大间隔分开。这个间隔的计算基于统计学中的间隔最大化原理,而模型的训练和验证则涉及大量计算和优化过程,这正是机器学习的典型应用。
### 2.1.2 支持向量机的数学原理
支持向量机是基于统计学习理论中的结构风险最小化原则构建的。其核心思想是,为了最小化泛化误差,需要在经验风险和置信范围之间找到一个平衡点。SVM通过最大化两个类别数据的间隔来控制模型的复杂度,从而达到良好的推广能力。
数学上,SVM通过构建一个超平面来区分两类数据,超平面的方程形式为 `w*x + b = 0`,其中 `w` 是超平面的法向量,`b` 是偏置项。SVM的目标是最大化两个类别数据点到该超平面的最小距离,即最大化间隔。这可以通过解决一个凸优化问题来实现,该问题在满足数据点正确分类的前提下,最小化 `||w||^2`,也即最大化间隔。
```math
minimize ||w||^2 / 2
subject to y_i * (w*x_i + b) >= 1, i=1,...,N
```
其中 `y_i` 是第 `i` 个数据点的类别标签,`x_i` 是第 `i` 个数据点的特征向量,N 是数据点的数量。
## 2.2 支持向量机的分类原理
### 2.2.1 最大间隔分类器
SVM的分类原理可以概括为最大间隔分类器。在特征空间中,数据被表示为点,而SVM的目标是找到一个分割超平面,使得最近的数据点(支持向量)到该超平面的距离最大。在二维空间中,这个超平面就是一个直线,而在更高维的空间中,则是一个超平面。
要正确分类所有的数据点,SVM还需要引入松弛变量(slack variables)来处理那些无法完全线性分割的数据。松弛变量允许某些点违反间隔最大化条件,但要为这些违规点支付一定的成本,这通过引入惩罚项 `C * sum(ξ_i)` 到目标函数中来实现,其中 `ξ_i` 是松弛变量,`C` 是惩罚参数。
```math
minimize ||w||^2 / 2 + C * sum(ξ_i)
subject to y_i * (w*x_i + b) >= 1 - ξ_i, i=1,...,N
ξ_i >= 0, i=1,...,N
```
### 2.2.2 核技巧与非线性可分问题
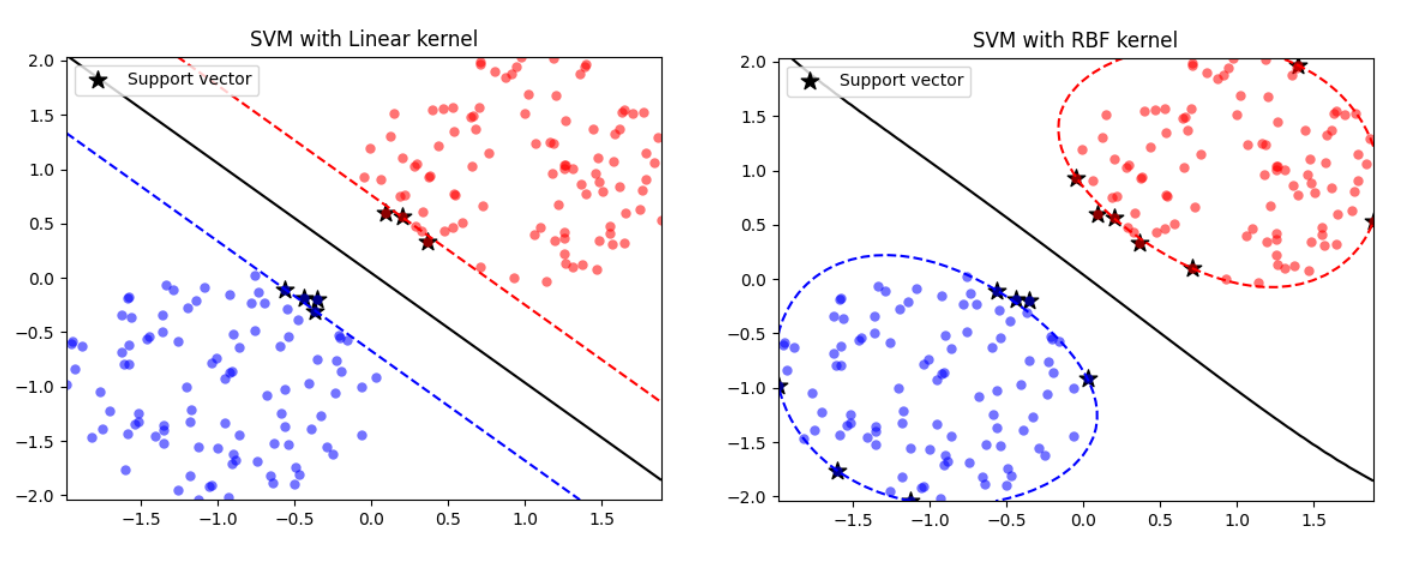
当数据是非线性可分时,即无法用一个线性超平面将两类数据完全分隔开,SVM使用核技巧来解决这一问题。核技巧的核心思想是通过映射将原始数据映射到一个更高维的空间,在这个新空间中数据可能变得线性可分。
核函数(kernel function)允许我们在不显式地计算高维特征空间的情况下,有效地计算高维空间中的点积。常见的核函数包括线性核、多项式核、径向基函数(RBF)核和sigmoid核。选择不同的核函数和相应的参数,可以构建出适用于不同数据集的SVM模型。
## 2.3 支持向量机的优化算法
### 2.3.1 序列最小优化(SMO)算法
序列最小优化算法是SVM训练中的一种有效且常用的方法。SMO算法的基本思想是,将大优化问题分解为一系列最小的优化问题。这些最小问题只需要考虑两个拉格朗日乘数(Lagrange multipliers),因此可以非常快速地解决。
SMO算法的步骤包括选择一对拉格朗日乘数进行优化,并且在优化过程中保持其他乘数不变。通过不断迭代选择和优化对,SMO逐步逼近全局最优解。其优势在于计算复杂度较低,尤其是在支持向量数量较多的情况下,可以显著减少训练时间。
### 2.3.2 软间隔与正则化参数选择
在实践中,很难找到一个超平面完美地将两类数据分开,而且过于严格的要求可能使模型过于复杂,容易过拟合。因此,SVM引入了软间隔的概念,允许一些数据点违反间隔最大化原则。
软间隔通过引入松弛变量来实现,而松弛变量的权重由正则化参数C控制。C的值越小,对间隔违规的容忍度越高,模型的泛化能力越好,但模型的风险也可能增加。选择合适的C值是一个重要的模型选择问题,通常通过交叉验证来确定最佳值。
```python
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV
param_grid = {'C': [0.1, 1, 10, 100]}
svc = SVC(kernel='rbf')
clf = GridSearchCV(svc, param_grid)
clf.fit(X_train, y_train)
best_params = clf.best_params_
print("Best parameters: ", best_params)
```
上述代码使用了Scikit-learn库中的SVC(支持向量分类器)和GridSearchCV(网格搜索)来通过交叉验证选择最佳的正则化参数C。
# 3. 基因表达数据分析实践
随着生物信息学的飞速发展,基因表达数据已成为研究者进行疾病机理研究、新药开发以及个性化医疗的重要资源。基因表达数据提供了细胞中基因活性的信息,是生物信息学分析中不可或缺的数据类型。在本章节中,我们将探索如何获取和预处理基因表达数据,并进一步介绍使用支持向量机(SVM)进行分类的实践操作,最后讨论如何进行有效的特征选择。
## 3.1 基因表达数据的获取与预处理
### 3.1.1 数据来源与格式
获取基因表达数据的第一步是确定可靠的数据来源。目前,公共数据库如Gene Expression Omnibus (GEO)、The Cancer Genome Atlas (TCGA)以及ArrayExpress等提供了大量经过同行评议的基因表达数据集。这些数据集通常以CEL、GCT或CSV等格式存储,包含了样本的原始读数、处理后的表达值以及其他注释信息。
### 3.1.2 数据清洗与标准化方法
数据预处理是分析之前必不可少的步骤,包括数据清洗和标准化。数据清洗主要是去除异常值、填补缺失值等。而标准化是将数据统一到相同的比例尺度上,常用的标准化方法包括Z-score标准化、log2变换等。标准化的目的是减少数据分散性,从而提升后续分析的效果。
```python
import numpy as np
from sklearn.preprocessing import StandardScaler
# 示例代码:数据标准化
data = np.array([[2, 4, 1], [5, 3, 2], [2, 2, 4]])
scaler = StandardScaler()
normalized_data = scaler.fit_transform(data)
```
以上代码使用了Python中的scikit-learn库来执行数据标准化。参数说明为`data`是原始数据集,`StandardScaler()`是scikit-learn中用于数据标准化的类,`fit_transform`方法则结合了拟合(计算均值和标准差)和转换(标准化数据)的过程。
## 3.2 使用支持向量机进行分类
### 3.2.1 构建分类器的步骤
构建SVM分类器通常包括以下步骤:选择合适的核函数、设置C参数(软间隔的正则化参数),然后训练模型。核函数用于处理非线性可分问题,常见的核函数包括线性核、多项式核、径向基函数(RBF)核等。选择合适的核函数
0
0





